
재조합효소-중합효소 증폭법(RPA) 및 중합효소 연쇄반응법(PCR)에서 생성된 특이 DNA 증폭산물 간의 비교
Abstract
RPA (Recombinase Polymerase Amplification) is a rapid, specific DNA-amplification method under isothermal condition. Recently, RPA was variously applied to detect specific pathogens using amplification of specific nucleotide sequence from specific pathogen. In this study, it was found that specific DNA amplification using RPA is significantly different than specific DNA amplification using PCR, during application of RPA to detect specific pathogens in honeybee. DNA-quantity of specific DNA amplification using RPA is more rapidly increased than DNA-quantity using PCR under same condition of time. However, it was estimated that quantity of specific nucleotide sequence among DNA amplification using RPA was very lower than quantity of specific nucleotide sequence using PCR. In specific amplification using RPA with target template, specific DNA and also significant quantity of unspecific DNAs were produced. In addition, it was also found that quantity of unspecific DNAs is increased in specific amplification using RPA without target template. For the RPA-application to detect specific pathogens, it should be overcome that unspecific DNAs are mass-produced by specific amplification using RPA.
Keywords:
RPA, PCR, DNA-amplification, Detection, Recombinase서 론
재조합효소-중합효소증폭법(Recombinase Polymerase Amplification; RPA)는 박테리오파지 T4 재조합효소를 이용하여 DNA 이중가닥의 해리를 일으키고 동시에 DNA 중합효소와 특정 primer들을 사용하여 특정DNA를 증폭해 내는 방법이다. 이는PCR의 경우와 같이 표적 기질(target template)과 한 쌍의 primer (oligonucleotide)를 사용하여 특정 염기서열의 DNA를 증폭시킬 수 있으나, PCR의 경우와 달리 일정온도(37°C-42°C)의 범위에서 등온조건으로 증폭반응을 일으킬 수 있는 장점이 있다(Piepenburg et al., 2006).
현재 RPA는 매우 빠르게 증폭산물을 만들 수 있도록 발전되었으며, 이는 등온조건의 장점과 함께 널리 인식되어 특이 유전자 증폭을 통한 특정 병원체의 검출법에 다양하게 RPA가 응용되게 하였다. 즉, 열대열원충인 Plasmodium falciparum을 대상으로 개발된 RPA검출법은 25분 내에 nested PCR과 동일한 검출 민감성을 보여주었으며(Kersting et al., 2014b), 그 외의 병원체 검출에서 20분 내 RPA를 통하여 101 분자의 DNA, 105개 분자의 mRNA 그리고 100fg의 낮은 genomic DNA에서 각 병원체 검출이 가능함을 보여주었다(Euler et al., 2012; Euler et al., 2013; Boyle et al., 2013).
그러나 꿀벌의 병원체들을 대상으로 RPA를 적용하여 새로운 검출법을 개발하고자하는 연구에서 RPA에 의한 증폭산물은 PCR의 증폭산물과는 크게 다른 특성을 가진다는 것이 인식되었으며, 특히 RPA에 의한 증폭산물들은 아가로오스젤 전기영동에 의한 분석에서 그리고 각 반응 중 형광강도의 분석에서 그리고 DNA-chip을 사용한 DNA:DNA 혼성화(hybridization) 분석에서 PCR에 의하여 생산된 같은 염기서열을 가진 DNA 증폭산물과 차이를 보인 바 있다(민 등, 2016; 임 등, 2016).
따라서 본 연구는 RPA 또는 PCR에 의해 생산된 증폭산물들의 각 특성을 보다 정밀하게 분석해 보고자 하였으며, 각 증폭의 정확성(accuracy)과 특이성(specificity)를 중점으로 양자를 정량적 비교를 하고자 하였다.
재료 및 방법
시료 수집과 DNA 확보
꿀벌의 시료는 2016년 경기대 양봉장으로부터 수집하였고 이를 -70°C에서 냉동보관하며 실험에 사용하였다. 꿀벌로부터 Genomic DNA는 Patho Genespin™ DNA/RNA Extraction kit (Intron, Korea)를 사용하여 순수분리하였고, 시료는 꿀벌 성체 또는 유충을 각기 2마리씩 별도로 사용하였다.
본 실험의 주형으로 사용된 재조합 DNA는 경기대 유전학 실험실에서 확보하였으며 해당 DNA는 꿀벌각 병원체에 특이적인 염기서열을 바탕으로 제작된 것이다. 각 재조합 DNA는 대장균으로부터 Intron사 DNA-spin™ Plasmid DNA purification kit를 사용하여 순수분리하였고, -20°C 보관하며 사용하였다(Table 1).
RPA (Recombinase Polymerase Amplification)
사용된 재조합 DNA는 pBX-SBV3와 pDrive-EFB이며, RPA와 PCR에서 각 2.45×108 copies, 2.10×108 copies를 기준으로 사용하였다. 각 병원체의 특이 primer는 주문 제작하였고, 최종 농도 0.48μM로 조성하여 사용하였다(Table 2; Bionics, Korea).
실시간 RPA 반응을 모니터링하기 위하여 최종 농도 1×SyBr green Ⅰ(Sigma-Aldrich)을 사용하였으며, DNA-chip에 적용하기 위한 RPA에는 각 병원체에 특이적인 개별 primer 외에 hybridization 표시자 primer(HC primer, Hybridization control)를 최종농도 0.01μM가 되도록 첨가하였다.
RPA는 TwistAmp Basic “Improved Formulation”(TwistDx, United Kingdom)을 사용하였으며 제작자의 지시에 따라 수행하였고, 이를 약술하면, 먼저 각 RPA당 Primer free rehydration buffer 29.5μl, 280mM magnesium acetate는 2.5μl를 준비하고 동결 건조 된 pellet에 magnesium acetate를 제외한 RPA조성 용액을 넣고 pellet을 완전히 녹인 후 0.2ml white tube로 옮기고 2.5μl 280mM magnesium acetate를 부가하여 RPA를 시작하였다. 최종 volume은 50μl이었다. RPA는 37°C 등온으로 90분간 수행하였고, 같은 목적의 DNA 증폭을 수행하는 PCR과 쉬운 비교를 위하여, ExicyclerTM Quantitative Thermal Block (Bioneer, Korea)을 사용하고, 30초를 1 cycle로 설정하여 RPA의 실시간 증폭 양상을 형광강도의 변화로 모니터링 하였다.
RPA 산물과 정량적 비교를 위한 PCR
90분의 RPA와 같은 조건으로 90분 30회전의PCR을 수행하기 위하여 주형은 pBX-SBV3를 primer 쌍은 SBV-DC-F1/R1를 사용하였으며 RPA와 같이 2.45×108 분자를 주형으로 사용하였고 각 primer의 최종 농도는 0.48μM이었다. 단 RPA의 경우 반응의 최종량은 50μl이었으나, PCR의 경우 20μl로 반응을 진행하였다. 차후 정량을 위한 시료는 RPA 및 PCR의 시작 후 반응 시간 10분, 20분, 40분, 90분에 각기 반응을 마치게 설정하여 각기 확보하였다. 각 반응 시간 10분, 20분, 40분, 90분은 PCR의 경우 변성, 혼성, 중합의 1회전수로 환산하면 각 1.8회전, 5.3회전, 12.4회전, 30회전에 해당한다.
RPA의 경우 2.5μl 280mM magnesium acetate를 첨가하는 시간을 시작 시각으로 하였으며, PCR의 경우 초기 변성 94°C를 시작으로 90분까지로 하였다. Real-Time PCR mix는 2× Greenstar master mix solution (Bioneer, Korea)를 사용하였고, PCR기기는 RPA와 같이 ExicyclerTM Quantitative Thermal Block (Bioneer, Korea)을 사용하였다. PCR 반응 조건은 초기 변성 94°C 5분 후, 변성 94°C, 중합 52°C, 신장 72°C의 1회전을 30회 반복하여 90분에 이르게 하였고 RPA는 37°C 등온으로 90분간 수행하였다.
RPA산물과 PCR산물의 분석을 위한 실시간 정량 PCR
먼저 각 주형 plasmid DNA를 2.45×108분자를 기준으로 1/10씩 연속 희석하여 정량 PCR의 주형으로 사용하였으며 각 주형 분자에 대한 CT (Threshold cycles)값을 구하여 정량의 기준으로 사용하였다.
정량 PCR에서 사용된 각 시료는 측정하고자 하는 RPA의 산물 및 PCR산물들을 각기 1:104, 1:105, 1:106로 희석한 것이었으며, 정량 기준으로 준비된 CT값과 희석비에 근거하여 초기 주형량, 즉 초기주형의 분자수를 계산하였다.
정량 PCR에서도 각 특이 primer쌍들은 각 최종농도 0.48μM로 하였으며, 이는 RPA의 경우와 일치시키기 위함이었다. 실시간 PCR mix는 2× Greenstar master mix solution (Bioneer, Korea)를 사용하였고 PCR기기는 RPA의 측정에서도 사용한 ExicyclerTM Quantitative Thermal Block (Bioneer, Korea)을 사용하였다. PCR 조건은 94°C에서 초기 변성 5분을 진행하였고, 94°C에서 변성 10초, 52°C에서 혼성 10초, 72°C에서 중합 10초을 30회 반복하였다.
RPA와 PCR 산물들의 정량은 모두 삼반복 이상으로 수행되었으며, 계산된 각 분자의 수들의 표준편차를 구하였다.
RPA 및 PCR산물에서 총 DNA양의 측정 및 특이도(Ratio of specificity)의 계산
각 RPA와 PCR에서 SBV 특이 primer 쌍인 SB-PCRF1/R1에 의한 SBV특이 증폭산물의 길이는 236bp이며, 이의 1.0×1010분자의 무게는 2.56ng으로 계산되었다.
각 RPA 및 PCR 용액은 페놀처리를 거쳐 단백질 등을 제거하였으며 약술하면, DNA수용액에 동량의 증류수로 포화시킨 페놀을 넣고, 1분간 강하게 흔든 후 13,000rpm으로 원심분리하여 상하층을 분리하였고, 상층의 DNA용액만을 회수하여 동량의 phenol/chloroform/isoamyl alcohol (25/24/1, v/v/v)을 넣고 1분간 강하게 흔든 후 13,000rpm으로 상하층을 분리하였다. 다시 상층의 DNA용액만을 회수하여 동량의 chloroform/Isoamyl alcohol (24/1, v/v)을 넣고 1분간 강하게 흔든 후 13,000rpm으로 상하층을 분리하고, 상층의DNA용액을 회수, 사용하였다.
Phenol처리한 DNA용액은 잔류량의 chloroform을 제거하기 위하여 에탄올 침전을 수행하였으며, 약술하면, DNA용액에 3M Na-acetate (pH 5.2)를 가하여 최종 농도가 0.3M이 되게 하고, 이의 2배 양의 0°C 100% ethanol을 넣어 혼합한 후, -70°C에 1시간 정치시키고, 15,000rpm, 4°C, 15분의 원심분리를 시켜DNA를 침전시켰다. 상등액을 제거하고, 침전된 DNA에 1ml, 0°C, 70% ethanol을 넣어 세척한 후, 다시 15,000rpm, 4°C, 15분의 원심분리로 DNA를 침전시켜 재차 회수하였다. 이 침전된 DNA를 적당량 TE-buffer에 녹여내고, MaestroNano® Spectrophotometer (MaestroGen, USA)를 사용하여 OD260의 광학측정으로 총 DNA 산물의 양을 측정하였다(50ng/μl, OD260=1.0 기준).
한편, 총 DNA산물 중 SBV 특이 증폭산물의 양을 측정하기 위하여 RPA 또는 PCR에 의한 총 DNA용액을 3단계 10배씩 희석하여 각 희석액 중 SBV 특이 증폭산물의 분자수를 측정하였으며, 이를 무게단위로 환산하였다. 각 RPA의 특이도(Ratio of specificity)는 100x SBV 특이 증폭산물의 양(ng)/ 총 DNA 산물의 양(ng)으로 계산하였으며, 편의상%단위를 사용하였다.
특이 PCR산물과 RPA산물의 DNA chip 적용
PCR산물은 20μl의 반응액 전부를, RPA 산물은 50μl에서 20μl만큼 0.2ml white tube에 분주하여 DNA-chip과 hybridization에 사용하였다. DNA 산물은 95°C에서 5분간 정치하여 DNA의 이중가닥을 변성시킨 후, 58°C에서 2시간 또는 12시간 꿀벌병원체 11종 검출용 DNA-chip(Wang et al., 2016)과 300rpm의 진동 하에서 hybridization을 진행하였다. 이 후의 수세 과정은 4×SSC (Saline sodium citrate) buffer, 0.2×SSC buffer, distilled water 순으로 300rpm의 진동 하에서 각 5분씩 진행하였고, DNA-chip 표면을 건조시키기 위하여, LightCycler® 480 Multi-well-plate 96 white (Roche, Korea)를 사용하여 3,500rpm, 2분 간 원심분리하였다. Hybridization과 수세에 사용된 진동 항온기는 Thermomixer comfort (Eppendorf, Germany)이었다.
Hybridization 결과의 평가는 K-SCAN-CAP™ (KMAC, Korea)을 사용하여 미세 영상을 촬영, 확보하였으며, 이 영상을 기반으로 각 spot의 SBR (Spot/Background ratio)값을 산출하고, 이를 비교·분석하였다.
결과 및 고찰
RPA 및 PCR 최종 산물의 정량
꿀벌 병원성 바이러스인 낭충봉아부패병 바이러스의 특이 염기서열을 탑재한 pBX-SBV3 1ng(2.45×108분자)을 주형으로 사용하고, 해당 특이 primer 쌍인 SB-PCR-F1/R1를 0.48μM사용한 RPA에서는 총 90분의 반응 후 1.69×1014분자(50μl)가 존재하는 것으로 측정되었다. 이는 μl당 3.38×1012분자로 계산되며, 초기주형량(2.45×108분자)과 비교하면, RPA에 의하여 6.90×105배 증폭된 것으로 계산된다(Fig. 1).
Quantitative analysis of SBV-specific RPA according to reaction time. Template used 2.45×108 copies of pBXSBV3 in RPA. Final primer concentration was each 0.48?M SB-PCR-F1 or SB-PCR-R1. Product of PRA was diluted serially with ddH2O to confirm quantitative analysis using qPCR. Specific amplified RPA product quantified 1.81× 1011, 7.07×1011, 1.64×1012, and 3.38×1012 molecules of specific DNAs/μl of each RPA (50μl) in 10 min, 20 min, 40 min and 90 min-long incubations, respectively.
한편 같은 주형과 primer 쌍을 사용한 90분(30회전) PCR의 경우, 1.20×1014분자(20μl)가 존재하는 것으로 측정되었다. 이는 μl당 6.00×1012분자로 계산되며, 초기주형량(2.45×108분자)과 비교하면, 30회전 PCR에 의하여 4.90×105배 증폭된 것으로 계산된다(Fig. 2).
Quantitative analysis of SBV-specific PCR according to reaction time (cycles). Template used 2.45×108 copies of pBX-SBV3 in Real-Time PCR. Final primer concentration was each 0.48μM in SB-PCR-F1 and SB-PCR-R1. Products of Real-Time PCR was diluted serially with ddH2O to confirm quantitative analysis using qPCR. Specific PCR products were calculated as 1.55×107 molecules (10 min; 1.8 cycles), 4.03×108 molecules (20 min; 5.3 cycles), 2.17×1010 molecules (40 min; 12.4 cycles) and 6.00×1012 molecules (90 min; 30 cycles) per μl of each PCR (20μl), respectively.
이 결과는 RPA에 의한 특이 DNA의 증폭효과가 약간 우세한 것으로 보일 수 있으나, RPA와 PCR의 반응액 중 단위 부피 당 DNA 증폭양은 후자가 2배 이상 우수한 것을 나타났으며, PCR의 경우 RPA와 비교를 위하여 30회전(90분)으로 제한한 것을 감안하여야 할 것이다.
반응 시간별 RPA 산물의 증폭특성
pBX-SBV3 1ng(2.45×108분자)을 주형으로 사용하고, 해당 특이 primer 쌍인 SB-PCR-F1/R1를 최종농도 0.48μM로 사용한 RPA는, 총 10분의 RPA에서 μl당 1.81×1011분자의 특이염기서열이 존재하는 것으로 측정되었으며, 이는 전체 RPA(50μl)에 9.05×1012분자가 존재하고, 이를 초기 주형량에 대비하면 3.69×104배 증폭된 것으로 계산되었다.
총 20분의 RPA에서 μl당 7.07×1011분자의 특이염기서열이 측정되며, 이는 전체 RPA(50μl)에 3.54×1013분자가 존재하고, 이를 초기 주형량에 대비하면 1.44×105배 증폭된 것으로 계산되었다. 총 40분의 RPA에서 μl당 1.64×1012분자의 특이염기서열이 측정되며, 이는 전체 RPA(50μl)에 8.20×1013분자가 존재하고, 이를 초기 주형량에 대비하면 3.35×105배 증폭된 것으로 계산되었다. 총 90분의 반응에서 μl당 3.38×1012분자의 특이염기서열이 측정되며, 이는 전체 RPA(50μl)에 1.69×1014분자가 존재하고, 이를 초기 주형량에 대비하면 6.90×105배 증폭된 것으로 계산되었다(Fig. 1).
이 RPA 증폭양상을 분석하기 위하여 간단히 구간별 분 당 증폭배수로 계산해 보면 초기 주형량에 대비한 초기 10분간 RPA에서는 3,690배/분으로 나타나 매우 급격한 증폭반응이 수행되었음을 볼 수 있었다. 그러나 10분 이후 20분까지의 분당 증폭배수는 0.39배로 급격한 감소가 나타났으며, 이런 급격한 증폭 배수의 감소는 20분 이후 40분까지 분당 증폭배수가 0.12배, 40분 이후 90분까지 분당 증폭배수는 0.04배로 계산됨에 따라 지속적으로 더욱 감소됨을 볼 수 있다. 이는 RPA 반응속도의 특성을 보여주는 것으로 초기 10분간 RPA의 대부분 증폭반응이 수행되고, 이후 급격한 감소 후 90분까지 지속적으로 계속 약화되는 것으로 해석된다.
반응 시간별 PCR 산물의 증폭특성
RPA의 경우와 같이 pBX-SBV3 1ng(2.45×108분자)을 주형으로 사용하고, 해당 특이 primer 쌍인 SBPCR-F1/R1를 0.48μM로 사용한 PCR에서 총 10분의 PCR(1.8회전)에서 μl당 1.55×107분자의 특이염기서열이 존재하는 것으로 측정되었으며, 이는 전체 PCR(20μl)에 3.10×108분자가 존재하고, 이를 초기 주형량에 대비하면 1.27배 증가된 것으로 계산되었다.
총 20분의 PCR(5.3회전)은 μl당 4.03×108분자의 특이염기서열이 있는 것으로 측정되며, 이는 전체 PCR(20μl)에 8.06×109분자가 존재하고, 이를 초기 주형량에 대비하면 32.9배 증폭된 것으로 계산되었다. 총 40분의 PCR(12.4회전)은 μl당 2.17×1010분자의 특이염기서열이 측정되며, 이는 전체 PCR(20μl)에 4.34×1011분자가 존재하고, 이를 초기 주형량에 대비하면 1,770배 증폭된 것으로 계산되었다. 총 90분의 PCR(30회전)은 μl당 6.00×1012분자의 특이염기서열의 존재가 측정되며, 이는 전체 PCR(20μl)에 1.20×1014분자가 존재하고, 이를 초기 주형량에 대비하면 4.90×105배 증폭된 것으로 계산되었다(Fig. 2).
이 PCR에 의한 특이 염기서열의 증폭양상을 간단히 구간별 분 당 증폭배수로 계산하여 나타내면 초기 주형량에 대비한 초기 10분간 PCR은 0.13배/분이었으나, 10분 이후 20분까지 분당 증폭배수 2.60배, 20분 이후 40분까지 분당 증폭배수 2.69배, 40분 이후 90분 까지 분당 증폭배수는 270.88배로 급격히 증가하는 것을 볼 수 있다. 이는 PCR에 의한 증폭 특성을 정확히 보여준 것으로, 반응시간보다 PCR 회전수의 증가에 의한 특이염기서열의 기하급수적 증폭을 보여준 것으로 해석된다.
반응 시간별 RPA에 의해 생산된 총 DNA 산물 중특이 증폭산물의 비
각 RPA는 pBX-SBV3 1ng(2.45×108분자)을 초기 주형으로 사용하였고, SBV 특이 primer 쌍인 SB-PCRF1/R1를 사용한 것이다. 따라서 SBV특이 증폭산물의 길이는 236bp이며, 이의 1.0×1010분자의 무게는 2.56ng으로 계산되었다.
각 RPA용액은 페놀처리를 거쳐 단백질 등을 제거하였으며, 에탄올 침전을 거쳐 비교적 순수한DNA만을 회수하고, OD260의 광학측정으로 총 DNA 산물의 양을 측정하였다(50ng/μl, OD260=1.0 기준). 한편 총 DNA산물 중 SBV 특이 증폭산물의 양을 측정하기 위하여, RPA에 의한 총 DNA용액을 3단계 10배씩 희석하여, 각 희석액 중 SBV 특이 증폭산물의 분자수를 측정하였으며, 이를 무게단위로 환산하였다. 각 RPA의 특이도(Ratio of specificity)는 100× SBV 특이 증폭 산물의 양/총 DNA 산물의 양으로 계산하였으며, 편의상%단위를 사용하였다.
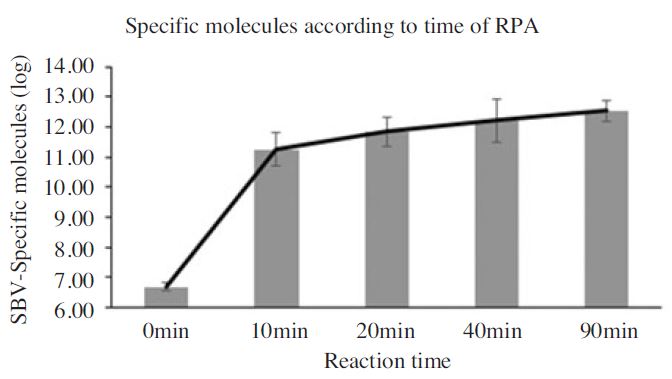
반응시간을 각기 10분, 20분, 40분, 90분으로 나누어 진행된 RPA들은 각 특이도가 4.437±0.345%, 16.232±6.426%, 34.545±5.505% 그리고 74.651±2.663%로 측정 후 계산되었다(Fig. 3; Table 3).
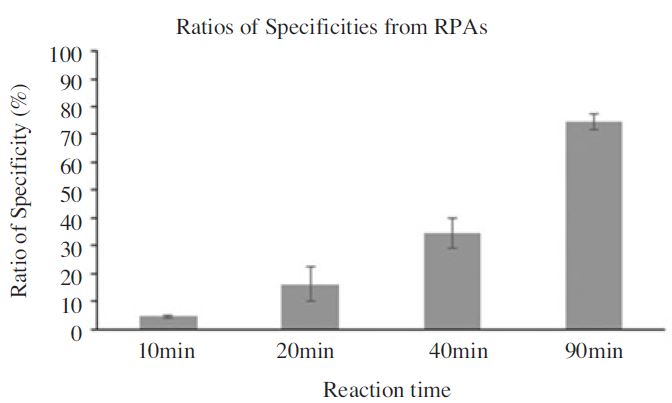
SBV-specific molecules among total amplified DNAs according to time of RPA. Using quantitative PCR, numbers of SBV-specific molecules were calculated. Initial template was used 2.45×108 molecules of pBX-SBV3 in each RPA. Total amplified DNAs of RPA were separately calculated based on Optical Density on 260 nm and size of SBVspecific amplicon. The ratio of specificity was calculated simply 100x specific molecules/total DNA molecules (even size) by OD260. The radios were 4.44%, 16.23%, 34.54%, 74.65% in 10 min, 20 min, 40 min and 90 min of RPAs, respectively.
각 RPA들의 증폭정도를 모니터링 한 형광곡선은 반응시작 10분에서도 상당한 형광값을 나타내어 신속히 증폭되는 것으로 예측하였으나(자료 미제시), 10분 반응한 RPA에서 특이 증폭산물은 총 DNA양 중 약 4.4% 수준이었으며, 이 특이도는 반응시간에 따라 점차 증가하여 최종 90분의 RPA에서 75%에 이르는 것으로 나타났다.
반응 시간별 PCR에 의해 생산된 총 DNA 산물 중특이 증폭산물의 비
각 PCR도 RPA의 경우 같이 pBX-SBV3 1ng(2.45×108분자)을 초기 주형으로 사용하였고, SBV 특이 primer인 SB-PCR-F1/R1를 사용한 것이다.
각 PCR용액도 페놀처리와 에탄올 침전을 거쳐 순수한 DNA만을 회수하였고, OD260의 광학측정으로 총 DNA 산물의 양을 측정하였다. 또한, 총 DNA산물 중 SBV 특이 증폭산물의 양을 3단계 희석 후, 각 희석액 중 SBV 특이 증폭산물의 분자수를 측정하였으며, 이를 무게단위로 환산하였다. 각 PCR의 특이도(Ratio of specificity)도 RPA의 특이도 계산과 같이, 100x SBV 특이 증폭산물의 양/ 총 DNA 산물의 양으로 계산하였다.
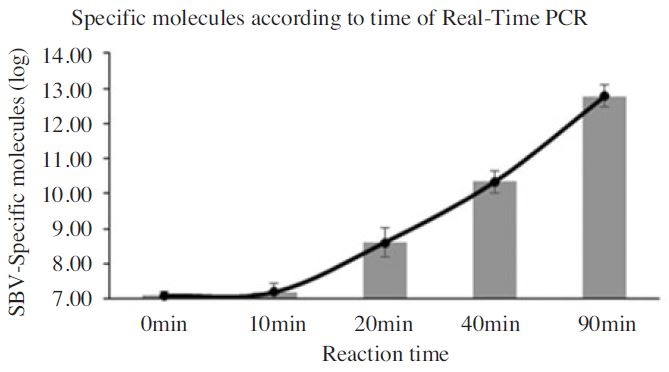
반응시간을 각기 10분, 20분, 40분, 90분으로 나누어 진행된 PCR들은, 각 특이도가 0.00±0.00%, 0.12±0.06%, 3.32±2.06% 그리고 1044.17±278.15%로 각기 계산되었다(Fig. 4; Table 4).
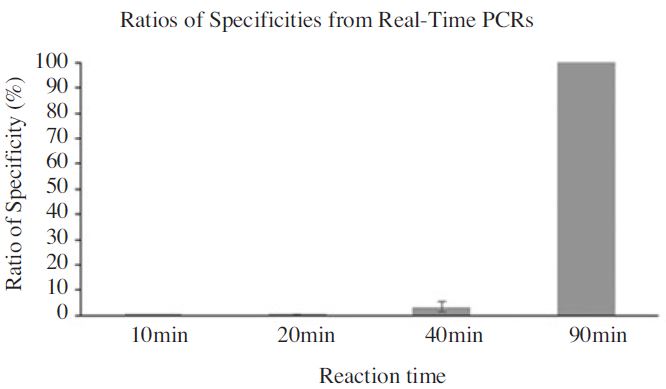
SBV-specific molecules among total amplified DNAs according to time of PCR. Using quantitative PCR, numbers of SBV-specific molecules were calculated. Initial template was used 2.45×108 molecules of pBX-SBV3 in each PCR. Total amplified DNAs of PCR were separately calculated based on Optical Density on 260 nm and size of SBVspecific amplicon. The ratio of specificity was calculated simply 100x specific molecules/total DNA molecules (even size) by OD260. The radios were 0.00%, 0.12%, 3.32%, 1,044.17% in 10 min, 20 min, 40 min and 90 min of PCRs, respectively.
각 PCR들의 증폭정도를 모니터링한 SyBr green의 형광곡선은 반응시작 40분(12.4회전)에서도 형광값의 증가를 관찰하지 못하였으며, 이후 신속히 형광값이 증가하여 증폭되는 것으로 예측할 수 있었다(자료 미제시).
PCR산물의 반응시간별(회전수 별) 특이도는 40분(12.4회전)까지 0.00-3.32%의 특이 증폭산물이 포함된 것으로 나타났으나, 90분(30회전)에서는 1044.17%의 특이적 산물을 포함하는 것으로 계산되였다. 여기서 1044.17%라는 특이도는 total DNA의 양과 236bp 특이 DNA의 분자수를 무게로 환산하는 과정에 기인하는 오차로 추측된다. 그러나 RPA산물과 PCR산물을 같은 방법으로 침전시키고 정량화하였기에, 상호 상대 비교는 어느 정도 가능할 것으로 사료되었다.
우선 RPA 90분에서 나타난 특이도는 본 실험에서 75%로 계산되었으며, PCR 90분(30회전)에서 나타난 특이도는 1,044%이었다. 이는 PCR의 특이도가 약 14배 높은 것을 나타내며, 역으로 PCR의 최종산물은 목적한 특이 증폭물의 비율이 높으며, RPA의 최종산물은 상대적으로 비특이 증폭물의 비율이 높은 것을 나타낸 것이라 할 것이다. 이런 추론은 해당 산물들의 Agarose Gel 전기영동에서 진한 단일밴드와 넓게 퍼진 밴드간의 차이로도 설명될 수 있을 듯 하였다(자료 미제시).
RPA와 PCR 증폭산물 간 차이에 따른 특이 증폭력의 차이
RPA에 의한 특이 염기서열의 증폭은 PCR의 그것에 비하여, 매우 짧은 시간에 급격하게 일어나며, 이이유 등으로 상대적으로 많은 비특이적 증폭산물이 생산되는 것으로 추론된다. RPA에서 주형으로 목적하는 재조합DNA로 사용하였을 경우, 생성되는 증폭산물들은 실시간 정량 PCR에서 충분한 특이 증폭산물이 포함되었음을 확인할 수 있었으나, 생성되는 RPA의 증폭산물을 형광으로 모니터링하였을 경우, 많은 비특이적 증폭산물로 인하여, 해당 template가 시료 중 존재하는지 또는 존재하지 않는지 분명히 판정하기에 어려움이 있었다.
이런 비특이적 증폭산물의 양산은 특정 병원체의 특정 염기서열을 증폭시킴으로써 특정 병원체의 존재를 확인하고자 하는 병원체 검색분야에 RPA를 응용하기 위하여 반드시 극복하여야 할 숙제이며, 판정방안의 개선 등 여러 방안이 강구될 수 있을 것이며, 이런 개선 요구는 여러 연구자에 의하여 제기된 바 있었다(민 등, 2016; Yang et al., 2015; Yehia et al., 2015).
사실 꿀벌의 병원균 Melissococcus plutonius의 특이 RPA 이후 DNA-chip을 이용한 DNA hybridization을 적용하였으며, 주형 DNA로는 M. plutonius의 genomic DNA를 사용하였고, M. plutonius의 특이 primer인 EFB-PCR-F1/R1를 각 최종농도 1.44μM로 사용하였다. 10ng, 1ng, 0.1ng 그리고 0ng (negative control)의 genomic DNA를 사용한 PCR 및RPA에서, 정량PCR들은 무난히 M. plutonius의 특이 증폭산물을 각 형광강도로써 정량적으로 보여줄 수 있었으나, RPA에서는 음성시료 포함, 모두 형광강도가 증가하는 것으로 나타나 판정을 보류하여야 하였다(1ng M. plutonius의 genomic DNA는 PCR에서 1.02×104분자로 정량됨; 자료 미제시).
이 RPA산물 및 PCR산물들은 바로 꿀벌 병원체 11종 검출용 DNA-chip(Wang et al., 2016)을 사용하여 DNA hybridization에 적용하였으며, PCR산물들은 58°C, 2시간의 혼성화 과정과 세척과정을 거쳐, 무난히 M. plutonius의 특이 증폭산물이 DNA-chip 중 M. plutonius의 특이 spot들에 혼성화되었음을 보여주었으나, RPA산물들은 제작사인 Twist DX (United Kingdom)의 조성물(특히 높은 염농도) 및 완충용액의 저해 때문에 DNA hybridization에 성립되지 아니하였다. 따라서 PCR 및 RPA산물들에 대한 회수와 hybridization 완충용액으로 교체가 진행되었으며, PCR산물의 경우, 58°C, 2시간의 혼성화 과정과 세척과정을 거쳐, 양성/음성 모두 인정되는 결과를 보였으나, RPA산물의 경우, 52℃, 12시간의 혼성화 과정과 세척과정을 거쳐서 정확한 양성/음성 판정을 할 수 있었다(자료 미제시).
혼성화에 정규 2시간이 아닌 12시간의 시간이 요구되는 것은 많은 비특이적 증폭산물과 특이적 증폭산물의 혼성화 경쟁 때문으로 추측되며, 온도변화에 의하여 primer들이 주형 DNA에 annealing되는 PCR의 상황과 달리, RPA는 recombinase에 의하여 annealing이 주도되기에, genomic DNA를 주형으로 annealing에서는 보다 불리하지 않는가 추측된다.
이 추측은 M. plutonius 가 발병된 감염시료에서 genomic DNAs(꿀벌 및 M. plutonius)를 추출하고, 그중 74ng을 주형으로 사용한 RPA 및 PCR의 결과에서 보다 분명하게 나타났다. PCR에서는 74ng의 genomic DNAs 주형 중에 12.48분자의 M. plutonius 초기 주형이 존재함을 정량적으로 보여주었으며, DNA-chip상의 M. plutonius의 특이 spot들에 선별적으로 hybridization됨을 보여주었으나, RPA의 산물들은 M. plutonius의 특이 산물이 증폭되었음을 형광강도 측정 및 DNAchip상의 hybridization에서도 확인될 수 없었다(자료 미제시).
등온의 조건에서 빠르게 특정 DNA를 증폭시킬 수 있는 방법은 여러 의미에서 매우 매력적인 기술일 것이다. RPA는 이런 조건을 충족시키는 매우 우수한 실험법 중의 하나이나, 본 연구에서 지적하는 몇 가지 단점을 보완하여야 할 것이며, 이러한 보완책으로 Lateral flow assay (LFA) 또는 DNA hybridization이 응용된 RPA 검출법들이 보고되었으나(Kersting et al., 2014a; Kersting et al., 2014b; Santiago-Felipe et al., 2014), 보다 우수한 새로운 기술이 고안되어 RPA가 특정 병원체의 분자적 검출법으로 널리 적용될 것으로 기대한다.
적 요
재조합효소-중합효소증폭법(Recombinase Polymerase Amplification; RPA)은 등온에서 특이 DNA를 빠르게 증폭시킬 수 있는 방법이다. 근래 RPA는, 특이 병원체가 보유한 특이 염기서열의 증폭을 통하여 특이 병원체들을 검출하는데 다양하게 응용되고 있다. 본 연구는 꿀벌 병원체에 대한 검색방법으로 RPA법을 적용하는 과정에서 RPA에 의한 특이 유전자의 증폭은 중합효소연쇄반응(PCR)에 의한 특이 유전자의 증폭과 유의미하게 다르다는 것을 발견하였다. RPA에 의한 특이 유전자의 증폭물의 양은 같은 시간 조건하의 PCR에 의한 특이 유전자 증폭산물의 양에 비하여 빠르게 증가되나, 증폭산물 중 중 목적한 특이 염기서열의 양은 RPA에 의한 것이 PCR에 의한 것에 비하여 크게 낮은 것으로 측정되었다. 목표 기질이 존재한 RPA의 특이 유전자 증폭에서, 특이DNA의 증폭과 함께 상당한 양의 비특이DNA들이 만들어 지며, 더욱이 목표 기질이 존재하지 않는 RPA의 특이 유전자 증폭에서도, 비특이 DNA양은 증가되는 것이 발견되었다. 특정 병원체의 검출을 위한 RPA의 응용을 위하여 RPA에 의한 특이 증폭에서 비특이적 DNA가 다량 생성되는 점은 마땅히 극복되어야 할 것이다.
Acknowledgments
본 연구는 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 수출전략기술개발사업(115067-02), 첨단생산기술개발사업(115058-02, 115102-03), 농생명산업기술개발사업(312027-03)과 2017학년도 경기대학교 대학원 연구원장학생 장학금 지원에 의하여 수행되었습니다.
인 용 문 헌
- 민상현, 왕지희, 임수진, 이칠우, 윤병수, (2016), 꿀벌 병원성 바이러스의 정량적 검출을 위한 정량 실시간 재조합 효소-중합효소 증폭법의 개발, Journal of Apiculture, 31(1), p41-50.
- 임수진, Luong, G. T. H., 민상현, 왕지희, 윤병수, (2016), 역전사 실시간 Recombinase Polymerase Amplification (RT/RT RPA)에 의한 꿀벌 Black Queen Cell Virus의 신속 검출, Journal of Apiculture, 31(1), p41-50.
-
Boyle, D. S., D. A. Lehman, L. Lillis, D. Peterson, M. Singhal, N. Armes, M. Parker, O. Piepenburg, and J. Overbaugh, (2013), Rapid Detection of HIV-1 Proviral DNA for Early Infant Diagnosis Using Recombinase Polymerase Amplification, mBio, 4(2), pe00135-13.
[https://doi.org/10.1128/mbio.00135-13]

-
Euler, M., Y. Wang, P. Otto, H. Tomaso, R. Escudero, P. Anda, F. T. Hufert, and M. Weidmann, (2012), Recombinase Polymerase Amplification Assay for Rapid Detection of Francisella tularensis, Journal of Clinical Microbiology, 50(7), p2234-2238.
[https://doi.org/10.1128/jcm.06504-11]

-
Euler, M., Y. Wang, D. Heidenreich, P. Patel, O. Strohmeier, S. Hakenberg, M. Niedrig, F. T. Hufert, and M. Weidmann, (2013), Development of a Panel of Recombinase Polymerase Amplification Assays for Detection of Biothreat Agents, Journal of Clinical Microbiology, 51(4), p1110-1117.
[https://doi.org/10.1128/jcm.02704-12]

- Ha, J. S., H. M. Lee, D. S. Kim, Y. K. Lim, and B. S. Yoon, (2005), A PCR Detection Method of Melissococcus pluton for Rapid Identification of European Foulbrood (Korean), Journal of Apiculture, 20(1), p9-18.
- Kersting, S., V. Rausch, F. F. Bier, M. von Nickisch-Rosenegk, (2014a), Multiplex isothermal solid-phase recombinase polymerase amplification for the specific and fast DNAbased detection of three bacterial pathogens, Microchim Acta, 181, p1715-1723.
- Kersting, S., V. Rausch, F. F. Bier, M. von Nickisch-Rosenegk, (2014b), Rapid detection of Plasmodium falciparum with isothermal recombinase polymerase amplification and lateral flow analysis, Malaria Journal, 13, p99.
- Nguyen, K. C. T., M.-S. Yoo, I.-W. Kim, M.-H. Kang, S.-H. Han, and B.-S. Yoon, (2008), Development of PCR Detection Method for Sacbrood Virus in Honeybee (Apis mellifera L.), Korean J. Apiculture, 23(3), p177-184.
-
Piepenburg, O., C. H. Williams, D. L. Stemple, and N. A. Armes, (2006), DNA Detection Using Recombination Proteins, PLoS Biol, 4(7), pe204.
[https://doi.org/10.1371/journal.pbio.0040204]

-
Santiago-Felipe, S., L. A. Tortajada-Genaro, S. Morais, R. Puchades, and Á. Maquieira, (2014), One-pot isothermal DNA amplification - Hybridisation and detectionby a disc-based method, Sensors and Actuators B, 204, p273-281.
[https://doi.org/10.1016/j.snb.2014.07.073]

- Wang, J. H., D. B. Lee, S. J. Ku, M. C. Peak, S. H. Min, S. J. Lim, C. W. Lee, and B. S. Yoon, (2016), Development of a Detection Method against 11 Major Pathogens of Honey Bee using Amplification of Multiplex PCR and Specific DNA-chip (Korean), Journal of Apiculture, 31(2), p133-146.
-
Yang, Y, X. Qin, G. Wang, Y. Zhang, Y. Shan, and Z. Zhang, (2015), Development of a fluorescent probe-based recombinase polymerase amplification assay for rapid detection of Orf virus, Virology Journal, 12, p206.
[https://doi.org/10.1186/s12985-015-0440-z]

-
Yehia, N., A.-S. Arafa, A. A. El Wahed, A. A. El-Sanousi, M. Weidmann, and M. A. Shalaby, (2015), Development of reverse transcription recombinase polymerase amplification assay for avian influenza H5N1 HA gene detection, Journal of Virological Methods, 223, p45-49.
[https://doi.org/10.1016/j.jviromet.2015.07.011]

