
한국형 낭충봉아부패병 바이러스(kSBV)의 정의를 위한 새로운 SBV 유전자형의 제안
Abstract
korean Sacbrood Virus (kSBV) was firstly found since 2008 in Korea, and it might be a main reason why 99% of populations belonged to Apis cerana in Korea were disappeared now. In this study, full length, reported DNA sequences of 32 Sacbrood Viruses (SBVs) were analysed based on alignments using nucleotides and/or deduced amino acid sequeces. In this analysis, variable deletions were found that are located around 2,100 bases in each CDS of SBVs. The genotyping depended on these deletions might be related with infection-patterns by these pathogens. In addition, it is not escaped our notice that the genotyping we have proposed immediately suggests a possible origin of kSBV for the quarantine and protection against further invasion.
Keywords:
Genotype, Deletion region, Polyprotein, Sacbrood virus, kSBV서 론
한국형 낭충봉아부패병 바이러스(korean Sacbrood Virus; kSBV)는 2008년 국내에서 처음 발견된, 낭충봉아부패병 바이러스(Sacbrood Virus; SBV)에 속하는 특정한 변형 바이러스이다(김 등, 2008). 이들은 국내 침입 이후 특히 국내 토종벌(Apis cerana)에 집중적으로 막심한 피해를 주어 왔으며, 상대적으로 국내 서양종 꿀벌(Apis mellifera)에는 큰 피해를 주지 않아, 강한 숙주 특이성을 보였다. 국내 토봉의 총 봉군수는 2009년 38만군이상으로 집계되었으나, 2016년 조사에서는 1만 봉군을 약간 넘는 것으로 나타나, kSBV 발생 이래, 국내의 99% 이상 토종벌이 사라졌음을 보여주고 있다(2015년 통계연보, 연도별 토종벌 사육군수 2016년 한봉협회 회원 토종벌 봉군수 조사).
국내 토종벌과는 달리, 국내 서양종 꿀벌은 kSBV에 대하여 저항성이 있는 것으로 파악되고 있으나, 서양종 꿀벌에서도 kSBV 존재가 특이 유전자의 증폭으로 발견되는 사례가 늘어가고 있다. 이 사실은 kSBV에 의한 국내 서양종 꿀벌의 감염 및 전파가 우려되는 바이나, 아직 kSBV 감염에 의한 개체내, 봉군내 질병의 증폭현상은 보고된 바가 없었다.
SBV의 종내 분류는 지금까지 서양종 꿀벌에서 검출되면 발견 지역에 관계없이 SBV라 통칭하여 왔으나, 동양종 꿀벌(Apis cerana)에서 검출되는 SBV는, 발견 국가에 따라 뚜렷한 감염성과 염기서열의 차이를 보이기 때문에, 검출지역에 따라 구분되어 왔으며(이 등, 2012), 중국에서 처음 보고된 것은 chinese Sacbrood Virus (cSBV)로(Ma et al., 2011) 국내에서 발견된 SBV는 korean Sacbrood Virus (kSBV)로 구분하여 왔다(Choi et al., 2010; Han et al., 2011).
SBV는 분류학적으로 Iflavirus에 속하며, 단일 양성가닥 RNA (positive single strain RNA)를 유전체로 가진 바이러스로, 타 Retrovirus와는 달리 DNA-stage가 없는 것으로 알려졌다(Ghosh et al., 1999; Mayo, 2002). SBV의 전체 염기 서열은 꿀벌 바이러스 중 처음으로 밝혀졌으며, 총 8,832 bases의 길이에, 2,858 아미노산 잔기를 포함하는 polyprotein을 암호화하고 있고, 이의 유전자는 179 염기에서 8,752 염기까지 이어져 있었다(Ghosh et al., 1999). 또한, SBV의 유전체는 5’-방향에는 주로 구조적 유전자가, 3’-방향에는 주로 비구조적 유전자가 위치하고 있다(Chen et al., 2006).
본 연구는 NCBI에 전체 염기서열이 보고된 32종의 Sacbrood virus 염기서열 정보를 분석대상으로 하였으며, 감염대상과 감염성에서 차이를 보이는 SBV들의 특성을 유전체 차원에서 그 차이점을 찾아보고자 하였다. 이 과정에서 SBV의 세분을 위한 genotyping을 시도하였으며, 약간의 유의미한 결과를 얻었기에 이에 보고하고자 한다.
재료 및 방법
본 연구는 NCBI (National Center for Biotechnological Information)의 GenBank에서“Sacbrood virus complete”로 검색하여 nucleotide sequence와 amino acid sequence를 수집하였고, NCBI의 GenBank Accession Number와 등록일 그리고 SBV를 분리해 낸 꿀벌의 종과 국가로 나누고, Sacbrood virus의 complete genome sequence를 통해 SBV를보다미세하게분류하고자하였다(Table 1).
먼저, 각각의 유전자 정보를 통합 비교하기 위하여 각 polyprotein의 CDS (coding sequence) 부분의 nucleotide sequence를 본 염기서열 정보와 별도로 정렬하였으며, 이 CDS들의 자료를 alignment 분석하였다(Clustal X Program, version 2.1: http://www.clustal.org/clustal2/#Download). Alignment 과정에서 발생한 염기서열의 틈새(sequence gap)들은 해당 amino acid sequence를 참고하여 조정하였으며, 전체적으로 가장 높은 아미노산 서열 상동성 및 염기서열 상동성이 될 수 있도록 조정하였다.
결과 및 고찰
SBV 유전체의 polyprotein CDS 중 특정부위의 결손
NCBI에 보고된 32종 Sacbrood virus complete genome의 염기서열 중 polyprotein의 CDS를 통합적으로 비교하였다. 32종의 염기서열은 그 CDS의 크기가 각기 8,583 bases(2,861 아미노산), 8,580 bases(2,860 아미노산), 8,577 bases(2,859 아미노산), 8,550 bases(2,850 아미노산), 8,544 bases(2,848 아미노산), 8,529 bases(2,843 아미노산)의 6가지로 정리되었으며, 이는 polyprotein의 CDS 중 특정부분에서 염기서열의 결손(deletion)이 여러 형식으로 발생되었고, 이에 따른 아미노산 서열의 결손이 각기 나타난 것으로 판단되었다.
한편, 이 등(2013)은 SBV의 CDS 중 이런 결손의 발생을 지적하였으며, 이를 기반으로 Genotyping을 시도하였으나, 2013년 당시 보고된 SBV의 염기서열 자료는 단 15개 이었으며, 또한 염기서열 정보에만 의존한 불충분한 분석으로 결손의 정확한 위치를 보여주지 못하였고, 따라서 제안한 genotype들은 현재에 와서 각 SBV의 특성들을 구분하기에는 다소 미흡한 면이 있었다.
본 연구에서는 아미노산 서열을 기반으로, 각 해당 염기서열을 재조정하고, 이들 염기서열을 비교함으로써 4가지 결손의 합리적 위치를 추론할 수 있었다.
32 SBV polyprotein들의 각 아미노산 서열에 따른 정렬
32종 SBV의 complete genome의 염기서열 중 polyprotein의 CDS에서 추론된 각 아미노산 서열들은 통합적으로 결손을 최소화하는 방식으로 분석하였다. 각 아미노산 서열들은 최소의 결손에서 비교적 쉽게 grouping 되었으며, 각 아미노산 서열들이 최대의 상동성을 갖도록 재조정되었고, 최종적으로 결손이 큰 순서부터 정렬하였다(Fig. 1).
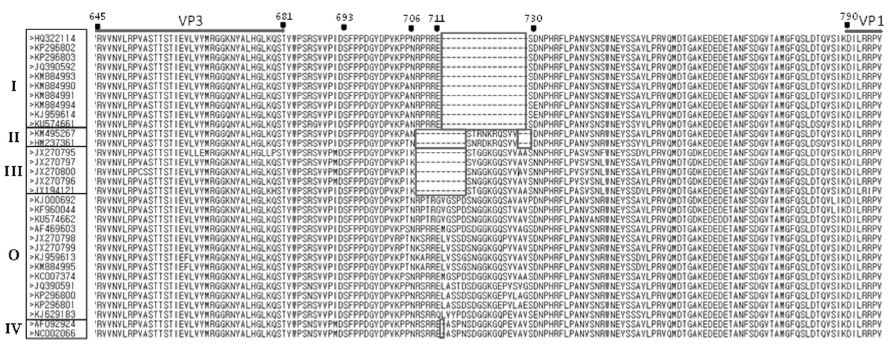
Alignment among deletion region of amino acid sequences in polyprotein of SBV. The deletion region (red boxes) showed multiple alignment of the amino acid sequences from SBV complete genome. Type I has 17 amino acids long deletion which located 712 to 728 amino acid from start of polyprotein. Type II has 13 amino acids long deletion which located 707 to 716, and 727 to 729 amino acid. Type III has 10 amino acids long deletion which located 707 to 716 amino acid. Type IV has only 1 amino acid deletion which located 712 amino acid residue. Type O is polyprotein sequences without deletion between structural protein, VP3 (to 681) and VP1 (from 790) in SBV.
결과는 아미노산 서열 712부터 728 아미노산 잔기까지 총 17개의 아미노산 잔기가 결손된 SBV들이 큰 group으로 나타났으며, 이어 707부터 716 아미노산 잔기까지, 그리고 727부터 729 아미노산 잔기까지 두 개의 작은 결손으로 총 13개 아미노산 잔기가 결손된 SBV들, 그리고 707부터 716 아미노산 잔기까지 총 10개 아미노산 잔기가 결손된 SBV들이 있었으며, 같은 위치에 712 아미노산 잔기, 단 하나가 결손된 것도 발견할 수 있었다. 한편, 다수의 SBV들에서 결손이 발견된 아미노산 서열 707부터 729까지의 부위에서, 결손없이, 상호 상동성이 높은 아미노산 서열이 존재하는 것도 다수 발견되었으며, 그 출처가 중국, 인도, 베트남, 한국, 호주로 다양하였고, 또한 숙주도 서양종 꿀벌(Apis mellifera), 동양종 꿀벌(Apis cerana) 모두에서 발견되었기에, 이들을 O형(원형, Origin형, Zero형)으로 판단하게 되었다(Fig. 1).
32 SBV polyprotein의 CDS 염기서열에 따른 정렬과 Genotyping
32종 SBV의 complete genome의 염기서열 중 polyprotein의 CDS 염기서열은 정렬된 아미노산 서열에 따라 상동성 정렬로 재조정하였으며, 염기서열 상에서 결손의 정확한 위치를 추론하였다.
아미노산 서열에서 확인된 결손(Fig. 1)은 32종 SBV의 CDS 염기서열들에서 결손위치가 2,119 염기에서 2,187 염기의 범위에 있음이 확인되었고, 각기 2,100bp 부근에서 51염기, 39염기, 30염기, 3염기의 결손이 있는 것으로 grouping되었다. 이들은 결손의 크기에 따라 정렬하였으며, 각 결손의 이름을 결손 시작 위치와 크기에 따라, 2134D51, 2119D39, 2119D30, 21340D3으로 명명하였으며, 원형으로 판단되는 SBV들은 O 형 또는 2100D0로 명명하였다(Fig. 2).

Alignment among deletion region of nucleotide sequences from CDS of polyprotein in SBV. (A) The deletion region among SBV complete genome showed on multiple alignment of the nucleotide sequences based on amino acid sequences from polyprotein in SBVs. (B) Alignment of identical amino acid sequences deduced from nucleotide sequences including deletion regions. 2134D51 indicated 51 bp-long deletion between 2,134 bp and 2,184 bp from coding sequence of polyprotein, 2119D39 indicated 39 bp-long deletion between 2,119-2,148 and 2,179-2,187 bp. 2119D30 indicated 30 bp-long deletion between 2,119-2,148. 2134D3 is only 3 bp-long deletion 2,134-2,136. 2100D0 has no deletion in this area.
정리하면, O형(Zero type)은 그 CDS의 크기가 8,580 염기, 2,860 아미노산 잔기이며, 타 SBV에서 나타난 결손 부위인 2,119 염기에서 2,187 염기사이에 결손이 없는 genotype을 말하며, 상기 결손 부위 이외의 위치에 3개 염기가 가감된 것들도 발견되어 총 CDS 염기의 수가 8,583염기, 8,577염기의 것도 포함하였다(2100D0).
I형(I type)은 가장 큰 51염기의 결손이 나타난 형으로, 총 CDS 크기는 8,529 염기이며(O type과 비교하면 2,134부터 2,184 염기까지 결손), 총 2,843 아미노산 잔기를 보유하고, 그 결손 위치는 아미노산 서열에서 712부터 728까지 총 17개의 아미노산 잔기가 결손된 것이다(2134D51형).
II형(II type)은 39염기의 결손이 있는 SBV로, 총 CDS 크기가 8,544 염기이며, O type에 비교하면 2,119부터 2,148까지 30염기가, 그리고 2,179부터 2,187까지 9개가 결손되어 있어, 총 2개의 결손으로 총 39염기가 결손된 것이다. 총 2,848 아미노산 잔기를 보유하며, 이는 아미노산 서열에서 707부터 716까지 그리고 727부터 729까지 총 13개 아미노산 잔기의 결손을 보이고 있다. 별도로 언급하면 이 II형은 상기의 결손부위 외의 위치에 별도로 3개 염기가 결손되어 있는 것으로 밝혀졌다(2119D39형).
III형(III type)은 30염기의 결손이 있는 SBV로, 총 CDS 크기가 8,550 염기이며, O type에 비교하여 2,119부터 2,148까지 30염기가 결손된 것이며, 이 결손은 II형의 2개 결손 중 하나와 동일한 위치이다. 총 2,850 아미노산 잔기를 보유하며, 이는 아미노산 서열에서 707부터 716까지 총 10개 아미노산 잔기의 결손을 보이고 있다(2119D30형).
IV형(IV type)은 단지 3염기가 2,134-2,136위치에서 결손된 SBV로, 총 CDS 크기가 8,577 염기이다. 2,859의 총 아미노산 잔기를 보유하며, 이는 아미노산 서열에서 712 아미노산 잔기 하나만 결손을 보이고 있다(2119D3형).
본 연구에서 발견된 결실(deletion)은 O형과 I, II, III, IV 형으로, 유전자형을 나눌 수 있는 근거가 되었으며, 이 결실은 CDS 염기서열 2100 부위에서 발견되었기에, 2100결실(2100 deletion)로 명명하였다.
이 2100결실에서 O형(원형)은 2100D0으로 표현할 수 있으며, 각 유전자형은 2134D51(I형), 2119D39(II형), 2119D30(III형), 2119D3(IV형)으로, 각기 결실 시작 위치와 그 크기를 나타낼 수 있도록 하였다.
각 유전자 형에 따른 SBV들의 특성들
종래 한국형 낭충봉아부패병 바이러스(korean Sacbrood Virus; kSBV)라 알려졌던 염기서열들은 모두 4종이며((HQ322114, JQ390592, KP296802, KP296803), 이들은 모두, 본 연구에서 제안하는 I형인 2134D51형에 속하였다. 이들은 2011년부터 2016년까지 NCBI에 보고된 것으로, 3종은 국내 토종 꿀벌에서 분리된 kSBV이나, JQ390592만은 서양종 꿀벌에서 분리된 kSBV임이 주목된다(Fig. 3).
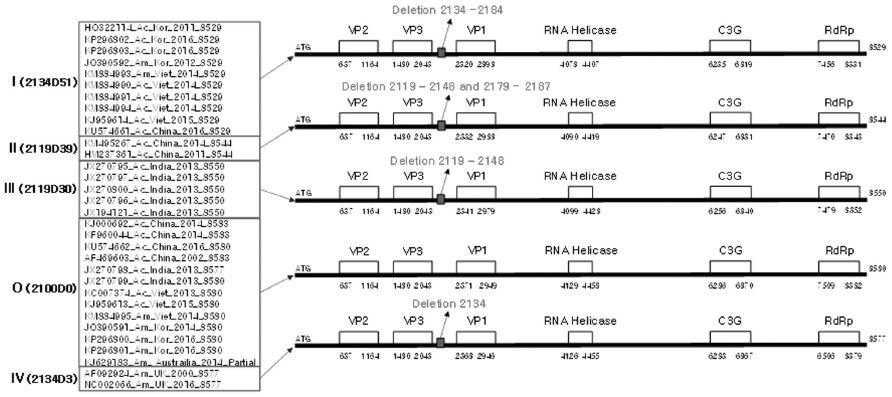
Deleted positions on the nucleotide sequences from CDS in polyprotein region of SBV. Each SBV full-length sequence is indicated GenBank accession number, host species of SBV (Am, Apis mellifera Ac, Apis cerana), name of nation, submitted year to GenBank, size of coding sequences in nucleotides. The positions of each genes(VP2, VP3, VP1, RNA helicase, C3G peptidase and RNA dependent RNA polymerase) in SBV were indicated. The deletion region is located between VP3 and VP1.
kSBV가 속한 2134D51형(I형)에는, 베트남에서 2014-2015년 보고된 5개의 염기서열이 있으며, 이들중 4개 염기서열은 동양종 꿀벌에서 분리된 SBV를 보고한 것이며, KM884993만은 서양종 꿀벌에서 분리한 SBV의 염기서열을 보고한 것이다. 한편, 2016년 보고된 중국산 SBV의 염기서열(KU574661)은, 국내 및 베트남에서 분리된 kSBV(본 연구의 I형 또는 2134D51형)와 같은 유전자형이며, 이는 중국산 SBV이기는 하나, 유전자형이 2134D51형(I형)에 속하기에, 종래 통칭하던 cSBV가 아니며, kSBV로 구분되어야 할 것이다. 정리하자면, 유전자형 2134D51형(I형)은 한국, 베트남, 중국에서 숙주의 구분 없이(주로 동양종 꿀벌에서 분리되었으나, 서양종 꿀벌에서도 분리됨) 분리된 SBV들이며, 최초 보고지인 국내의 연고를 살려 kSBV형이라 지칭하는 것이 합리적이라 판단된다.
2119D39(II형)은 모두 중국에서 보고된 2개의 염기서열(2011, 2014년)이며, 이들은 모두 동양종 꿀벌에서 분리된 SBV를 보고한 것이다. 이는 지역적으로 중국에서 처음 보고된 유전자형이기에, cSBV형으로 지칭되는 것이 혼란을 줄일 수 있을 것으로 판단된다.
2119D30(III형)은 모두 인도에서 보고된 5개의 염기서열(2014년)이며, 이들 역시 모두 동양종 꿀벌에서 분리된 SBV를 보고한 것이다. 이는 지역적으로 인도에서 처음 보고된 유전자형이기에, iSBV형으로 지칭되는 것이 합리적일 것이다.
2134D3(IV형)은 영국에서 보고된 2개의 염기서열이며(2000, 2016년), 모두 서양종 꿀벌에서 분리된 SBV들이었다. 이들은 아래 기술된 O형(2100D0형)의 또 다른 변이형으로 추측하며, 이들에 대한 자료가 조금 더 축적되어야 이 변이의 의미를 해석할 수 있을 것으로 기대한다.
한편, O형(2100D0형)에는 13개의 염기서열들이 포함되었으며, 4개의 중국산, 3개의 한국산, 3개의 베트남산, 2개의 인도산, 1개의 호주산 SBV 염기서열이 속하였다. 서양종 꿀벌에서 분리된 SBV는 한국산 모두와 호주산, 그리고 3개 중 하나의 베트남산 SBV 염기서열이었으며, 중국산 및 인도산 모두, 그리고 3개 중 2개의 베트남산 SBV 염기서열들은 동양종 꿀벌에서 분리한 SBV들이었다.
가장 다양한 지역에서 분리된 이유로 이 SBV의 O형(2100D0형)은 서양종 꿀벌에서 처음 분리하였던 낭충봉아부패병 바이러스(SBV)의 원형에 가장 가까운 것으로 추론된다. 이들을 서양형 낭충봉아부패병 바이러스(western SBV; wSBV)라 호칭하면, 이 wSBV는 영국, 호주, 한국, 베트남, 중국, 인도에 모두 실존하는 것이 확인되는 것이며, 2017년 현재 서양종 꿀벌과 동양종 꿀벌에서 모두 발견되는 상황으로 파악된다. 따라서, O형(2100D0형)에 속하는 모든 SBV는 발견지역과 관계없이 모두 wSBV라 통칭되어야 할 것이며, 이는 유전자형의 검사를 통하여 확인할 수 있을 것이다.
상기의 결과들을 근거로, 본 연구에서 제안하는 유전자형들을 통칭되던 cSBV, kSBV 등과 다음과 같이 재정리하여 연결한다면 SBV변이형들의 명칭에 대한 혼동을 최소화할 수 있을 것이라 생각한다.
1. 즉, SBV의 원형은 본 연구의 O형(2100D0형)이며, 이는 최초 발견지역이 영국임을 감안하고, 이미 전 세계의 다양한 지역에서 발견되기에 wSBV(western SBV)라 통칭하고,
2. 본 연구의 I형(2134D51형)은 현재 한국, 베트남, 중국에서 발견되었으나, 최초 발견지역이 국내임을 감안하여 kSBV(korean SBV)라 통칭하며,
3. 본 연구의 II형(2119D39형)은 현재 중국에서만 발견되었고, 최초 발견지역도 중국이기에 cSBV(chinese SBV) 라 통칭하고,
4. 본 연구의 III형(2119D30형)은 현재 인도에서만 발견되었기에, 최초 발견지역인 인도를 사용하여 iSBV(indian SBV)라 통칭하며,
5. 본 연구의 IV형(2134D3형)은 현재 영국에서만 보고된 변이형이기에, 우선 eSBV(england SBV)라 통칭하는 것이다.
2134D51형(I형)에 한국산, 베트남산, 중국산이 혼재하는 이유
베트남 유래 vSBV가 국내의 kSBV와 같은 유전자형임을 보여준 것은 이 유전자형의 기원에 대하여 많은 숙고를 하게 한다. 본 연구에 포함된 32종의 SBV 염기서열 분석에서 베트남산 SBV는 모두 8종이 포함되었고, 그 중 5종은 kSBV와 같은 유전자형인 2134D51형(I형)에 속하나, 나머지 3종은 O형(2100D0형 wSBV)에 속하는 것으로 나타났다. 또한 한국산 SBV도 7종 중 4종이 2134D51형(I 형 kSBV)로, 3종이 O형(2100D0형 wSBV)에 속하는 것으로 나타났기에, 양국의 SBV는 본 연구에서 제안하는 통칭 wSBV와 kSBV가 모두 존재하는 것으로 파악되었다. 또한 양국 모두에서 서양종 꿀벌에는 주로 wSBV가, 동양종 꿀벌에는 주로 kSBV가 발견되는 경향을 보이고 있다.
또한, 국내와 베트남 모두 통칭 kSBV(2134D51형; I형)가 서양종 꿀벌과 동양종 꿀벌에서 각기 분리되었다는 사실은 kSBV가 종간 장벽을 넘어 동양종 꿀벌에서 서양종 꿀벌로 그 숙주범위를 넓히고 있는 과정이 아닌가 우려되고 있으며, 현재 kSBV는 동양종 꿀벌에서 서양종 꿀벌로 적응 숙주의 범위를 넓히는 변이가 진행되고 있는지 면밀히 연구되어, 이런 위험에 대비하여야 할 것이다.
한편, 중국의 동양종 꿀벌에서도 통칭 kSBV(2134D51형, I형)가 발견된 것은 유의미한 결과이며, 과연 2134D51형(I형, 통칭 kSBV)이 어느 국가에서 기원되었는지는 보다 많은 연구가 진행되어야 보다 분명해 질 것으로 판단된다.
현재의 자료로 판단하자면, 한국, 베트남, 중국 중 한 곳에서 kSBV (2134D51형, I형)가 변이-발생하였으며, 이 변이형이 나머지 2개 국가로 전파되어 각기 발견된 것으로 추론할 수 있을 것이다(Fig. 3).
SBV 각 유전자 부위 중 2100결실의 위치
SBV의 polyprotein은 기본형(본 연구의 O형)에서 2,860 아미노산 잔기를 포함하고 있으며, 이는 각 특정 단백질로 절단되어, 효소(비구조 단백질) 또는 구조 단백질로 기능을 하여 SBV를 재구성하는 것으로 알려져 있다. Polyprotein의 CDS상에서 5’쪽은 구조 유전자 부위(VP2, VP3, VP1)가 있으며, 이어 비구조단백질 유전자 부위(RNA helicase, C3G peptidase, RNA dependent RNA polymerase)가 3’방향에 위치하고 있다(Chen et al., 2006).
본 연구에서 발견된 SBV의 2100결실은 구조단백질의 유전자 VP3와 VP1 사이에 존재하며, polyprotein 상에서 이 부위는 단백질로 번역되어 존재할 것이나, 각특정단백질(예 VP3, VP1)로 분절화 될 때, 이결실 부위는 VP1단백질에도, VP3단백질에도 포함되지 아니하여, 재구성된 SBV에서 구조 단백질로 발견되지 않는다. 따라서 이 부위는 SBV의 생존과는 관련이 적은 유전자 부위로 파악되며, 이 이유로 가장 많은 변이가 SBV의 생존과 무관하게 발생된 것이 아닌가 생각된다(Fig. 1; Fig. 3).
2009년 이전에 국내에 존재하였던 SBV(통칭 wSBV)는 국내의 서양종 꿀벌 및 동양종 꿀벌 모두에게 큰 피해는 입히지 않은 것으로 파악되고 있었으나, 2009년 이후 kSBV(2134D51형 또는 I형)가 유독 국내 토봉에 극심한 피해를 입힌 사실은 양자의 유전자 차이에서 기인되었을 것이라 추론된다. O형과 I형, 양자의 유전자 차이는 본 연구에서 제안한 2100 결실에 가장 큰 차이가 있으며, 각 구조단백질들과 비구조단백질들에서는 두드러진 차이를 현재까지 발견할 수 없었다. kSBV가 국내 토봉에게 보여준 강한 감염력과 살상력의 근거는 본 연구에서 제안하는 2100 결실 부위를 포함한 SBV의 여러 특정유전자를 중심으로 보다 심층적 분석과 연구가 필요하다 할 것이다.
결론적으로 본 연구는 전세계적으로 보고된 SBV들의 염기서열 분석을 통하여 구조유전자 VP3과 VP1 사이에 존재하는 2100 결실(deletion)을 발견하였으며, 이를 기반으로 SBV들을 genotyping하였고, 제안하는 유전자형에 속하는 각 SBV들의 주요 정보를 정리하였다. 제안된 SBV의 유전자형은 SBV의 전파차단과 극복을 위한 심층적 연구에 유용히 사용되기를 기대한다(Table 2).
적 요
한국형 낭충봉아부패병 바이러스는 2009년 국내에 발생하여, 현재 국내 토봉의 99% 이상을 소멸시킨 주요 원인으로 추정되고 있다. 본 연구는 보고된 낭충봉아부패병 바이러스(SBV) 32종의 전체 염기서열을 비교 분석하여, 2,100 염기 부근에 특징적인 결실들(deletions)을 발견하였으며, 이에 기반으로 SBV의 유전자형을 제안하게 되었다. 제안된 유전자형에 의한 분류에서, 각 SBV의 지역성 및 범지역성, 감염특성 등에 유관함을 볼 수 있었다. 아울러, 제안된 이 유전자형은, 검역과 이후의 침임에 대한 방호를 위하여, kSBV의 기원도 바로 암시하고 있음을 우리는 잘 인식하고 있다.
Acknowledgments
본 연구는 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 수출전략기술개발사업(115067-02), 첨단생산기술개발사업(115058-02, 115102-03), 농생명산업기술개발사업(312027-03)과 2017학년도 경기대학교 대학원 연구원장학생 장학금 지원에 의하여 수행되었습니다.
References
- 김혜경, 최용수, 이명렬, 이만영, 이광길, 안난희, (2008), 국내 꿀벌에서의 Sacbrood Virus(SBV) 진단, Korean J. Apiculture, 23(2), p103-109.
- 이보람, Nguyen Van Phu, 유미선, 노지나, 윤병수, (2012), Sacbrood Virus와 korean Sacbrood Virus의 구별 검출을 위한 PCR법 개발, 한국양봉학회지, 27(2), p129-135.
- 이중구, 백준수, 윤병수, (2013), Polyprotein에 기반한 Sacbrood Virus(SBV)와 한국산 SBV의 분류, Journal of Apiculture, 28(5), p373-378.
-
Chen, Y., Evans, J., Feldlaufer, M., (2006), Horizontal and vertical transmission of viruses in the honey bee, Apis mellifera, J. Invertebr. Pathol, 92(3), p152-159.
[https://doi.org/10.1016/j.jip.2006.03.010]

- Choi, Y. S., M. Y. Lee, I. P. Hong, N. S. Kim, H. K. Kim, K. G. Lee, and M. L. Lee, (2010), Occurrence of Sacbrood Virus in Korean Apiaries from Apis cerana (Hymenoptera: Apidae), J. Apiculture, 25(3), p187-191.
-
Ghosh, R. C., Ball, B. V., Willcocks, M. M., Carter, M. J., (1999), The nucleotide sequence of sacbrood virus of the honey bee: an insect picorna-like virus, J. Gen. Virol, 80(Pt. 6), p1541-1549.
[https://doi.org/10.1099/0022-1317-80-6-1541]

- Han, S.H., Y.S. Choi, and M.L. Lee, (2011), Development of Highly Specific Quantative Real-Time PCR Method for the Detection of Sacbrood Virus in Korean Honeybees, Apis cerana, J. Apiculture, 26(3), p233-240.
- Ma, M., C. Ma, M. Li, S. Wang, S. Yang, and S. Wang, (2011), 377 Polyprotein에 기반한 Sacbrood Virus(SBV)와 한국산 SBV의 분류 Loop-mediated isothermal amplification for rapid detection of Chinese sacbrood virus, J. Virol. Methods, 176, p115-119.
- Mayo, M.A., (2002), Virus taxonomy - Houston 2002, Arch. Virol, 147, p1071-1076.5.
