
뒤영벌 병원체 11종에 대한 실시간 중합효소 연쇄반응 검출법 개발
Abstract
The multiple real-time PCRs against pathogens of Bombus species including DWV, IAPV, KBV, SBV, BQCV, kSBV, SBPV and Paenibacillus larvae, Mellisococcus plutonius, Lysinibacillusfusiformis, and Klebsiella oxytoca have been developed. One extracted nucleic acid from Beesample could be applied to 11 different PCRs in same time and condition. Specific PCR-products were amplified qualitative and quantitative manner inner 20 minutes successfully, when each 1000 molecules of pathogen-specific target DNA is existed as template, respectively. The multiple PCR detection that we propose would be expected to apply to quarantine test for international exchange of Bombus species.
Keywords:
Bombus, PCR detection, Quarantine test, Pollinator서 론
화분매개곤충의 이용은 시설채소 농법의 발달에 큰 영향을 주었으며, 꿀벌(Apis mellifera) 그리고 서양뒤영벌(Bombus terrestris)이 주요 화분매개 곤충으로 사용되고 있다. 뒤영벌에 대한 실용화 연구는 유럽을 비롯하여 미국, 캐나다에서 이미 활발하게 이루어져 상품화 되었으며, 일본, 중국, 대만 그리고 한국 등은 화분매개용으로 뒤영벌을 수입하여 사용하고 있다(Ruijter et al., 1997; Dafni et al., 1998; Masahiro et al., 2000; Velthuis and Van Doorn, 2006). 국내의 경우, 시설재배 면적이 증가하면서 1994년에 처음 2,300봉군의 서양뒤영벌(Bombus terrestris)이 수입되기 시작하였으며, 2004년에는 약 30,000봉군 이상이 수입된 바 있었으나(윤 등, 2008), 이후 뒤영벌의 국내 생산은 크게 증가하여, 2010년 이래 국내 뒤영벌 수요를 넘어서면서 새로이 뒤영벌 수출의 길을 모색하고 있다.
Dino 등(2015)은 A. mellifera와 Bombus species에서 각각 Deformed wing virus (DWV), Black queen cell virus(BQCV), Acute bee paralysis virus (ABPV), Slow bee paralysis virus (SBPV)들을 정량적으로 검출한 바 있었으며, 특히 A. mellifera에는 DWV의 발병률이 높은 것을, Bombus species 중 일부에서 SBPV의 발병률이 높음을 보여 주었다. 한편, 국내의 연구로 최 등(2009)은 꿀벌의 바이러스인 DWV가 서양뒤영벌과 호박벌에도 존재함을 처음으로 확인하였고, 이어서 서양뒤영벌에서 7종의 꿀벌바이러스, 즉 DWV, BQCV, Israeli acute paralysis virus (IAPV), Cloudy wing virus (CWV), Chronic bee paralysis virus (CBPV), Sacbrood virus (SBV), Kashmir bee virus (KBV)들이 존재함을 확인한 바 있었다(최 등, 2010). 또한 김 등(2008)은 서양뒤영벌에서 Lysinibacillus fusiformis, Klebsiella oxytoca를 분리하고, 이들이 새로운 뒤영벌의 병원성 세균일 가능성을 제시하였다.
꿀벌 병원체 검색법의 경우, American foulbrood disease (AFB)의 원인균인 Paenibacillus larvae 그리고 주요 병원성 바이러스들, 즉 IAPV, CBPV, korean Sacbrood Virus (kSBV) 등에 대하여, 해당 병원체의 존재를 빠르게 확인하는 방법으로 초고속 실시간 PCR법을 개발되었으며(유 등, 2009; 유 등, 2010; Han et al., 2011), 꿀벌 주요 병원체의 종합적 검출을 위한 DNAchip도 개발된 바 있으나(Wang et al., 2016), 뒤영벌을 검사대상으로 하여 다양한 병원체를 동시에 검사할 수 있는 검출법은 제시된 바 없었다. 또한 근래에 대두되고 있는 국내 뒤영벌의 수출 추진에서도, 뒤영벌의 검역기준은 국내외로 정립된 바가 매우 미약한 수준이기에, 이를 명확하게 정립하기 위하여 뒤영벌 수출입을 위한 병원체 검사법, 또는 검역기준은 매우 긴요하다 할 것이다.
본 연구에서는 서양뒤영벌의 병원체들에 대한 표준검사법을 개발하고자 하였으며, 이를 뒤영벌 수출입을 위한 검역 기준으로 제시하고자 하였다. 검출대상 병원체들은 꿀벌 및 뒤영벌에서 교차 감염을 야기하는 8종의 병원체, 즉 BQCV, CBPV, DWV, IAPV, SBV, kSBV, P. larvae, Mellisococcus plutonius와 뒤영벌 특이 병원체(후보) 3종, 즉 SBPV, L. fusiformis, K. oxytoca를 선발하였으며, 이들에 대한 특이 primer쌍을 새로이 제작 또는 선발하여, 병원체 총 11종의 존재여부를 한 번의 시험으로 판정할 수 있는 초고속 유전자 검출법을 개발하고자 하였다.
재료 및 방법
시료의 확보 및 DNA와 RNA의 분리
뒤영벌 시료는 2016년 8월 경기도 광주, 전라도 보성, 경상북도 청송, 경상남도 밀양, 경상남도 창원에서 확보된 서양뒤영벌(Bombus terrestris)을 사용하였다.
Genomic DNA (gDNA)의 분리는 G-spin™ Total DNA Extraction Kit (iNtRON, Korea)의 시행방법을 따랐으며, 약술하면, 뒤영벌 시료 1마리를 2.0ml tube에 넣고 glass bead (pore size : 1mm, Germany)와 혼합 후, MagNa Lyser (Roche, Switzerland)로 분쇄하였다. 분쇄된 시료에 300μl lysis buffer를 넣고 15초 vortexing하여 혼합하였으며, 500μl binding buffer를 추가로 넣어 완전히 혼합한 후, 전용 column에 loading하였다. 이를 13,000 rpm에서 1분간 원심분리 한 후, collection tube에 나온 용액을 제거하였고, column에 500μl의 washing buffer 를 넣고, 13,000 rpm에서 1분간 원심분리 하여 세척하였다. 이후 column을 1.5ml 원심분리관으로 옮기고, 60μl의 증류수를 column에 넣어 부착된 gDNA를 용출시키고 13,000 rpm에서 1분간 원심분리로 DNA 용액을 수집하였다. 추출된 total gDNA를 spectrophotometer로 정량하였으며, -70°C에 보관하며 이후 실험에 사용하였다.
Total RNA 분리는 R&A Blue Kit (Takara, Japan)를 사용하여 제작자의 지시에 따라 수행하였다. 약술하면, 뒤영벌 시료 1마리를 2.0ml tube에 넣고 glass bead (pore size : 1mm, Germany)와 혼합한 후 1.0ml R&A Blue 시약을 넣고 MagNa Lyser (Roche, Switzerland)로 분쇄하였다. 분쇄된 시료에 200μl chloroform을 넣고 15초간 vortexing 하여 혼합하였으며, 4°C에서 13,000 rpm 10분간 원심 분리하여, 상등액만을 1.5ml tube에 옮겼다. 시료 상등액과 같은 부피의 isopropyl alcohol을 넣고 15초간 vortexing하여 혼합하였으며, 4°C에서 13,000 rpm, 10분간 원심 분리하고 상등액을 제거하였다. RNA 침전물은 0°C로 준비한 75% ethanol을 사용하여 washing 과정 진행한 후, DEPC 처리된 증류수를 사용하여 total RNA의 용액으로 확보하였다. 추출된 total RNA는 spectrophotometer로 정량하였고 -70°C에 보관하며 이후 실험에 사용하였다.
뒤영벌 특이 primer들의 제작 및 재조합 plasmid DNA 확보
뒤영벌 병원체 11종에 대하여 GenBank (NCBI)에서 염기서열 정보를 확인한 후 각각 한 쌍의 특이 primer들을 설계 하였고, 해당 PCR 산물의 길이는 정량 초고속 PCR에 적합하도록 120bp부터 250bp 사이가 되도록 설계하였다. 각 검출용 primer들은 설계대로 전문회사(Bionics, Korea)에 의뢰하여 제조하였다(Table 3).
뒤영벌 병원체로 의심되는 L. fusiformis는 ATCC 3454를 입수하여 배양하였고, genomic DNA를 추출하여 Glutamyl-tRNA amidotransferase 유전자의 부분을 PCR 증폭하고, pBX-vector에 클론화하여 표준기질로 사용하였다. 제작된 L. fusiformis특이 재조합 DNA는 pBX-L. fusiformis 159라 명명하였고, 이는 GenBank CP010820.1 (L. fusiformis full genome)의 4568067-4568195염기(길이 159염기)를 탑재하고 있다(Table 1).
또한, K. oxytoca는 ATCC 1686을 입수하여 배양하였고, genomic DNA를 추출하여 diol dehydratase-reactivating factor 유전자의 부분을 PCR 증폭하고, pBX-vector에 클론화하여 표준기질로 사용하였다. 제작 된 K. oxytoca 특 이 재 조 합 DNA는 pBX-K. oxytoca 208이라 명명하였고, 이는 GenBank AF017781의 2,065-2,272염기(길이 208염기)를 탑재하고 있다(Table 3).
한편, Slow bee paralysis virus (SBPV)의 경우, GenBank EU035616의 염기서열을 기준으로 330 특이 염기서열들을 설계하였으며, 이 염기서열의 제작에 필요한 long-nucleotide들은 전문회사(Bionics, Korea)에 의뢰하여 공급받았고, 혼합 신장 및 각 말단 primer에 의한 PCR로 합성 제조하였다. 증폭된 330염기의 PCR산물은 pBX-vector에 클론화하여 표준기질로 사용하였으며, 제작된 SBPV 특이 재조합 DNA는 pBX-SBPV-C3G이라 명명하였다. 이는 GenBank EU035616의 7,190-7,519염기(길이 330염기)를 탑재하고 있다(Table 2).
실시간 PCR 최적 조건 확립을 위한 온도 구배 실시간 PCR
뒤영벌 병원체 11종에 대한 각 primer 쌍의 PCR 조건에서 최적 annealing 온도 조건을 찾기 위해 온도구배 실시간 PCR (Temperature-gradient real-time PCR)을 수행하였다. 실시간 PCR은 2xfast RT-PCR premix(Nanohelix, Korea)를 사용하였고, 최종농도 각 0.5μM forward/reverse primer, 각 1.0×104 분자 수의 DNA가 주형으로 사용되었으며 총 20μl volume으로 수행하였다. 실시간 PCR은 Exicycler™ Quantitative Thermal Block (Bioneer, Korea)로 수행하였고, PCR 조건은 94°C, 3분간 pre-denaturation 진행한 후, 94°C, 10초간 denaturation, 42~62°C의 온도구배에 10초간 annealing, 72°C, 10초간 extension의 조건으로 40 cycle을 진행하였다. PCR 산물의 융점온도(melting temperature) 분석은 70°C에서 90°C까지 수행하였다. 뒤영벌 병원체 11종에 대한 각 최적 온도 조건 결과에서 Ct (threshold cycle) 그래프의 최종 형광 값 및 PCR 산물의 밀도를 측정하여 최적 조건을 확인하였고, 이들 자료는 그래프로 나타내었다.
병원체 검역을 위한 뒤영벌 gDNA 적정 사용량
뒤영벌 병원체 각 11종에 대하여 1×106부터 1×100 분자 수의 재조합 plasmid DNA를 주형 DNA로 하여, 이들을 서양 뒤영벌 gDNA 50ng이 포함된 것과 포함되지 않은 것을 실시간 PCR로 함께 또는 각각 수행하여 비교하였다. Real-time PCR 조건은 94°C, 3분간 predenaturation 진행하였고, 94°C, 10초간 denaturation, 53.7°C, 10초간 annealing, 72°C, 10초간 extension의 조건으로 40 cycle 진행하였다. PCR 산물의 융점 온도 분석은 60°C에서 90°C까지 1초 간격으로 1°C씩 증가시키며 형광 값을 측정하여 그래프로 나타내었고, 그래프를 미분화하여 peak 그래프를 확인하였다.
뒤영벌 병원체 특이 검출을 위한 각 primer 쌍의 융점 분석 및 검출 한계 측정
뒤영벌 병원체 각 11종에 대하여 각각 실시간 PCR 반응 후 증폭산물의 융점온도 분석을 실시하였다. 이 융점온도 분석도 60°C부터 90°C까지 1초 간격으로 1°C씩 증가하며 형광 값을 측정하여 그래프로 나타내었고, 그래프를 미분화하여 온도변화의 최고점을 확인하였다. 이 최고점은 Tm (Temperature of midpoint)와 같은 의미이며, 이들을 각각의 PCR 산물에 대한 Tm으로 제시하였다. 표준 기질에 대한 PCR 산물의 Tm값을 기준으로 비특이적 반응 산물을 구분할 수 있었다.
검출 한계 측정을 위하여, 뒤영벌 병원체 각 11종의 특이 유전자에 대한 재조합 plasmid DNA들을 spectrophotometer로 260nm의 흡광도에서 정밀 정량 측정하여, 각 DNA용액 중 표준기질 분자의 수를 계산하였다. 각 재조합 plasmid DNA는 1×106 분자부터 1×100 분자까지 10배씩 연속 희석하여 각 PCR에서 주형 DNA로 사용하였다. 연속 희석된 주형 DNA와 각 PCR에서 primer의 각 최종농도는 0.5μM이 되도록 하였으며, 2x fast RT-PCR premix (Nanohelix, Korea)를 사용하여 뒤영벌 병원체 특이 PCR의 검출한계를 측정하였다. 실시간 PCR의 반응액 조성 중 서양뒤영벌의 50ng gDNA를 추가하여 이하의 PCR 반응들을 수행하였다. Real-time PCR은 GenecheckerTM (Gene System Co., Korea)을 사용하였으며, 조건은 94°C, 1분 pre-denaturation을 진행하였고, 94°C, 10초 denaturation, 54°C, 10초 annealing, 72°C, 10초 extension 의 조건으로 40 cycle 진행하였다.
서양뒤영벌 검체에서 다중 실시간 PCR로 감염된 병원체 검출 및 판정
국내에 유통되고 있는 서양뒤영벌 시료의 병원체 감염여부를 판단하기 위하여, 경기도 광주, 전라도 보성, 경상북도 청송, 경상남도 밀양, 경상남도 창원에서 생산된 뒤영벌 각 1마리에 대하여 gDNA와 total RNA를 분리하였다. Total RNA 중 1μg을 complementary DNA (cDNA)로 합성하여 virus에 대한 검출을 시도하였고, gDNA, cDNA는 각 50ng을 사용하여 뒤영벌 병원체 BQCV, CBPV, DWV, IAPV, SBV, kSBV, SBPV, P. larvae, M. plutonius, L. fusiformis, K. oxytoca 등 11종에 대하여 다중 실시간 PCR (multiple real-time PCR)을 수행하였다. 각 뒤영벌 병원체에 대하여 각 0.5μM primer 쌍 (각 최종농도)이 사용되었으며, 총 20μl volume으로 수행하였다. 다중 실시간 PCR은 GenecheckerTM (Gene System Co., Korea)를 사용하였으며, PCR 조건은 94°C, 3분 pre-denaturation 후, 94°C, 10초 denaturation, 54°C, 10초 annealing, 72°C, 10초 extension의 조건으로 40 cycle을 진행하였으며, PCR 증폭산물의 융점분석은 70°C에서 90°C까지 수행하였다.
결과 및 고찰
뒤영벌 시료의 확보 및 DNA와 RNA의 분리
뒤영벌 성체 1마리는 무게가 70mg내외로 측정되었으며, 여기서 추출된 gDNA는 농도가 평균 114ng/μl 수준이었으며, 뒤영벌 1마리 당 평균 5.7μg의 DNA를 얻을 수 있었다. 한편 뒤영벌 성체(평균 70mg)로부터 추출된 total RNA는 평균 농도가 2.0μg/μl 수준이었으며, 평균 약 98μg을 얻을 수 있었다. cDNA의 제작은 2.0μg total RNA를 사용하여, 역전사 반응을 실시하였으며, 얻어진 20μl cDNA 중 1μl를 주형 DNA으로 사용하여 DNA 병원체와 같이 PCR에서 주형으로 사용하였다.
뒤영벌 병원체에 대한 특이 primer쌍 선발 및 표준 DNA
각 primer의 설계는 Genbank (NCBI)에 등록된 해당 유전자를 확인하고 특이성이 높은 것들을 선발 또는 재설계하였고, 그 특이성, 민감성, 정확성, annealing 온도 적합성을 기준으로 선발하였다(Table 3).
모든 병원균과 바이러스들의 통일된 PCR 검출조건을 알아내기 위하여, 각기 클론화된 표준 기질(standard template)이 필요하였으며, 결국 국내외로 감염체 확보가 특히 어려운 SBPV (Genbank, Accession No. EU035616)는 peptidase-c3 like 유전자에서, L. fusiformis (Genbank, Accession No. CP010820.1)는 Glutamyl-tRNA amidotranferase 유전자에서, K. oxytoca(Genbank, Accession No. AF017781)는 diol dehydratase-reactivating factor 유전자에서 각기 특이 유전자부위를 인공합성 또는 PCR 증폭물의 클론화를 통하여 확보할 수 있었다.
Bombus pathogen specific Real-time PCR의 최적 혼성 온도
뒤영벌의 11종 병원체 검출을 위한 primer쌍들을 사용한 최적 PCR 조건들을 확립하고자, 각각의 primer 쌍을 사용한 PCR에서 최적 annealing 온도를 구하였다. 실시간 PCR들의 결과에서, BQCV, DWV, SBV, SBPV, M. plutonius, L. fusiformis와 K. oxytoca의 경우는 각 특이 유전자가 42~62°C annealing 온도범위에서 최고량의 PCR 증폭산물을 만드는 것이 관찰되었으며, CBPV, IAPV, kSBV와 P. larvae들은 48~58°C annealing 온도에서 특이 PCR 증폭이 유효한 것으로 나타났다.
또한, 최적의 annealing 온도 조건은 각기 45°C(SBV), 52°C(SBPV) 53°C(BQCV, M. plutonius, K. oxytoca), 54°C(CBPV, IAPV, kSBV, P. larvae), 56°C(L. fusiformis), 57°C (DWV)로 다양히 측정되었으나, 하나의 조건에서 11종 병원체 검출을 위한 PCR의 annealing 온도는 53.2°C로 통합하였다. 각 annealing 온도의 허용범위와 함께 각 최적 온도는 세로선과 점으로 나타내었다(Fig. 1).
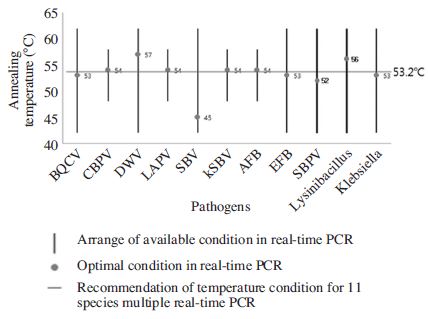
Optimal annealing temperature for detection of Bombus pathogens. Real-time PCR conditions for each target were optimized excluding annealing temperature. PCRs were performed using 42~62°C or 48~58°C as annealing temperatures. The template DNA was used for all real-time PCRs as same amount of 1.0×105 molecules of each specific target. Best annealing temperature was determined as 53.2°C for all PCRs against 11 pathogens (horizontal red line). Vertical line was indicated accepted temperatures for annealing in each successful PCR. Black point was best annealing temperature for each specific PCR.
이들 각 병원체에 대한 최적 annealing 온도는 개별 실시간 PCR에서 보다 유리할 수 있을 것이나, 본 연구의 목적인 11종 모두의 다중 실시간 PCR의 수행에 있어, 11종에 대한 PCR 모두가 단일 annealing 온도로 수행될 수 있다면 다수의 시료에 대한 검사를 수행할 경우 보다 유리할 것이다. 따라서, 각각의 표준 유전자 주형에서 동일한 수준의 분자 수(105 copies)를 사용한 실시간 PCR을 수행하였으며, 이의 Ct 값 분포를 고려하고 각 primer의 binding efficiency를 가름하여, 11종 전체의 PCR에서 각기 특이 유전자 검출에 가장 유리하다고 판단되는 annealing 온도는 53.2°C로 계산되었다(Fig. 1의 가로선). 11종 모두에 대한 annealing 온도로 53.2°C를 설정 하였을 때, BQCV, CBPV, IAPV, kSBV, SBPV, P. larvae, M. plutonius, K. oxytoca는 각 최적 annealing 온도를 사용하였던 PCR들과 유사한 결과를 보였으나, DWV, L. fusiformis는 각 PCR annealing 온도와 각 3.8°C, 2.8°C의 차이를 보이고, 또한 SBV의 경우는 8.2°C의 차이를 보인 만큼 최적 온도조건에서 벗어난 PCR 결과를 보였다. 이들의 경우 Ct 값에서 평균 1.06 cycles의 손실을 보였으나, 각 PCR 산물은 충분한 수준의 DNA 증폭이 나타나 이후의 분석에서 큰 불리함이 발견되지 아니하였다.
병원체 검역을 위한 뒤영벌 gDNA 적정 사용량
Real-time PCR법을 사용한 서양뒤영벌 검역에서 병원체 감염 시료로부터 DNA를 분리하였고, 정량화된 주형 DNA으로서 병원체 특이 유전자를 검출하고자 하였다. Real-time PCR 검출 한계 측정에서 서양뒤영벌의 gDNA를 각 50ng을 포함시켜 실시간 PCR을 수행하였고, 그 결과는 각 병원체 11종에 대한 특이 유전자의 증폭이 서양뒤영벌의 gDNA 유무와 관련없이 각기 특이 유전체들만 증폭됨이 확인되었으며, 각 Ct 값에서도 큰 불리함은 발견되지 아니하였다. 11종에 각각에 대한 PCR을 수행하였으며, kSBV-specific 유전자 검출 PCR의 경우, 50 ng gDNA의 존재 유무와 무관하게 1.0×102 분자까지무난히검출할수있었다(Fig. 2).
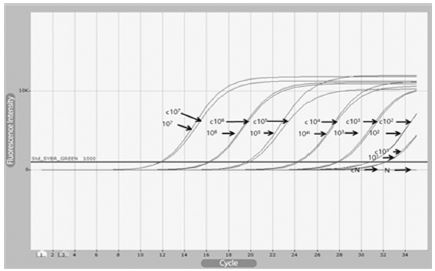
The limit of kSBV-specific detection with gDNA or without gDNA. Template DNA were serial diluted from 107 to 101 molecules of kSBV specific gene. kSBV-specific PCRs were performed using each diluted template, with and or without 50ng gDNA from Bombus for estimation of detection limit. “c” were indicated PCRs with 50ng chromosomal (genomic) DNA from Bombus. N is a negative control using deionized water instead of template DNA. kSBV-specific primer could detect 102 molecule of target successfully, with or without gDNA.
그러나 101 분자에 대한 특이 유전자의 증폭은 뒤영벌 gDNA에 의한 비 특이적 증폭산물의 간섭으로 특이유전자 증폭의 정확성이 떨어지는 것으로 관찰되었으며(융점 분석 결과), 100ng의 뒤영벌 gDNA을 사용하였을 경우, 이런 경향은 조금 더 높아지는 것으로 파악되었다(자료 미제시). 따라서 감염 의심 시료로부터 분리한 주형 DNA를 50ng 이하로 사용하는 것이, 뒤영벌 병원체의 특이 유전자 증폭에서 정확성과 민감성을 가장 높이는 조건으로 사료된다.
뒤영벌 병원체의 특이 PCR 산물에 대한 융점 분석과 검출 한계
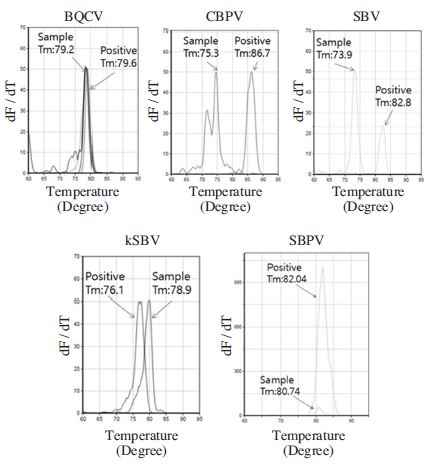
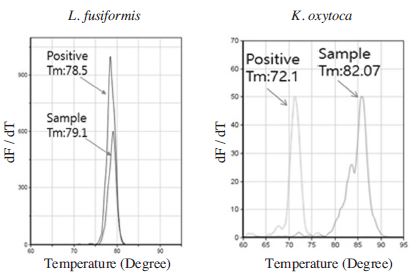
11종 뒤영벌 병원체 각각의 특이 유전자에 대하여 실시간 PCR에서 각 primer 쌍의 민감도를 측정하였다. 서양뒤영벌 gDNA 50ng이 포함되게 하였고, 각 특이 기질은 10배씩 연속 희석시켜 실시간 PCR을 수행하였으며, 다중 PCR을 위한, annealing 최적 온도 53.2°C (또는 53°C)를 사용하여 실시되었다. 특이 유전자 증폭 확인을 위하여 융점 온도 분석을 실시하였으며, 각각의 Tm에 따라 특이 유전자 증폭과 비 특이적 유전자 증폭을 구분 할 수 있었다. 검출 한계 측정에서 낮은 수준의 특이 분자 수(101~102)에 대한 유전자 증폭은 형광그래프에서 모두 증가하는 모양을 보였으나, Tm값 분석을 수행하여 비 특이적 반응 산물을 구분하고 검출 한계를 판단하였다. 뒤영벌로부터 각각의 병원체에 대한 특이 primer 쌍에 의한 증폭산물의 정성적 판단은 Tm값 분석에서 쉽게 비 특이적 반응 산물을 구별할 수 있었다. CBPV의 경우 비 특이적 증폭산물이 낮은 수준의 특이 분자 수(101~102)의 증폭 PCR에서 관찰되었으며, BQCV 특이 증폭산물의 Tm은 87.8°C이고, 비 특이적 증폭산물의 Tm은 78.5°C로 상호 9.3°C의 차이가 있어 쉽게 구별할 수 있었다(Fig. 3).
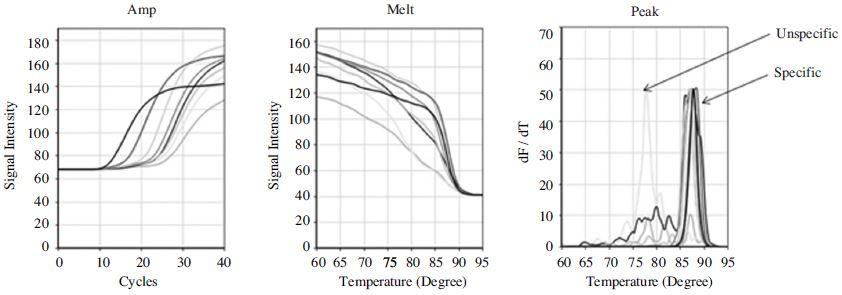
CBPV-Specific detection using CBPV-specific PCR and its Tm analysis. CBPV-specific DNA were amplified with standard condition using CBPV-specific real-time PCR. Target DNA were serial diluted from 106 to 101 molecules per PCR including 50ng Bombus gDNA. Deionized water were used as negative control. Tms were caculated as 87.8°C for specific targets and as 78.5°C for unspecific amplicon (101 molecules of CBPV-specific DNA). Amp, Melt, Peak were indicated changes of fluorescence in each PCR amplification, reassociation, and dF/dT, respectively. N, P, 106, 105, 104, 103, 102, 101 molecules of specific target.
BQCV, CBPV, DWV, IAPV, SBV, kSBV, SBPV, P. larvae, M. plutonius, L. fusiformis, K. oxytoca도 각기 같은 방법으로 특이 유전자 증폭산물의 Tm값의 측정하였으며, 각 병원체에 대한 Tm의 평균은 각각 79.21°C, 87.91°C, 81.98°C, 80.73°C, 83.26°C, 76.09°C, 81.82°C, 88.86°C, 86.24°C, 78.80°C, 72.50°C로 계산되었다. 이 과정에서 발견된 대표적 비 특이적 증폭산물도 Tm값을 측정하였으며, 특이 증폭산물의 Tm값과 함께 병기하였다(Table 4).

Tm values for the amplicons of specific targets and unspecific amplicons in Bombus pathogen-specific PCRs
본 연구인 뒤영벌 병원체 검출을 위한 다중 PCR 검출법은, 실험실적 표준 검사법으로 민감도를 측정하였으며, 이를 약술하면, 뒤영벌 시료의 total DNA와 total RNA를 추출하고, DNA 병원체에 대하여는 그대로 DNA 시료를 사용하며, RNA 병원체(RNA viruses)에 대하여 cDNA를 만들어 각기 11종의 PCR들을 동시에 진행하는 것이다. 본 검사법의 실험실적 민감도는 50ng 핵산을 각 PCR에 사용하고, 각 특이 기질을 계단 희석하여 각각의 PCR에서 증폭에 의한 검출 가능 범위를 측정하는 것이었다. 결과로써 DWV, SBV의 경우 1.0×101의 분자까지 특이적으로 검출할 수 있었으며, CBPV, IAPV, kSBV, SBPV 의 특이 증폭에서는 1.0×102의 민감도를 보였고, BQCV, P. larvae, M. plutonius, L. fusiformis, K. oxytoca에서는 1.0×103의 민감도를 보여 주었으며(Fig. 4), 이는 뒤영벌 gDNA 50ng의 존재 하에 각각의 병원체에 대한 특이 유전자 증폭 검출 한계를 의미한다.
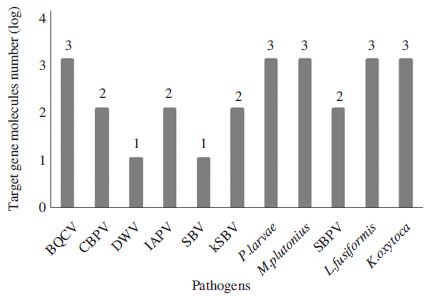
Detection limit of Bombus pathogen-specific real-time PCR. PCRs were performed with serially diluted, each specific DNAs including 50ng Bombus DNA. Accurate detections were available under condition of limited molecules of specific targets. “1”, “2”, and “3” were indicated 1.0×101, 1.0×102, and 1.0×103 molecules of specific targets, respectively. Each accuracy under these conditions was calculated over 0.98 in regression coefficient.
다중 실시간 PCR을 사용한 서양뒤영벌 시료의 병원체 검출 및 판정
국내에 유통되고 있는 서양 뒤영벌에서 뒤영벌 병원체의 존재여부를 밝히기 위하여, 경기도 광주, 전라도 보성, 경상북도 청송, 경상남도 밀양, 경상남도 창원에서 생산된 뒤영벌을 검출용 시료로 사용하였으며, 보통 뒤영벌 성체 1마리로부터 gDNA와 total RNA를 각기 분리하여 검사하였다. 뒤영벌 병원체 특이 실시간 PCR은 동일 시료에서 분리된 RNA에서 제작된 cDNA를 주형으로 11종의 PCR을 동시에 수행하는 것으로써, 병원체 특이 증폭산물을 확인하여 그 존재여부를 판정하였다.
먼저, 전라도 보성산 서양 뒤영벌에 대한 검사에서, BQCV-specific PCR은 BQCV-특이 산물을 형성하였고, 79.2°C의 정확한 Tm값을 보여주어 BQCV 양성(+)으로 판정되었으나, 70.0°C, 86.5°C의 Tm값을 가진 판정과는 무관한 작은 비 특이증폭도 나타났다. 이 BQCV-특이 증폭은 전기영동에서도 계산된 크기인 233bp와 일치하는 것으로 나타나 전기영동과 무관하게 다중 실시간 PCR에 의한 판정이 가능함을 보여주었다(자료 미제시). L. fusiformis 특이 PCR에서 특이 산물을 형성하였으며, 보성 시료에서 79.1°C의 Tm을 보여주어 양성(+)으로 판정하였다. 또한 CBPV, SBV, kSBV의 PCR에서도 증폭산물은 관찰되었으나, 각각의 표준산물의 Tm값과 다른 결과가 나와 비 특이증폭으로 판단되었다. 또한 K. oxytoca 특이 PCR에서도 증폭산물은 관찰되었으나, 각각 표준과 다른 Tm값을 보이고, 융점곡선이 다수의 비 특이증폭임을 나타내어, 쉽게 음성으로 판정할 수 있었다. P. larvae 증폭산물의 경우 Ct 그래프의 증폭 곡선도 약하게나마 확인이 되었고, Tm값도 88.5°C로 측정되어 P. larvae 특이 증폭으로 보이나, 극히 소량의 P. larvae 특이 DNA가 존재하였다판단하고, 음성(-*)으로판정하였다(Fig. 5; Fig. 6).
Tm and electrophoretic analysis of Bombus viral pathogensspecific real-time PCR using Bombus species produced from Boseong, Korea. cDNA originated from Bombus sample was used to perform 7 specific real-time PCRs. After PCR amplifications, melting temperature analysis were performed independently against 7 viral pathogens. BQCV-specific DNA (233 bp) was accurately detected based on Tm analysis. CBPV- (230 bp), SBV- (236 bp) and kSBV- (121 bp) specific DNAs were not found. With only these results, the existence of SBPV-specific DNA (224 bp) could not be determined.
Tm and electrophoretic analysis of Bombus bacterial pathogens-specific real-time PCR using Bombus species produced from Boseong, Korea. DNA originated from Bombus sample was used to perform 4 specific real-time PCRs. Melting temperature analysis were performed independently against 4 bacterial pathogens. It is determined that all 4 bacterial-specific DNAs (P. larvae 243 bp, M.plutonius 208 bp, L. fusiformis 129 bp, K. oxytoca 208 bp) were not amplified from Bombus sample.
또한, 경기도 광주의 서양뒤영벌에서는, BQCV, SBV, SBPV, P. larvae에 대하여 각기 DNA 증폭을 보여주었고, 각 융점분석에서 SBV 증폭산물은 83.0°C의 Tm값을 나타내어 양성(+)으로 판단되었으며, BQCV 증폭산물은 72.5, 80.5, 84.5°C의 Tm값을 보여 specific 유전자가 있는 것으로 판단되나, 확정검사가 요구되기에 양성의심(+/-)으로 판정하였고, SBPV 증폭산물의 경우 81.5°C의 Tm값을 나타내는 PCR산물이 관찰되었으나, 확정 검사가 요구되기에, 양성의심(+/-)으로 판정하였다(자료 미제시).
경상남도 밀양의 서양 뒤영벌에서는, SBV, SBPV의 증폭산물이 관찰되었으며, SBV 증폭산물 융점분석에서 83.5°C의 Tm값이 계산되어 양성(+)으로 판정하였으며, SBPV 증폭산물은 81.5°C의 Tm값을 나타내는 PCR 증폭산물이 관찰되었으나, 확정 검사가 요구되기에, 양성의심(+/-)으로 판정하였다(자료 미제시).
경상북도 청송의 서양뒤영벌에서는 SBV 증폭산물이 84.0°C의 Tm값을 나타내고, 이 또한 확정 실험을 요구하기에, 양성의심(+/-)으로 판정하였다. 경상남도 창원의 서양 뒤영벌에서는 양성의견을 보이는 병원체가 발견되지 아니하였다(자료 미제시).
국내 5개소에서 생산된 뒤영벌들에 대한 11종의 병원체 유전자 검색은 각각의 병원체 특이 유전자의 PCR 산물의 Ct값과 Tm값 그리고 전기영동에 의한 재확인과정을 통하여 판정하였으며, 모든 자료가 너무 방대하여 정리된 결과를 제시하지 아니하였다(자료 미제시).
한편, SBPV 증폭산물들은 광주, 밀양, 보성의 시료에서, 형광값이 크게 증가되는 것으로 관찰되었고, 각 증폭산물들에 대한 Tm 분석에서 각기 80.5°C 또는 81.5°C로 측정되었으며, 전기영동의 결과 예상 SBPV 증폭산물의 크기인 224 bp의 DNA 산물들이 약하게 관찰되었다(자료 미제시). 국내에 SBPV 존재는 아직 보고된 바 없으며, 본 연구를 위해 Oligonucleotide를 이용하여 DNA를 합성 후 재조합 DNA를 사용하였기에 본 연구의 후속 연구로써 보완되어야 할 것이다.
본 연구는 그간 주목을 받지 못하였던 서양 뒤영벌의 병원체들을 쉽게 모니터링하고 적절히 제어하기 위하여 해당 병원체들을 빠르고 쉽게 검출할 수 있도록 고안된 것이다. 서양 뒤영벌의 병원체들에 대한 관심은 이 화분매개 곤충의 수출입에 대한 관심과 함께 증대되고 있으며, 국내외로 미흡하였던 수출입 검역검사의 요구에 부응하는 것이라 할 것이다. 본 연구에서 제안하는 뒤영벌 병원체 특이 다중 실시간 PCR(Bombus pathogens specific multiple real-time PCR)법이 질병제어 및 검역현장에서 널리 사용되기를 기대한다.
적 요
DWV, IAPV, KBV, SBV, BQCV, kSBV, SBPV and Paenibacillus larvae, Mellisococcus plutonius, Lysinibacillus fusiformis, Klebsiella oxytoca의 뒤영벌 병원체들에 대한 다중 실시간 중합효소 연쇄반응법(PCR)을 개발하였다. 하나의 시료에서 추출된 핵산은 11종 PCR들에 같은 시간 및 조건으로 사용될 수 있으며, 각 병원체 특이 표적 DNA가 PCR 기질로 1000분자가 존재한다면, 해당 특이 PCR 증폭산물들은 정성적, 정량적으로 20분안에 성공적으로 증폭되었다.
우리가 제안하는 이 다중 PCR 검출법이 뒤영벌의 국제교역을 위한 검역검사에 사용되기를 기대한다.
Acknowledgments
본 연구는, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(과제번호 115058-02, 과제번호 115102-03), 농생명산업기술개발사업(312027-03) 및 수출전략기술개발사업(과제번호 115067-02)과 2017학년도 경기대학교 대학원연구원장학생 장학금 지원에 의하여 수행되었음.
References
- 김원태, 최지영, 김종길, 황석조, 최영철, 한상미, 김미애, 윤형주, (2008), 서양뒤영벌(Bombus terrestris)과 호박벌(B. ignitus)에 대한 병원성 세균 분리, Journal of Apiculture, 23(1), p13-20.
- 유미선, 김동수, 김일욱, 권순환, 윤병수, (2009), Israel Acute Paralysis Virus의 진단을 위한 Real-Time PCR 진단법의 개발, Journal of Apiculture, 24(1), p31-36.
- 유미선, 최용수, 박용하, 윤병수, (2010), Chronic Bee Paralysis Virus의 진단을 위한 Real-Time PCR 진단법의 개발, Journal of Apiculture, 25(1), p31-37.
- 윤형주, 김삼은, 이경용, 이상범, 박인균, (2008), 온도처리에 따른 뒤영벌 일벌과 여왕벌의 생산 효과, J, Apiculture, 23(4), p283-287.
- 최용수, 이만영, 이명렬, 김혜경, 김남숙, 홍인표, 변규호, 이광길, 윤형주, (2009), 호박벌(Bombus ignitus)에서의 Deformed Wing Virus(DWV)진단, Journal of Apiculture, 24(2), p83-91.
- 최용수, 이만영, 홍인표, 김남숙, 김혜경, 변규호, 윤형주, (2010), 뒤영벌류(Bombus terrestris와 Bombus ignitus)에서 꿀벌바이러스 진단, Journal of Apicultrue, 25(4), p259-266.
- Dafni, A., (1998), The threat of Bombus terrestris spread, Bee World, 79, p113-114.
- Dino, P. M., M. A. Furst, J. Caspar, P. Theodorou, M. J. F. Brown, and R. J. Paxton, (2015), A sting in the spit: widespread cross-infection of multiple RNA viruses across wild and managed bees, Journal of Animal Ecology, 84, p615-624.
- Han, S. H., Y. S. Choi, and M. L. Lee, (2011), Development of Highly Specific Quantitative Real-Time PCR Method for the Detection of Sacbrood Virus in Korean Honeybees, Apis cerana, J. Apiculture, 26(3), p233-240.
- Masahiro, M., (2000), Pollination of crops with bumblebee colonies in Japan, Honeybee Sci, 2, p17-25.
- Min, S. H., J. H. Wang, S. J. Lim, C. W. Lee, and B. S. Yoon, (2016), Development of Quantitative Real-time Recombinase Polymerase Amplification (qRT-RPA) Method for Quantitative Detection against Pathogenic Virus in Honeybee (Korean), Journal of Apiculture, 31(2), p147-156.
- Ruijter, A. D., (1996), Commercial bumblebee rearing and its implications, In: KW Richards (ed), Proceedings of the 7th international symposium on pollination. ISHS Acta Horticulturae Belgium, 437, p261-269.
-
Velthuis, H. H. W., and A. Van Doorn, (2006), A century of advances in bumble bee domestication and the economic and environmental aspects of its commercialization for pollination, Apidologie, 37, p421-451.
[https://doi.org/10.1051/apido:2006019]

- Wang, J. H., D. B. Lee, S. J. Ku, M. C. Peak, S. H. Min, S. J. Lim, C. W. Lee, and B. S. Yoon, (2016), Development of a Detection Method against 11 Major Pathogens of Honey Bee using Amplification of Multiplex PCR and Specific DNA-chip (Korean), Journal of Apiculture, 31(2), p133-146.
-
Yang, X., D. L. Cox-Foster, (2005), Impact of an ectoparasite on the immunity and pathology of an invertebrate: evidence for host immunosuppression and viral amplification, Proceedings of the National Academy of Sciences of the United States of America, 102(21), p7470-7475.
[https://doi.org/10.1073/pnas.0501860102]
