
초고속 유전자 증폭법을 이용한 벌집꼬마밑빠진벌레 (Aethina tumida)의 신속한 검출 기법 개발
Abstract
For the Rapid detection of small hive beetle (SHB; Aethina tumida) and for the mass-survey against SHB invasion, SHB-specific ultra-rapid PCR system was developed. Three different pairs of Aethina tumida-specific primers were deduced from cytochrome oxidase subunit I (COI) gene in mitochondrial DNA of SHB. Using optimized SHB-specific ultra-rapid PCR, 2.1×101 molecules of COI gene belonged to SHB could be detected specifically and quantitatively within 18 minutes 40 seconds. For the purpose of the application in apiary field, a DNA extraction method from bee debris was separatedly developed. When 105 SHB-specific COI molecules (1/1000 body of SHB larvae) are existed in 1g of bee debris, it could be verified inner 10 minutes as qualitative and quantitative manner. SHB-specific ultra-rapid PCR we proposed would be expected to apply widely, either in apiary field or laboratory, for the rapid detections and the control against SHB-invasion.
Keywords:
Ultra-Rapid PCR, Small Hive Beetle, SHB, Parasites, Honeybee서 론
벌집꼬마밑빠진벌레(Small Hive beetle; SHB; Aethina tumida Murray)는 아프리카에서 처음 발견되었으나, 1998년 미국의 플로리다에 상륙한 이래(Thomas, 1998), 미국 전역으로 퍼지며 피해를 증가시켜 세계적으로 주목 받게 된 꿀벌의 기생곤충이다. 현재 미국, 호주, 남아프리카 등에서 큰 피해를 입히고 있으며, 2014년 이탈리아에서 그 존재가 발견되어, 유럽의 양봉산업을 위협하고 있다.
벌집꼬마밑빠진벌레가 양봉산업에 미치는 피해는 우선 벌꿀을 산폐시키는 효모 Kodamaea ohmeri의 숙주라는 점이며, 이 효모는 벌집꼬마밑빠진벌레 유충의 배변을 통하여 벌꿀에 전달되며, 벌꿀을 발효시켜 산폐되게 한다(Benda et al., 2008). 또한 벌집꼬마밑빠진벌레는 꿀벌의 병원성 바이러스인 Deformed Wing Virus (DWV)의 숙주로써 꿀벌의 질병을 야기할 수 있음이 보고된 바 있다(Eyer et al., 2008).
2016년 9월 경상남도 밀양 및 창령에서 국내 최초로 벌집꼬마밑빠진벌레의 서식이 확인되어, 결국 벌집꼬마밑빠진벌레는 국내의 문제가 되었으며, 이들로부터 국내 양봉산업을 보호하기 위한 대책을 시급히 강구하게 되었다(윤병수, 2017).
벌집꼬마밑빠진벌레의 적절한 제어는 세계적으로 매우 어려운 난제이며, 성공적인 침입저지의 예도 자연적 소멸인지 방제를 위한 노력의 결과인지 애매한 경우가 많다. 미국의 경우 1998년 플로리다에 상륙한 것을 확인하고, 적절한 대비가 미흡했던 이유로 10여 년에 걸쳐 미국전역에 이 기생해충을 널리 퍼지게 하였으며, 현재도 벌집꼬마밑빠진벌레의 성충만을 포집하는 유인 Trap들로 개체수 저감에 노력하는 것이 주요 대책으로 파악된다.
국내의 경우, 2016년 9월 일부 지역의 양봉에서 이들이 발견된 것은, 시기적으로, 아직 국내 벌집꼬마밑빠진벌레의 박멸에 희망이 있다 할 것이다. 이들은 벌집에서만 산란과 유충의 서식이 가능하다는 것은 큰 약점이며, 국내의 관리 양봉의 장점을 살려, 매 봉군을 철저히 검색하고, 구제해 나간다면 꿀벌응애의 국내 전파 이래 또 다른 가장 위험한 꿀벌 기생해충인 벌집꼬마밑빠진벌레를 박멸할 수 있을 것으로 기대한다.
이를 위하여, 각 봉군에 대한 벌집꼬마밑빠진벌레의 존재 유무 판정은 이들의 구제에 가장 필수적인 판단이 될 것이나, 육안관찰 및 포집기의 사용만으로는 알, 초기 유충, 숨어있는 소수의 성충들에 대한 존재여부의 판정은 매우 부정확한 것으로 알려져 있다(Murihas, 2004; Spiewok and Neumann, 2006).
이런 단점을 보완하기 위한 방법이 봉변을 이용한 벌집꼬마밑빠진벌레의 특이 유전자 검출이 될 수 있을 것이며, 근래 크게 적용범위를 넓혀나가고 있는 초고속 PCR법은 벌집꼬마밑빠진벌레의 특이 유전자 발견에 크게 도움이 될 것으로 생각되었다.
초고속 PCR법은 PCR의 각 회전(cycle)중 각 단계(step; 변성, 혼성, 중합)의 반응시간을 최소화 한 것으로, 보통 40회전의 PCR에서 10분 이내에 목적한 특이 증폭산물을 확인할 수 있게 하였으며, 계속 그 적용범위를 확대해 가고 있다(Lee et al., 2007; Han et al., 2008; Yoo et al., 2012).
한편, 미토콘드리아 유전자 COI(cytochrome oxidase subunit I) 유전자는 진핵생물의 분자계통연구에 사용될 정도로 잘 알려진 유전자이며(Evans et al., 2000; Evans et al., 2002), 또한 1개 세포는 다수의 미토콘드리아를 가진다는 점, 그리고 이 유전체(mitochondrial DNA; mtDNA)는 환형이라는 점이, 환경 DNA(eDNA; environmental DNA)의 검출에서 이 유전자가 타 유전자보다 보존성에서 유리한 것으로 사료되며, 벌집꼬마밑빠진벌레 특이 COI 유전자를 검출의 표적으로 사용한다면, 1개 봉군의 봉변이라는 미소환경(Microenvironment)에서 벌집꼬마밑빠진벌레의 존재를 능히 검출할 수 있을 것이라 판단되었다.
사실, Ward 등(2007)은 봉변으로부터 벌집꼬마밑빠진벌레 특이 염기서열을 실시간 PCR (Real-Time PCR)로 증폭시켜, 이들의 존재여부를 판정하는 방법을 제시한 바 있었으며, 이 방법의 유용성을 입증하였다. 이들의 방법은 실험실적 검사로써 우수하였으나, 양봉현장에서 시험을 실시하기에는 긴 실험시간이 요구되는 등 그 적용에 한계를 보인 바 있었다.
따라서, 본 연구는 초고속 정량 PCR법을 적용하여 실험실 및 양봉 현장 실험이 모두 가능한 검출법을 추구하고, 환경 DNA의 개념에서 보다 특이성을 높인 새로운 SHB 특이 초고속 정량 PCR법을 개발하여 각 봉군마다 벌집꼬마밑빠진벌레의 존재 여부를 보다 정확히 판정할 수 있는 방법을 개발하고자 하였다.
재료 및 방법
벌집꼬마밑빠진벌레 시료 확보 및 genomic DNA의 순수 분리
벌집꼬마밑빠진벌레(SHB)는 순천대학교 홍기정 교수가 직접 채집한 국내 침입 SHB의 유충을 사용하였으며, 확보된 SHB 유충는 90% EtOH에 침전시켜 -20°C에서 냉동보관 하며 실험에 사용하였다.
COI 유전자의 확보를 위하여 SHB의 genomic DNA(gDNA)를 순수분리하였으며, 이는 유충을 막자사발에 넣고 액체질소로 동결시킨 후 분쇄하고, 분쇄액을 DNeasyⓇ Blood & Tissue kit (Qiagen Co, Germany)를 사용하여 제작사의 지시에 따라 순수분리과정을 진행하였다. 순수분리된 gDNA는 COI 유전자가 위치한 mtDNA가 포함된 것으로, Biophotometer (Eppendorf Co, Germany)를 사용하여 DNA를 정량한 후 -70°C에서 보관하였다.
꿀벌(Apis mellifera)의 gDNA는 경기대학교 양봉장에서 수집한 유충으로부터 추출하였으며, 이는 본 SHB특이 유전자 증폭에서 대조 기질로 사용하였다. 추출법은 SHB의 방법과 동일하였다.
봉변으로부터 SHB COI 유전자 검출
SHB가 오염되지 않은 봉변(Bee debris)은 경기대학교 양봉장으로부터 서양종 꿀벌의 벌통 바닥에서 수거하였으며, 봉변 2g에 SHB 유충 1마리를 넣고 액체질소를 이용하여 막자사발에서 곱게 분쇄하여 DNA의 순수분리를 시작하였다. 분쇄한 SHB 애벌레 혼합 봉변 시료는 DNeasyⓇ Blood & Tissue kit (Qiagen Co, Germany)를 이용하여 gDNA를 순수 분리하고, -70°C에서 보관하였다.
2g의 봉변과 SHB 유충 1마리에서 추출된 혼합 gDNA (mtDNA 포함)의 총량은 3.59μg이었으며, 농도는 37ng/μl로 측정 되었다. 본 연구에는 30ng의 혼합 gDNA를 사용하여 COI 유전자의 특이 증폭 반응들을 확인하였다.
특이 primer 제작 및 특이 재조합 plasmid DNA 확보
본 연구에서 사용된 SHB에서 SHB 특이 COI 유전자를 확보하기 위하여 클론화(molecular cloning)를 수행하였다(Table 1).
GenBank의 Database를 기반으로 SHB COI 유전자의 공통염기서열을 추론하였으며, 그 크기는 683bp가 되도록 설계하였다. 설계된 primer들은 각 SHB-COI F/R로 명명하였으며, 전문회사인 바이오닉스사에 제작을 의뢰하였다(Table 2).
제작된 SHB-COI F/R 과 국내 채집된 SHB의 gDNA를 사용한 일반 PCR에서 683 염기크기의 PCR산물을 얻을 수 있었으며(자료 미제시), 이는 T-vector cloning을 통하여 클론화를 수행하였고, 얻어진 클론은 pTop-SHB COI라 명명하였다. pTop-SHB COI에 삽입된 염기서열은 전문회사인 (주)솔젠트에 의뢰하여 염기서열을 결정하였으며, 이는 GenBank에 기 보고된 KP134137(미국산 SHB, 2014)과 가장 높은 유사도를 보였다(683염기 중 682염기 일치 자료 미제시).
얻어진 염기서열 정보를 기반으로 초고속 PCR용의 primer 2쌍을 설계하였으며, 이 과정에서 NCBI 사이트의 primer Blast 프로그램을 통해 검출의 특이성을 확인하였고, 그 결과로써 특이적으로 SHB 만 검출이 가능하며, 서양종 꿀벌(Apis mellifera) 및 토종벌(Apis cerena), 그리고 정년기(2013)가 제시한 벌집 기생해충인 큰꿀벌부채명나방(Galleria mellonella L.), 작은꿀벌부채명나방(Achroia grisella F.), 또한 포식해충인 장수말벌(Vespa mandarinia Smith), 왕잠자리(Anax parthenope Julius), 왕사마귀(Tenodera aridifolia Stoll), 고등털개미(Lasius niger) 등도 검출되지 않을 것임을 유전정보 상으로 확인하였다.
또한, Ward 등(2007)이 제시한 SHB 특이 primer 쌍도 pTop-SHB COI이 PCR 기질로 사용 가능함이 확인되어, 별도 제작하여 본 연구에서 비교하였으며, 초고속 PCR의 최적 조건을 점검하기 위하여, 또한 봉변에 존재하는 꿀벌 특이 유전자들의 정량측정을 위하여, 꿀벌의 β-actin 유전자와 40s Ribosomal Protein S18 (RPS) 유전자의 증폭을 위한 primer를 별도 설계하였다. 각 primer의 제작은 전문회사인 바이오닉스사에 의뢰하여 제작하였다(Table 2).
SHB COI 유전자의 초고속 검출을 위한 최적 annealing 온도
SHB COI 특이 초고속 증폭을 위한 최적조건을 구하고자, 각 primer의 최적 annealing 온도를 측정하였다.
이는 55°C±10°C의 범위에서 온도구배 PCR을 통하여 수행하였으며, 각 반응 당 재조합 DNA 주형인 pTop-SHB COI를 2.09×106 분자(약 0.003 femtomole=약 3 attomole)를 사용하였으며, 각 primer의 최종농도는 0.5μM, 10μl 2xGreenstar qPCR Master Mix (Bioneer, Korea)를 사용하여, 최종 20μl PCR 반응액으로 조성하였다.
PCR 증폭 및 형광값 측정은 ExicyclerTM Quantit ative Thermal Block (Bioneer, Korea)기기에서 실시하였으며, 초기변성은 95°C 5min으로 실시하였고, 이후 변성 95°C 30초, annealing 각 55°C±10°C에서 각 30초, 신장 72°C 30초를 1회전으로 하여 총 35회전 조건을 수행하였다.
결과는 Ct (Threshold Cycles)값 및 형광 증폭 값의 비교를 통하여 각 primer들의 최적 annealing 온도를 추론하였으며, 이 온도 조건은 Genechecker™(Genesystem, Korea)를 사용한 초고속 PCR에서 검정하였다. 초고속 PCR은 각 반응 당 pTop-SHB COI 2.09×106 분자를 사용하였으며, 각 primer의 최종농도는 1μM, 5μl 2×RapiMix (Genesystem, Korea)를 사용하여, 최종 10μl PCR 반응액으로 조성하였다. 초고속 PCR의 조건은 초기 변성 95°C, 30초 후, 변성 95°C 5초, annealing 각 52~60°C 각 5초, 신장 72°C 5초를 1회전으로 하여 총 40회전을 실시하였다. 이후 온도별 Ct값 및 형광 증폭그래프 비교를 통해 공통 최적 annealing 온도를 정립하였다.
SHB COI 유전자의 초고속 검출을 위한 최적 primer 최종 농도
SHB COI 특이 초고속 증폭을 위한 최적조건 중 최적 primer의 농도를 확인하고자, 각 primer의 최종 농도를 10μM, 5μM, 2μM, 1μM, 500nM 로 조성하여 초고속 PCR을 수행하였다.
각 반응액은 10μl로 조성하였으며, 각 반응 당 pTop-SHB COI 2.09×106 분자, 2×RapiMix에 다양한 농도의 각 primer쌍을 넣고 초고속 PCR을 시행하였다. 이때 조건은 초기 변성 95°C, 30초 후, 변성 95°C 5초, annealing 각 52~60°C 각 5초, 신장 72°C 5초를 1회전으로 하여 총 40회전이었다.
검출용 primer 3쌍에 의한 최소 검출 시간 및 검출 한계 측정
SHB 특이 염기서열을 탑재한 pTop-SHB COI을 2.09×106 분자부터 2.09×100 분자까지 연속 정량 희석하여 각 초고속 PCR의 주형으로 사용하였으며, 2×RapiMix를 사용하였고, 각 primer에 대해 확립 된 최적 농도의 Forward 및 Reverse primer를 사용하여 총 10μl PCR 반응액으로 조성하였다.
초고속 PCR 증폭 반응은 초기 변성 95°C, 30초로 고정하고 총 회전수를 50회전에서 진행하였으며, 각 회전의 각 단계, 즉 변성, annealing, 신장의 각 온도를 95°C(고정), 각 primer의 최적 annealing온도(52~60°C), 72°C(고정)의 온도조건으로, 회전 중 각 단계를 모두 3초, 2초, 1초로 감소시키면서 초고속 PCR에 필요한 최소 증폭시간과 검출한계를 동시에 측정하였다.
꿀벌의 gDNA상에서 검출 primer 쌍의 증폭 확인
꿀벌의 gDNA에서 SHB 특이 각 primer쌍의 유전자 증폭여부를 확인하기 위해 100ng의 꿀벌 gDNA를 사용하였으며, 이 초고속 PCR에서는 SHB 특이 primer 3쌍에 의한 PCR외에, 꿀벌의 고유 유전자인 rDNA와 β-actin1을 증폭시키는 특이 primer를 사용하였다.
각 반응액은 10로 조성하였으며, 각 반응 당 pTop-SHB COI 2.09×106 분자 또는 100ng 꿀벌 gDNA를 주형으로써 사용하였고, 최적 농도의 각 primer쌍, 2×RapiMix로 초고속 PCR을 시행하였다. 조건은 초기변성 95°C, 30초 후, 변성 95°C 2초, annealing 각 52~60°C 각 2초, 신장 72°C 2초를 1회전으로 하여 총 40회전 수행하였다.
꿀벌의 gDNA와 SHB 특이 유전자의 혼합물을 주형으로 한 초고속 PCR
꿀벌 gDNA와 SHB특이 염기서열을 탑재한 pTop-SHB COI를 정량 혼합하여, 초고속 PCR의 기질로 사용하였으며, 이로써 민감도와 정확성을 측정하였다. 반응 당 꿀벌 gDNA는 100ng씩을 고정적으로 사용하였으며, pTop-SHB COI를 2.09×106 분자부터 2.09×100분자까지 연속 희석하여 주형으로 사용하였다. PCR 조성은 100ng 꿀벌 애벌레의 gDNA와 2.09×106 분자 - 2.09×100 분자의 재조합 DNA를 사용하였으며, 2×Rapi master Mix, 최적 Primer 농도로 조성하였으며, 조건은 초기 변성 95°C, 30초 후, 변성 95°C 2초, annealing 각 52~60°C 각 2초, 신장 72°C 2초를 1회전으로 하여 총 50회전으로 초고속 PCR을 시행하였다.
봉변-SHB시료부터 SHB COI 특이 유전자의 초고속 정량 검출
SHB-특이 정량 PCR의 현장 적용 능력을 살피기 위하여 SHB를 인위적으로 오염시킨 봉변 시료를 제조하였다. 2g의 봉변과 1마리의 SHB유충으로부터 추출된 혼합 gDNA는 총 3.59μg이었으며, 이중 30ng을 SHB 특이 초고속 정량 PCR의 초기 기질로 사용하였다. 또한 이 total DNA를 단계별로 희석하여 SHB 특이 초고속 PCR에 적용하였으며, 어느 수준의 SHB의 사체가 봉변에 혼합되어 있을 때 이의 존재 입증이 가능한지를 측정, 계산하였다. Ward 등(2007)은 자연적 조건에서 SHB가 발견된 봉군의 봉변에서 PCR검사로써 SHB-특이 유전자의 존재를 밝힌 바 있었으나, 그 양을 다만 Ct값(29.69 cycles)로 제시하였다.
한편, 꿀벌의 gDNA가 봉변에서 어느 수준으로 검출되는지를 확인하고자 꿀벌의 고유 유전자인 RPS 및 β-actin gene을 초고속 정량 PCR로 증폭시켜 양의 측정 및 정량 계산을 수행하였다.
결과 및 고찰
SHB COI 유전자의 초고속 검출을 위한 최적 혼성 온도
SHB 특이 검출용 primer 3쌍에 대한 최적 annealing 온도는 실시간 PCR(Real-Time PCR) 기기인 ExicyclerTM Quantitative Thermal Block (Bioneer CO., Korea)을 사용한 온도구배 PCR로 수행하였다.
SHB 특이 검출용 primer SHB-DP F1/R1쌍, SHB-DP F2/R2쌍의 경우, 45~65°C 범위에서 비교적 고른 증폭산물이 관찰되었으며, 최적의 온도 범위는 SHB-DP F1/R1 primer 쌍의 경우 56°C±4°C에서, SHB-DP F2/R2 primer 쌍의 경우 54°C±4°C 범위 이었다. SHB 207F/315R primer 쌍의 경우 57°C±3°C 범위가 최적 annealing온도로 측정되었기에, 3쌍의 primer들은 52~60°C의 annealing온도 범위에서 초고속 PCR을 위한 최적 annealing 온도를 구하고자 하였다(자료미제시).
초고속 PCR은 Genechecker™(Genesystem, Korea)를 사용하여 수행되었으며, 52~60°C의 annealing온도 범위에서 Ct값과 형광의 최고점을 기준으로 평가하였다(Table 3).
SHB-DP F1/R1 primer 쌍의 경우 56°C에서 가장 빠른 Ct값 및 최종 형광값을 보였으며, 54°C에서도 증폭 양상이 비슷하게 나타나는 것으로 확인되었다. 또한 융점 분석을 통해 74.91±0.33℃에서 특이적인 유전자 증폭산물이 나타났다. SHB-DP F2/R2 primer 쌍의 경우 54°C에서 가장 빠른 Ct값 및 최종형광값을 나타내었으며, 융점 분석을 통해 77.02±0.16°C에서 특이적인 증폭산물이 나타났음을 확인하였다. 또한 SHB 207F/315R primer 쌍에서 Ct값이 54, 56°C에서 Ct값이 거의 동일하게 나오나 최종 형광값 비교에서 56°C의 것이 조금 우세한 것으로 판정되었다.
초고속 PCR의 조건에서 3쌍의 SHB 특이 primer쌍들의 공통 annealing온도는, 각 primer쌍들의 최적온도들을 감안하여, 54°C로 설정하였다.
SHB COI 유전자의 초고속 검출을 위한 최적 primer 최종 농도
초고속 PCR 방법에 적합한 각 primer 농도를 확립하기 위하여 500nM(5pmole/μl), 1μM(10pmole/μl), 2μM(20pmole/μl), 5μM(50pmole/μl) 10μM(100pmole/μl)의 최종 농도로 조성하여 초고속 PCR을 진행하였다(Table 4).
최적 primer 농도는 각기 3회 반복하여 결과를 얻었으며, 측정 된 각 농도별 Ct값 및 최종 형광 값 그리고 Melting graph에서 비특이적 증폭 양상을 보이지 않는 조건에서 최적 농도를 결정하였다.
이에 SHB-DP F1, R1 primer 쌍은 1μM 최종 농도를 사용하였을 때 유리한 Ct값 및 높은 최종 형광값을 나타내어 최적 primer 농도로 설정하였으며, SHB-DP F2, R2 primer 쌍의 경우 2μM에서 가장 빠른 Ct값 및 높은 최종 형광값을 나타내었다. SHB 207F, 315R primer 쌍은 2μM에서 Ct값 및 최종 형광값 비교를 통해 10μM, 5μM Primer의 농도에서 Ct값에서는 유리함을 보이나 형광값 확인에서 낮게 측정되며, 1μM, 0.5μM의 낮은 Primer농도에서는 최종 형광값은 높게 측정이 되나 Ct값이 20cycle 이상으로 불리함을 보기에 2μM을 최적 농도로 설정하였다.
SHB 특이 검출용 primer 3쌍에 의한 최소 검출 시간 및 검출 한계 측정
3쌍의 primer에 대해 민감도를 확인하고자 앞서 확립된 최적 annealing 온도 및 primer 농도에서 재조합 DNA 주형을 2.09×108 분자부터 2.09×100 분자까지 연속 희석하여 검출 한계를 측정하였다.
PCR 조건은 각 회전 당 변성, 혼성, 신장 각 단계를 2초, 2초, 2초의 시간을 주어 실시하였으며, 총 3회 반복을 통해 Ct 및 Tm값의 평균과 표준편차 값을 구하였다. 이때 PCR 반응은 총 50회전이 총 18분 40초 동안 진행되었다(Fig. 1).
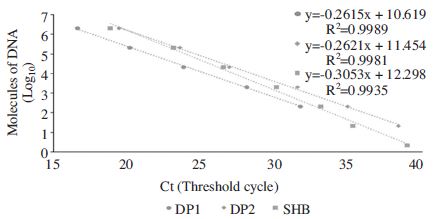
Detection limit and Tm using three primer pairs for SHB COI specific Ultra-rapid quantitative PCR. Ultra-rapid PCR with each primer pairs was performed using optimal condition. pTOP-SHB COI was used as template in PCR that was diluted from 2.09×106 to 2.09×100 molecules /PCR, respectively. 2.09×101 molecules template were amplified successfully by UR-PCR using SHB DP F2/R2 pairs and SHB 207F/315R pairs, except SHB DP F1/R1 pairs.
SHB-DP F1/R1 primer 쌍을 사용한 초고속 정량 PCR에서, 주형 2.09×102 분자이상이 있을 경우 정확히 정량적 증폭이 됨을 확인하였으며, 이는 형광의 증폭 곡선을 이용한 Ct값 측정, 그리고 Ct값과 주형 분자수를 사용한 회귀식 (Regression equation) Y=-0.2615X+10.619 및 회귀상수(Regression coefficient; R2)가 0.9989로 입증할 수 있었다. 사실, 2.09×101 분자의 주형에서도 총 3회반복실험중 1회는 검출되는 것으로 나타났다. Tm (Temperature of mid-point)은 76.0°C로 측정되었으며, 2.09×100 분자 및 주형을 넣지 않은 대조군에서는 Tm이 각 73.3°C, 73.0°C로 나타나, 특이적 증폭 산물의 Tm과 차이를 보였다.
SHB-DP F2/R2 primer 쌍을 사용한 초고속 정량 PCR에서, 주형 2.09×101 분자이상이 있을 경우 정확히 정량적 증폭이 됨을 확인하였으며, 이 역시 회귀식 Y=-0.2621X+11.454와 회귀상 수, R2=0.9981로 입증할 수 있었다. 이때 Tm은 77.7°C로 측정되었으며, 2.09×100 분자 및 주형을 넣지 않은 대조군에서는 Tm이 각 74.3°C, 72.3°C로 나타나, 특이적 증폭 산물의 Tm과 차이를 보였다.
SHB 207F/315R primer 쌍을 사용한 초고속 정량 PCR에서, 주형 2.09×101 분자이상이 있을 경우 정확히 정량적 증폭이 됨을 확인하였으며, 이 역시 회귀식 Y=-0.3053X+12.298과 회귀상 수, R2=0.9935로 입증할 수 있었다. Tm은 77.6°C로 측정되었으며, 주형을 넣지 않은 대조군에서는 Tm이 69.0°C로 나타나, 특이적 증폭 산물의 Tm과 차이를 보였다(Table 5).
꿀벌의 total DNA에서 SHB검출용 및 꿀벌 DNA검출용 primer 쌍들의 차이
SHB 특이 검출용 3쌍의 primer들과 꿀벌 DNA 특이 primer 쌍들을 사용하여 100ng의 꿀벌 gDNA의 존재하에 주형으로 초고속 유전자 증폭을 실시하였다.
RPS 유전자는 꿀벌의 gDNA 중의 해당 유전자를 정확하게 증폭시켰으며, RPA-재조합 DNA를 주형으로한 PCR산물과는 약간의 Tm값의 차이를 보였으나, 다량의 꿀벌 gDNA의 존재 여부에 따른 작은 차이로 해석되었다.
SHB 특이 3쌍의 primer쌍을 사용하여 꿀벌 gDNA를 주형으로한 초고속 PCR에서 약간의 비특이적 증폭을 관찰할 수 있었으나, 이들은 모두 비특이적 증폭의 형광값을 보였으고, 또한 모든 비특이적 산물의 Tm 값은 특이 주형을 사용한 초고속 PCR의 산물들과 각기 3°C 이상의 분명한 차이를 보이고 있어 쉬운 구분이 가능하였다. 꿀벌의 gDNA와 SHB특이 DNA를 혼합하여 주형으로 사용하였을 경우, 2.09×102 이상의 SHB특이 분자가 존재하는 PCR에서는 비특이적 산물의 증폭이 측정되지 아니하였다(Fig. 2).
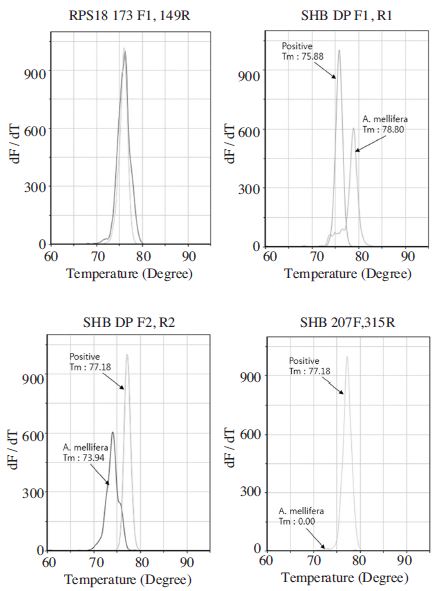
Melting temperature analysis of ultra-rapid PCR with genomic DNA from honeybee and/or SHB-specific DNA using various primer pairs. RPS genes were successfully amplified by both ultra-rapid PCRs with gDNA from honeybee and/or RPA-specific DNA. All ultra-rapid PCRs using SHB-DP F1/R1, -DP F2/R2, -207F/315R pairs with gDNA from honeybee and/or SHB-specific DNA produced correct or incorrect product, respectively.
꿀벌의 gDNA와 SHB 특이 유전자의 혼합물을 주형으로 한 초고속 PCR
꿀벌의 gDNA와 SHB특이 DNA가 혼합된 상태에서 각 primer 쌍의 SHB특이 검출의 한계를 측정하였다. SHB특이 염기서열을 탑재한 pTop-SHB COI 2.09×106 분자에서 2.09×100 분자까지 연속 희석한 후, 이들을 각기 100ng 꿀벌 gDNA와 정량 혼합하여 각기 초고속 PCR의 주형으로 사용하였다(Fig. 3; 1ng의 3kb DNA는 0.5 femtomole이며, 이는 약 3×108 molecules로 계산).
Primer 3쌍을 각기 사용한 SHB 특이 초고속 PCR들은 모두 100ng 꿀벌 gDNA 존재 하에도 2.09×101 분자까지 검출 가능함을 보여 주었으며, 특이적 증폭의 Tm은 SHB DP F1/R1 primer 쌍에서 75.82°C±0.21°C, SHB DP F2/R2 primer 쌍은 77.82°C±0.25°C, SHB 207F/315R primer 쌍은 77.82°C±0.17°C로 측정되었다.
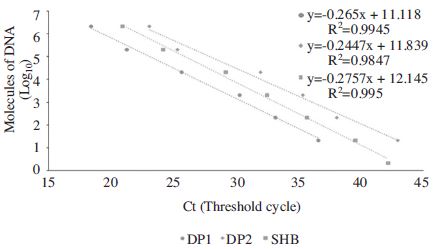
Detection limit of ultra-rapid PCR using three SHB-specific primer pairs with SHB-specific DNA and honeybee gDNA. Ultra-rapid PCR reactions were performed at optimal conditions for each primer pairs with 100ng honeybee gDNA. All ultra-rapid PCRs using SHB DP1, SHB DP2, SHB 207F/315R primer pairs could be detected with 2.09× 101 molecules of SHB-specific DNA under 100 ng honeybee gDNA.
SHB 특이 DNA 기질이 주어지지 않은 경우와, 극미량인 2.09×100 분자가 주어진 PCR에서는 비 특이 증폭산물이 발견되었으며, 이 경우, 각 primer 쌍에 의한 비특이 증폭산물의 Tm은 각기, 79.38°C±0.59°C(SHB DP F1/R1 primer 쌍), 79.53°C±1.38°C(SHB DP F2/R2 primer 쌍), 71.59°C±0.57°C(SHB 207F/315R primer 쌍)으로 측정되었으며, 이 Tm들은 해당 SHB-특이산물의 Tm들과 쉽게 구분되는 온도범위에 있었다.
이로써, 본 연구에 사용된 3쌍의 SHB 특이 primer쌍들은 모두 SHB-특이 초고속 PCR조건에서 SHB주형 2.09×101 분자이상이 주어지면 다른 염기서열의 존재에 방해됨이 없이 SHB 특이 정량 증폭 반응을 나타내는 것으로 판단되었으며, 아울러 비특이적 증폭산물을 쉽게 구분할 수 있었다(Table 6).
봉변-SHB 인공시료부터 SHB COI특이 유전자의 초고속 정량 검출
실제 봉변에서 SHB특이 유전자의 검출이 가능한지를 확인하고자, 꿀벌 gDNA 특이 primer들 2쌍(RPA, β-actin)과 SHB 특이 primer들 3쌍(SHB-DP F1/R1, -DP F2/R2, -207F/315R)을 각기 사용하는 초고속 PCR을시행하였다(Fig. 4).
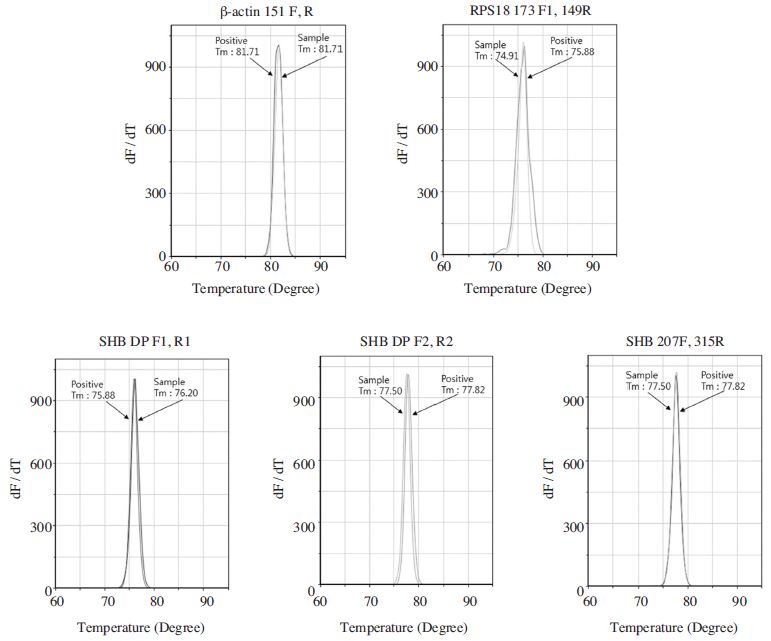
Melting temperature analysis of Ultra-rapid PCRs using 5 different primer pairs with total DNA from bee debris and SHB larvae. 3.59μg total DNA was isolated from 2 gram bee debris and 1 SHB larvae. Ultra-rapid PCRs using each primer pairs with 30ng total DNA were performed. All results demonstrated successful amplification according to primers-specificity.
β-actin 및 RPS 유전자는 봉변으로부터 gDNA의 추출이 정확히 일어났는지 확인하고자 증폭 반응을 실시하였으며, 두 개의 유전자에서 적정 범위의 Tm값을 형성하였으며, 전기영동에서도 동일한 크기의 DNA가 형성된 것을 확인하였으며(자료 미제시), 이는 봉변으로부터 꿀벌의 gDNA가 추출됨이 확인된 것이다.
SHB 특이 primer들 3쌍을 각기 사용하는 초고속 PCR에서 모두 그리고 각기 SHB 특이 표적유전자인 COI의 염기서열이 증폭되었으며, 정확한 SHB유전자가 증폭되었음은 해당 Tm의 분석으로 그리고 각기 전기영동에 의한 분석으로 확인할 수 있었다.
본 연구와 같이, 2g 봉변에 SHB 유충 한 마리가 존재하는 비율이라면, 여기에서 추출된 total DNA는 3.59μg이었으며, 각 초고속 유전자 증폭에 사용된 30ng total DNA의 양은 8/1,000마리의 SHB유충에서 추출된 SHB의 mtDNA가 포함된 것이며, 초고속 PCR은 이 분자들을 기질로 특이 증폭시켜 보여준 것이다.
과연 봉변 중 SHB 사체의 비율이 어느 정도가 될 때까지 SHB 특이 초고속 PCR에 의한 검출이 가능할 지 실험하였다. SHB 혼합 봉변에서 분리한 total DNA를 1/10씩 연속 희석하여 이 희석된 total DNA를 초기 기질로 SHB 특이 초고속 PCR을 시행하였다(Fig. 5).
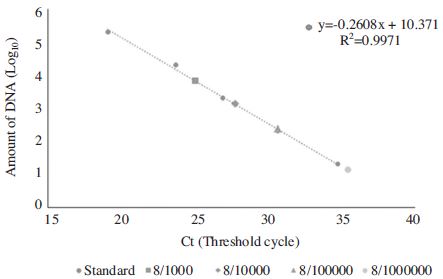
CTs of Ultra-rapid PCRs using serially diluted total DNA from bee debris and SHB larvae on regression equation using CTs and SHB-specific DNAs. Total DNA was isolated from 2 gram of bee debris and 1 SHB larvae. Serially diluted total DNA were measured as 0.008 (6.64×103 molecules), 0.0008 (1.37×103 molecules), 0.00008 (2.38×102 molecules), 0.000008 (1.31×101 molecules) SHB larvae, respectively. Standard is indicated PCRs using serially diluted SHB-specific DNA. 8/1000, 8/10000, 8/100000, 8/1000000 are indicated 0.008, 0.0008, 0.00008, 0.000008 SHB larvae, respectively.
초기 기질로 total DNA 30ng, 3ng, 0.3ng, 0.03ng을 사용하였으며, SHB-DP F1/R1, -DP F2/R2 primer 쌍을 각기 사용한 초고속 정량 PCR에서 모두 SHB특이 염기서열이 정량적으로 존재함을 확인하였다.
초기 기질로 사용된 total DNA 30ng, 3ng, 0.3ng, 0.03ng은 각기 8/1,000, 8/10,000, 8/100,000, 8/1,000,000마리의 SHB 유충에서 추출된 DNA가 포함된 것으로, 본 실험의 결과는 10만분의 1 마리의 SHB 유충 사체가 있다면, 그 존재를 특이적, 정량적으로 입증할 수 있다고 계산되었다. 이를 SHB 특이 DNA의 수로 정량화 하면, 0.008마리의 SHB 유충에서 SHB 특이 6.64×103 분자가 추출되었다는 것이며, 0.0008마리의 SHB 유충에서 SHB 특이 1.37×103 분자가, 0.00008마리의 SHB 유충에서 SHB 특이 분자가 2.38×102 분자가, 0.000008마리의 SHB 유충에서 SHB 특이 분자가 1.31×101 분자가각기추출되었던것으로계산되었다.
이는 SHB 유충의 수많은 다리 중 반의 반토막이라도 봉변에 낙하되어 수집된다면, 이는 이미 SHB의 존재를 입증하기에는 충분한 양의 시료라는 뜻이 될 것이다.
본 연구는 근래 국내 침입이 확인된 벌집꼬마밑빠진벌레 (SHB)의 신속하고 정확한 검출을 위하여 추진된 것이다.
육안에 의한 관찰만으로 봉군내 SHB 존재를 확인하기 위하여 봉군의 정밀한 내검은 필수적이나, 현실적으로 SHB의 확인이 가능한 것은“심한”SHB감염 봉군만으로 한정될 수 밖에 없으며, 더욱이 SHB가 존재하지 않음을 입증하는 것은 매우 어렵다할 것이다. 사실 육안에 의한 확인이 어려운 SHB의 알, 소수의 SHB 유충, 소수의 SHB 성충이 있는 봉군에서 이를 발견해내는 일은 내검 상황(훈연기의 연기, 방충망으로 가려진 시야, 봉군 내 많은 소비의 각 소비면을 면밀히 관찰, 엄폐한 SHB 성충들 등)을 감안한다면, 소수 SHB감염과 초기 감염의 경우 오판의 가능성을 더욱 크게 한다(Murihas, 2004; Spiewok and Neumann, 2006).
한편 벌통의 소문에서 간단히 받침판을 밀어 넣고, 이를 다시 회수함으로 쉽게 봉변을 채취할 수 있는 방법은 이미 고안되어 있다. 이 방법은 내검과는 달리 꿀벌들에게 스트레스를 주지 아니하며, 봉변이라는 봉군내 미소환경(micro-environment)의 종합적 특이 결과물들을 우리에게 제공해줄수있을것이다. 봉군에서 쉽게 채취될 수 있는 봉변을 보다 미세 분석하는 기법을 발전시킨다면 우리는 이 특수 미소환경의 결과물로부터 더 많은 유용한 정보를 얻을 수 있을 것으로 기대된다.
본 연구는 봉군내 SHB 존재 여부를 확인할 수 있는 방법으로, 봉변의 환경 DNA (eDNA; environmental DNA) 중 SHB 특이 DNA 검출법을 제안하는 것이며, 그간 발전된 초고속 PCR법의 신속성과 민감성은 봉변에서 추출된 total DNA 중 극소량을 가지고도 그 중 초극미량인 SHB 특이 DNA의 증폭이 가능함을 보여줄 수 있었다.
선행연구인Ward 등 (2007)의 PCR은 전형적 실험실적 PCR 검사로써, 우선 PCR의 조건을, 초기 변성 50°C 2 분 및 95°C 10분 후, 각 회전은 변성 95°C 15초, 혼성 및 중합과정 60°C 1분으로 하여 40회전에 이르게 하였다. 이는 총 PCR시간 단축을 위한 그 당시의 최신기법인 2단계/회전의 PCR법을 사용한 것이나, 각 작동온도(50°C, 95°C, 및 60°C)로 이동되는 시간(Ramping time)을 제외한, 작동온도에서 주어진 시간만 62분에 이르며, PCR에 사용한 7900 Sequence Detection System (Applied Biosystems, USA)의 온도변환 능력을 고려한다면 PCR의 작동시간은 능히 2시간을 초과할 것으로 계산된다.
한편, 본 개발에 사용된 초고속 PCR은, 초기 변성 95°C 30초 후, 변성 95°C 2초, 혼성 56°C 2초, 중합 72°C 2초를 1회전으로 하여 총 50회전으로 Genechecker™(Genesystem, Korea)를 사용하였다. 이 초고속 PCR에 소요된 총 시간은 18분 40초였으며, 대부분의 시료들은 40회전(15분) 이전에 증폭여부와 Ct값을 측정할 수 있었다(Table 6).
이런 검사시간의 단축은 현장실험의 가능성을 크게 높여주는 것으로, SHB존재여부의 판정을 양봉현장에서도 바로 시행할 수 있을 것이다
초고속 PCR의 또 다른 실용적 장점은 40 cycles 부근의 Ct값이 정확한 정량성을 보인다는 것이다(Table 6). 이는 PCR 중 중합효소(polymerase)가 변성 온도(95°C)에 노출되는 시간의 합이 상대적으로 매우 짧은 것에 기인하는 것으로 판단되며, 40 cycles 이상에서도 PCR의 증폭반등이 실제로 작동됨을 보여준다. 본 연구의 초고속 PCR 조건에서 변성온도에 노출되는 시간의 합은 단 100초(2초×50회전)이었으나, Ward 등(2007)의 조건에서는 40회전일 때 이미 10분(600초)에 달하게 되고, 이 이유로 초고속 PCR의 경우, 20개 수준의 표적 분자들 만이라도 정량적으로 증폭시킬 수 있음을 보여주었다(Table 6).
작년 9월 국내에 SHB의 침입이 확인된 이래, 이제 SHB는 전국의 봉군으로 서서히 또는 빠르게 전파될 것으로 예상되고 있다. 그러나 국내 양봉이 미국의 경우와는 다르게, 높은 관리양봉의 수준에 있음을 감안한다면, 우리의 노력으로 이 SHB의 박멸은 어렵지만 가능할 수도 있다는 점을 강조하고자 한다.
본 연구에서 제안된 벌집꼬마밑빠진벌레 특이 초고속 PCR법은 실험실에서 그리고 양봉현장에서도 적용이 가능하며, 나름 우수한 검출능력을 보여주고 있다. 지역의 모든 봉군을 대상으로 하는 벌집꼬마밑빠진벌레의 광역 전수 조사나 1개 봉장의 개별 봉군 조사에 모두 유용할 것이며, 본 연구에서 제안하는 봉변으로부터 SHB 유전자의 초고속 검출법이 벌집꼬마밑빠진벌레 검출 및 제어에 널리 사용되길 기대한다.
적 요
벌집꼬마밑빠진벌레 (Small hive beetle; SHB; Aethina tumida)의 신속검출과 대량 조사를 위하여 SHB 특이 초고속 유전자 증폭법을 개발하였다. 3쌍의 Aethina tumida-특이 유전자 증폭용 프라이머들은 벌집꼬마밑빠진벌레의 미토콘드리아 유전체 중 cytochrome oxidase subunit I (COI) 유전자에 근거하여 선발하였다. 최적화된 초고속 PCR은 2.1×101 분자의 작은벌집딱정벌레 COI 유전자를 18분 40초만에 특이적으로 그리고 정량적으로 검출할 수 있었다. 양봉현장의 적용을 위하여, 봉변으로부터 쉽게 DNA를 추출하는 방법을 고안하였으며, 봉변 1g 중 105 분자의 벌집꼬마밑빠진벌레 COI 유전자가 존재할 경우(1/1000의 SHB 유충 사체), 10분 이내에 벌집꼬마밑빠진벌레의 존재와 분자적 정량을 마칠 수 있었다. 제안하는 이 실험법이 양봉현장에 널리 적용되어, 벌통 내 벌집꼬마밑빠진벌레의 침입여부 판단, 증식의 수준, 그리고 침입 지역의 파악 및 제어에 활용되기를 기대한다.
Acknowledgments
본 연구는, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(과제번호 115058-02, 과제번호 115102-03), 농생명산업기술개발사업(312027-03) 및 수출전략기술개발사업(과제번호 115067-02), 그리고 2017학년도 경기대학교 대학원 연구원장학생 장학금 지원에 의하여 수행되었음.
References
- 윤병수, (2017), 꿀벌 질병 방역관리 방안 낭충봉아부패병 및 SHB감염증에 대한 대책, 농림축산식품부(보고서), 등록번호 11-1543000-001739-01. 20-21.
- 정년기, (2013), 학술 4 - 꿀벌에 해로운 동물들 - 1, Journal of the Korean Veterinary Medical Association, 49(2), p116-122.
-
Benda, N. D., D. Boucias, B. Torto, and P. Teal, (2008), Detection and characterization of Kodamaea ohmeri associated with small hive beetle Aethina tumida infesting honey bee hive, J. Apic. Res, 47(3), p194-201.
[https://doi.org/10.1080/00218839.2008.11101459]

- Evans, J. D., J. S. Pettis, and H. Shimanuki, (2000), Mitochondrial DNA Relationships in an Emergent Pest of Honey Bees: Aethina tumid (Coleopters: Nitidulidae) From the United States and Africa, Entomological Society of America, Vol. 93(3).
- Evans, J. D., J. S. Pettis, W. M. Hood, and H. Shimanuki, (2002), Tracking an invasive honey bee pest: mitochondrial DNA variation in North American small hive beetles, Apidologie, 34, p103-109.
- Eyer, M., Y. P. Chen, M. O. Schafer, J. Petter, and P. Neumann, (2008), Small hive beetle, Aethina tumida, as a potential biological vector of honeybee viruses, Apidologie, 40, p419-428.
-
Han, S. H., D. B. Lee, D. W. Lee, E. H. Kim, and B. S. Yoon, (2008), Ultra-rapid real-time PCR for the detection of Paenibacillus larvae, the causative agent of American Foulbrood (AFB), J. Invertebr. Pathol, 99, p8-13.
[https://doi.org/10.1016/j.jip.2008.04.010]

- Lee, D. W., E. H. Kim, M. S. Yoo, S. H. Han, and B. S. Yoon, (2007), Ultra-rapid real-time PCR for the detection of human immunodeficiency virus (HIV), Kor. J. Microbiol, 43, p91-99.
- Lee, S., K. J. Hong, Y. S. Cho, Y. S. Choi, M. S. Yoo, and S. H. Lee, (2017), Review of the subgenus Aethina Erichson s. str. (Coleoptera: Nitidulidae: Nitidulinae) in Korea, reporting recent invasion of small hive beetle, Aethina tumida, Journal of Asia-Pacific Entomology, 20(2), p553-558.
- Murihas, A. M., (2004), Aethina tumida arrives in Portugal. Will it be eradicated?, Eurbee Newsletter, 2, p7-9.
-
Spiewok, S., P. Neumann, (2006), Cryptic low-level reproduction of small hive beetles in honeybee colonies, J. Apic. Res, 45, p47-48.
[https://doi.org/10.1080/00218839.2006.11101313]

- Thomas, M. C., (1998), Florida pest alert - the small hive beetle, American bee Journal, 138(8), p565.
-
Ward, L., M. Brown, P. Neumann, S. Wilkins, J. Pettis, and N. Boonham, (2007), A DNA method for screening hive debris for the presence of small hive beetle (Aethina tumida), Apidologie, 38, p1-9.
[https://doi.org/10.1051/apido:2007004]

-
Yoo, M. S., K. C. N. Tai, P. V. Nguyen, S. H. Han, S. W. Kwon, and B. S. Yoon, (2012), Rapid detection of Sacbrood virus in honeybee using ultra-rapid real-time polymerase chain reaction, J. Virol. Methods.
[https://doi.org/10.1016/j.jviromet.2011.10.014]

- Yang, X., and Cox-Foster, D. L., (2005), Impact of an ectoparasite on the immunity and pathology of an invertebrate: evidence for host immunosuppression and viral amplification, PNAS, 21, p7470-7475.
