Slow Bee Paralysis Virus (SBPV) 신속 검출을 위한 초고속 역전사 중합효소 연쇄반응법의 개발
Abstract
Slow Bee Paralysis Virus (SBPV) is a pathogenic virus against honeybee and bumblebee, causes the death of adult bee by paralyzing the fore-leg of bee. In this study, for rapid detection of SBPV from bumblebee, SBPV-specific Ultra-rapid Reverse transcription PCR was developed. After optimizing of SBPV-specific Ultra-rapid PCR, the existence of 1.0×108 SBPV-specific DNA molecules could be recognized in 3 minute and 35 seconds. Even 1.0×101 molecules of SBPVspecific DNA could be measured with quantitative manner. Meanwhile, from both imported bumblebee and bumblebee produced in Korea, SBPV were detected using proposed method. In the laboratory as well as in the field, SBPV-specific Ultra-rapid Reverse transcription PCR would be applied and might be expected as useful tools at production of bumblebee or inspection for the import and export system of bumblebee.
Keywords:
Slow Bee Paralysis Virus, SBPV, Bumblebee, Bombus terrestris, Ultra-rapid PCR, Rapid detection서 론
서양뒤영벌(Bombus terrestris)에 대한 병원성 바이러스는 모두 서양종 꿀벌(Apis mellifera)에 대한 병원체로 처음 알려진 것들이며, 국내에서도 Black queen cell virus (BQCV), Chronic bee paralysis virus (CBPV), Cloudy wing virus (CWV), Deformed wing virus (DWV), Israel acute paralysis virus (IAPV), Kashmir bee virus (KBV), Sacbrood virus (SBV)의 7종이 서양뒤영벌에서 검출된 바 있다(최 등, 2009; 최 등, 2010). 또한, A. mellifera와 Bombus species에서 개별적으로 Acute bee paralysis virus (ABPV), BQCV, DWV, Slow bee paralysis virus (SBPV)들이 병원체로 검출된 바 있으며, 특히 Bombus species 중 일부에서 SBPV의 발병률이 특히 높음이 보고된 바 있었다(Dino 등, 2015). 이들은 잠복기간이 길며, 효과적으로 알려진 예방 및 방제법이 없으며, 수시로 다른 감염체와 혼합감염 등으로 봉군에 위해를 가하는 것으로 알려져 왔다.
이들 바이러스 중 SBPV는 다른 꿀벌바이러스와 같이 Iflaviridae family에 속하며, 약 9.5kb정도의 positivestrand RNA virus이다. SBPV는 영국에서 1974년에 벌바이러스 X와 관련된 연구 중 우연히 발견되었으며, SBPV에 감염된 성체는 사멸하기 하루 전후로 앞의 두 다리가 마비된다고 하여 SBPV라 명명되었다(Bailey et al., 1974).
SBPV 검출에 대한 연구는 뒤영벌의 다양한 병원체들에 대한 실시간 다중 PCR법(민 등, 2017)에서 처음 제시된 바 있었으나, 국내에서 SBPV에 감염된 뒤영벌을 발견하지 못하였고, SBPV의 검출을 위한 보다 신속하며, 현장 적용성이 가능한 방법은 개발된 바가 없었다. 국내의 뒤영벌에서 이들의 감염 현황을 파악하는 것은, SBPV 검출의 보고도 없었기에, 유관된 자료조차 찾기 어려웠다.
한편, 초고속 PCR (Ultra-Rapid PCR)은 특이 유전자를 10분 이내 증폭해 낼 수 있는 진보된 PCR기법을 말하며(Han et al., 2008), 석고병의 원인균인 Ascosphaera apis와 백묵병의 원인균인 Aspergillus flavus의 특이적 검출을 가능하게 하였고(왕 등, 2016), 또 역전사 반응을 최소화한 korean Sacbrood Virus의 초고속 RTPCR이 개발되었다(민 등, 2016). 이는 근래에 더 발전되어 봉변으로부터 벌집 침입 해충인 SHB (Small Hive Beetle)의 특이 유전자 검출, 각 꿀벌 병원체들의 초신속 검출 등에 적용되어, 수 분내 목적한 특이유전자를 증폭하며, 이의 존재와 함께 정량이 가능함을 보여주었다(김 등, 2017; 임 등, 2017).
따라서 본 연구는 서양뒤영벌의 병원체로 SBPV의 보다 빠른 검출과 이 검출법의 보다 간편한 현장 활용을 위하여, SBPV 특이 초고속 PCR법을 새로이 개발하고자 하였으며, 이 SBPV 특이 초고속 PCR법을 이용하여 국내 뒤영벌에서 SBPV를 검출하고 현황을 파악하는데 기여하고자 하였다.
재료 및 방법
뒤영벌 시료 확보 및 total RNA의 추출
뒤영벌 시료는 2016년 8월 경기도 수원, 전라남도 보성, 경상북도 청송, 벨기에에서 구입된 서양뒤영벌(Bombus terrestris)을 사용하였다.
Total RNA 분리는 RNAiso Plus Kit (Takara, Japan)를 사용하여 제작자의 지시에 따라 수행하였다. 약술하면, 날개를 제거한 뒤영벌 시료 1마리를 2.0ml tube에 넣고 glass bead (pore size : 1mm, Germany)와 혼합한 후, 1.0ml RNAiso Plus 시약을 넣고 MagNa Lyser (Roche, Switzerland)로 분쇄하였다. 분쇄된 시료에 200μl chloroform을 넣고 15초간 vortexing하여 혼합하였으며, 4°C에서 13,000rpm 10분간 원심분리하여, 상등액만을 1.5ml tube에 옮겼다. 시료상등액과 같은 부피의 isopropyl alcohol을 넣고 15초간 vortexing하여 혼합하였으며, 4°C에서 13,000rpm, 10분간 원심분리하고 상등액을 제거하였다. RNA 침전물은 75% ethanol을 사용하여 washing 과정을 진행한 후, DEPC 처리된 증류수를 사용하여 total RNA의 용액으로 확보하였다. 추출된 total RNA는 Biophotometer (Eppendorf Co., Germany)를 사용하여 OD260 (Optical Density 260nm)으로 정량하였고 -70°C에 보관하며 이후 실험에 사용하였다.
SBPV 특이 유전자의 재조합 DNA 제작 및 Primer 선별
SBPV 특이 유전자 재조합 DNA (pSBPV-329)는 경기대학교 생명과학과 분자생물학 연구실에 확보된 것을 사용하였으며, 여러 SBPV primer 쌍 중 Ultrarapid PCR에 최적인 primer를 선별하여 사용하였다. 유전자 정보 및 참고문헌은 Table 1과 Supplement Table S1에 나타내었다.
SBPV 특이 유전자 초고속 검출을 위한 PCR 조건 최적화
초고속 PCR의 최적화를 위하여, 주형으로 재조합 DNA pSBPV-329를 사용하였으며, OD260으로 정량한 후, 1.0×105 분자를 기준으로 초고속 PCR에 사용하였다. 초고속 PCR을 위한 Thermocycler는 GENECHECKER(Gene System Co., Korea)를 사용하였으며, 초고속 PCR의 온도 및 시간 조건은 95°C 초기 변성 15초 후, 95°C 변성 3초, 혼성 3초, 72°C 중합 3초를 기준으로 하였고, 총 50 회전을 진행하였다. 최적 annealing 온도는 50~65°C 범위에서 구하였으며, 최적의 primer 농도는 10μM, 8μM, 4μM, 2μM, 1μM, 0.5μM, 0.25μM에서 각기CT (Threshold Cycles)값과 최대 형광값의 측정으로 조정하였다.
SBPV 특이 유전자의 최소 검출 시간에 따른 검출 한계 측정
SBPV 특이 초고속 PCR의 최소 검출 시간에서 특이 검출의 한계를 측정하기 위하여 초고속 PCR의 각 회전 중 혼성, 중합의 시간을 각각 3초에서 1초까지 감소시켜 각 온도와 시간 조건에 따른 CT 값과 CT 값이 측정되는 시간(CT time)을 측정하였다. 이 측정에서는 재조합 DNA인 pSBPV-329를 1.0×108 분자부터 1.0×100 분자까지 연속 희석하여, 초고속 PCR의 주형으로 사용하였으며, 초고속 PCR의 각 단계에서 시간 및 온도 조건도 가감하여, SBPV 특이 초고속 PCR이 최소 검출 시간 내에 검출 가능한 최소 한계를 추구하였다.
뒤영벌 gDNA 존재하에 SBPV 특이 초고속 PCR의 검출한계 및 정량 범위
뒤영벌 gDNA 존재하에 SBPV 검출한계를 측정하기 위하여, SBPV 에서 추출한 핵산을 주형으로 SBPV 특이 유전자의 증폭량을 확인하였다. PCR 반응은 5μl의 2 × Rapi master mix와 1μl씩의 SBPV F/SBPV-C3G peptidase R (최종 농도 2μM) primer에 뒤영벌 gDNA(100ng)를 첨가하여 총 10μl로 조성하여 수행하였으며, 초고속 PCR의 각 단계에서 시간 및 온도 조건은 앞선 실험의 최적화한 조건에 근거하여, 95°C 초기 변성 15초 후, 95°C 변성 1초, 59°C 혼성 2초, 72°C 중합 1초를 표준으로 하였고, 총 50 회전을 진행하였다.
국내 뒤영벌에서 SBPV 특이 초고속 RT-PCR을 적용한 검출
국내에 판매되고 있는 서양뒤영벌의 SBPV 감염여부를 판단하기 위하여, 경기도 수원, 전라남도 보성, 경상북도 청송, 벨기에에서 생산된 뒤영벌 각 1마리에 대하여 Total RNA를 분리하였다. Total RNA는 각 100ng을 사용하여 뒤영벌에 대하여 SBPV 특이 초고속 역전사 PCR을 수행하였다. PCR 반응은 5μl의 2×Rapi RT master mix와 1μl씩의 SBPV F/SBPV-C3G peptidase R (최종 농도 2μM) primer로 총 10μl로 조성하여 수행하였다. SBPV 특이 초고속 역전사 PCR은 GENECHECKER (Gene System Co., Korea)를 사용하였으며, 최적화된 PCR의 온도 및 시간 조건은 50°C, 1분 역전사 반응 후, 95°C, 15초 초기 변성, 95°C 1초 변성, 59°C, 2초 혼성, 72°C, 1초 중합을 1회전으로 하여 총 50 회전을 진행하였다.
결과 및 고찰
SBPV 특이 유전자 초고속 검출을 위한 PCR 조건 최적화
SBPV 특이 초고속 PCR에서 최적의 혼성 온도를 파악하고자 재조합 DNA 1.0×105 분자의 pSBPV-329를 사용하였으며, 초고속 PCR의 온도 및 시간 조건은 95°C 초기 변성 15초 후, 95°C 변성 3초, 50~65°C 혼성 3초, 72°C 중합 3초를 표준으로 총 50 회전 진행하였다.
SBPV 특이 유전자 초고속 검출가능의 온도는 53~65°C의 범위이며, 50°C의 경우, 정량적 검출이 어려워 최적조건 정립에서 제외하였다. 가장 빠른 CT는 혼성 온도 59°C일 때 23.14 회전으로 측정되었으며, 이에 이르기까지 소요된 PCR 진행 시간(CT time)은 8분 22초이었다. 따라서 SBPV 특이 초고속 PCR에서 최적 혼성 온도는 최저 CT 값 및 CT time, 그리고 최종 형광값(증폭량, F=262) 모두에서 우수한 결과를 보인 59°C로 결정하였다(자료 미제시).
또한, 초고속 PCR에서 SBPV 특이 primer의 최적 농도를 구하기 위하여 재조합 DNA 1.0×105 분자의 pSBPV-329를 사용하였으며, 각 10μM, 8μM, 4μM, 2μM, 1μM, 0.5μM, 0.25μM(각 최종농도)의 농도의 primer를 각각 사용하여 실험하였다. 특이 primer의 최적 농도는 가장 빠른 CT 값과 최종 형광값을 기준으로 판단하였다.
고농도, 즉 10μM와 8μM의 primer를 사용한 초고속 PCR은 각각 7.96, 8.39 회전의 빠른 CT 값을 보였고, 최종 형광값 또한 우수한 수준으로 측정되었다. 이고농도 primer의 초고속 PCR은 4μM 또는 2μM의 중농도 primer를 사용한 초고속 PCR보다 12 회전이상 빠른 CT 값을 보인 것으로, 이는 1.0×101 분자 이내의 극미량 초기 주형에 대한 증폭에서 보다 유리할 것으로 판단되었다. 그러나, 이 고농도 primer는 초고속 PCR의 안정성에서 불안정한 면을 보였으며, 1.0×105 분자 이상의 초기 주형을 사용하였을 경우 정량성이 현저히 떨어지는 것으로 나타났다(자료 미제시).
이런 이유 등으로 SBPV 특이 초고속 PCR에서 최적 primer의 농도는 CT (22.08 회전)와 CT time (7분 59초)에서 빠른 검출시간을 보였으며, 최종 형광값(262) 모두에서 우수한 결과를 보인 2μM을 최적 primer 농도로 결정하였다(Fig. 1).
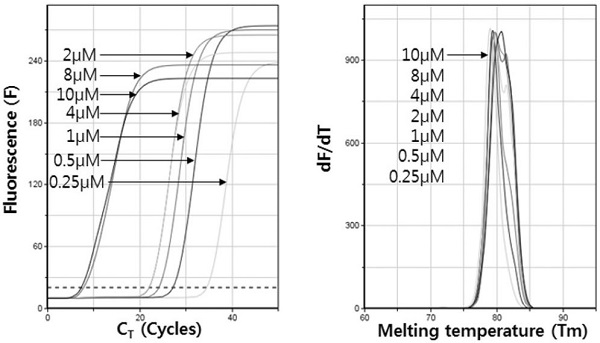
Optimization of primer concentration using SBPV-specific Ultra-rapid PCR. Using various concentrations of primers, optimal concentration of SBPV-specific primer were selected for Ultra-rapid PCR. At the final concentrations of 10μM and 8μM of primer, Ultra-rapid PCR showed the fastest CT values of 7.96 and 8.39 cycles and the final fluorescence values were also measured to be excellent. However, when the concentration of the primer was set to 10μM and 8μM, the CT values were unstable and no exact quantitative manner using higher numbers than 1.0×105 SBPV-specific molecules, as initial template. Based on these reasons, optimal concentration of specific primer was selected as 2μM each (CT=22.08, CT time=7min 59sec, F=262).
SBPV 특이 유전자의 최소 검출 시간에 따른 검출 한계 측정
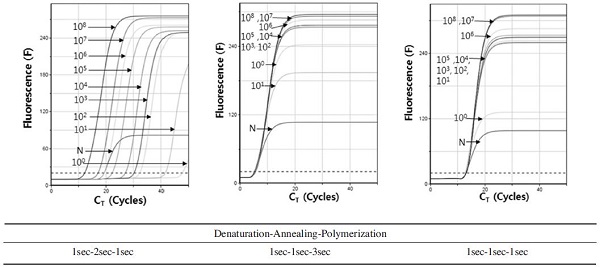
PCR의 각 단계 시간 단축은 초고속 검출에 있어서 전체 검사 시간을 줄일 수 있는 가장 중요한 요소로써 작용한다. SBPV 유전자의 최단 검출 시간을 위해, PCR 각 단계(혼성, 중합)의 시간을 3초에서 1초까지 조절하여 초고속 PCR을 시행하였고 변성시간은 1초로 고정하여 초고속PCR을 수행하였다.
1.0×103 분자인 pSBPV-329를 기질로 사용한 초고속 PCR에서 각 회전의 혼성시간을 3초에서 1초까지 조정한 결과, 각CT 값은 3초에서 28.92 회전(CT time=9분 37초), 2초에서 29.89 회전(CT time= 9분 30초)으로 측정되었으며 SBPV 특이 유전자의 증폭을 확인할 수 있었다. 혼성시간이 1초일 경우, DNA 증폭을 나타내는 형광곡선이 1.0×108 분자부터 1.0×100 분자까지 동일한 CT 값인 6 회전에서 동시에 증폭됨을 볼 수 있어 정량적 측정이 불완전하다고 판단하였다. 같은 조건에서 각 혼성시간만 3초와 2초로 감소시킨 초고속 PCR을 비교한 결과, 해당 CT 값이 혼성시간이 짧아질수록 증가하는 결과를 보여주었으며, CT time은 혼성시간 2초에서 9분 30초로 혼성시간 3초와 비교했을 때, 7초 차이로 감소세를 보였다. 따라서 혼성시간을 3초에서 1초로 조정하여 비교하였을 때의 검출 가능 최소 혼성시간은 2초로 판단되었다.
1.0×103 분자인 pSBPV-329를 기질로 사용한 초고속 PCR에서 각 회전의 중합시간을 3초에서 1초까지 조정한 결과, 각 CT 값은 3초에서 29.89 회전(CT time= 9분 30초), 2초에서 29.97 회전(CT time= 9분 1초), 1초에서 30.02 회전(CT time= 8분 31초)으로 측정되었으며 SBPV 특이 유전자의 증폭을 확인할 수 있었다. 같은 조건에서 각 중합시간만 3초, 2초, 1초로 감소시켰을 때, 해당 CT 값은 중합시간이 짧아질수록 증가하는 결과를 보여주었으나, CT time에서는 점차 감소하는 양상을 보였다. CT time은 중합시간 1초에서 8분 31초로 중합 시간 3초와 비교했을 때, 59초 차이로 큰 감소세를 보였다. 따라서 중합시간을 3초에서 1초로 조정하여 비교하였을 때의 검출 가능 최소 중합시간은 1초로 판단되었다(Fig. 2).
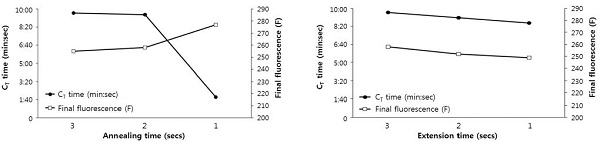
Minimum time of annealing and/or polymerization in SBPV-specific Ultra-rapid PCR. The annealing time and polymerization time were shortened to set the minimum detection time in SBPV-specific Ultra-rapid PCR. Denaturation step was fixed at 1 sec in each cycle of PCR and the times of annealing step and/or polymerization step were shortened from 3 sec to 1 sec.
한편, 변성, 혼성, 중합시간을 각 1초, 2초, 1초로 단축한 결과, DNA 증폭을 나타내는 형광곡선은 초기주형량에 따라 정상적인 증가를 보여주었으며 1.0×101 분자까지 SBPV 특이 유전자의 증폭을 확인할 수 있었고 1.0×102 분자까지 정량적으로 검출됨을 확인하였다. 또한 혼성시간을 1초 조정한 1초, 1초, 3초 조건으로 하였을 때, DNA 증폭을 나타내는 형광곡선이 1.0×108 분자부터 1.0×100 분자까지 동일한 CT 값에서 동시에 증폭됨을 볼 수 있어 정량적 측정이 불가능하다고 판단하였다(Supplement Fig. S1).
따라서 SBPV 특이 초고속 PCR은 PCR 시스템의 안정성 또한 고려하여 각 회전의 변성, 혼합, 중합 시간을 각각1초, 2초, 1초로 수행하였다.
또한, SBPV 특이 초고속 PCR에서 SBPV 유전자의 특이 증폭을 위한 최소 검출 시간을 측정하였다. 1.0×108 pSBPV-329 분자를 주형으로 사용하였으며, 최적화된 SBPV 특이 초고속 PCR의 조성으로, 최적화 된 온도조건하에 각 변성, 혼성, 중합 시간을 각 1초, 2초, 1초로 설정하고 50 회전을 수행하였다. 초기 변성 15초 후, 12.60 회전만에 CT line에 도달하였으며, CT time은 3분 35초였다. 37 회전(CT time은 10분 31초) 전후에 최대 형광값을 보여주었으며, 50 회전의 초고속 PCR은 15분 45초에 종료되었다(Supplement Table S2).
SBPV 특이 초고속 PCR의 검출한계 및 정량 범위
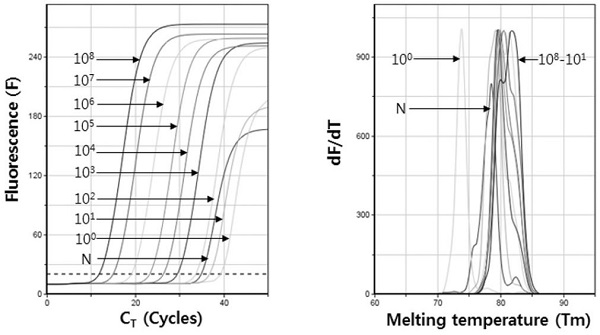
SBPV 특이 초고속 PCR의 안정적 시간 조건(각 회전 중 변성, 혼성, 중합시간 각 1초, 2초, 1초)에서 SBPV 유전자의 최소 검출한계를 확인하였다. SBPV 특이 유전자 재조합 DNA 1.0×108 분자를 연속 희석하여, 1.0×100 분자까지 초고속 PCR의 주형으로 사용하여 각 초기주형수에 따른 검출한계를 측정하였다(Fig. 3).
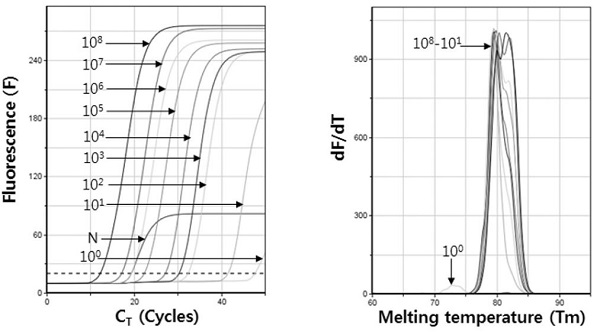
Detection limit of SBPV-specific Ultra-rapid real-time PCR with 10-fold diluted specific DNAs. The detection limit of SBPV gene was estimated using SBPV specific Ultra-rapid PCR under the minimum times of thermocycling (1 sec of denaturation, 2 sec of annealing, 1 sec of polymerization in single cycles). Until 1.0×101 molecules of pSBPV-329, specific products were amplified, respectively. Tms of all PCR products were measured as 80.45±1.15°C, except 1.0 ×100 molecule and PCR product using non template.
상기 조건에서 SBPV 유전자는 1.0×101 분자의 초기 주형에서도 성공적으로 증폭이 가능함을 보여 주었다. 10배씩 감소시킨 초기분자량 간의CT 값의 차이를 dCT10 라 정의하며, 1.0×108 분자에서 1.0×102 분자까지의 평균 dCT 값은 약 3.20 회전으로 계산되었다. 1.0×101 분자의 초기 주형에서 평균CT 값은 40.74회전이었고, 평균 CT time은 11분 45초이었고, Tm 값 또한 79.63°C로 SBPV의 유효범위에 포함되었다. 따라서 안정적인 검출에 필요한 주형의 양은 1.0×101 분자 이상이라 판단하였다.
SBPV 특이 유전자 대신 증류수를 넣어준 SBPV 특이 초고속 PCR (N; non-template, Fig. 4)은 19 회전 부근에서 CT line을 넘는 증폭이 관찰되었으나, 이 Tm 값은 측정되지도 않는 낮은 수준의 산물을 생성한 것으로 보이며, 이는 특이적 증폭이 아닌 것으로 판단할 수 있었다. 또한 1.0×100 분자의 증폭에서도 46 회전부근에서 CT line을 넘는 증폭이 관찰되었으나 이 또한 Tm 값이 그래프상에서 측정되지도 않아 특이적 증폭이 아닌 것으로 판단하였다.
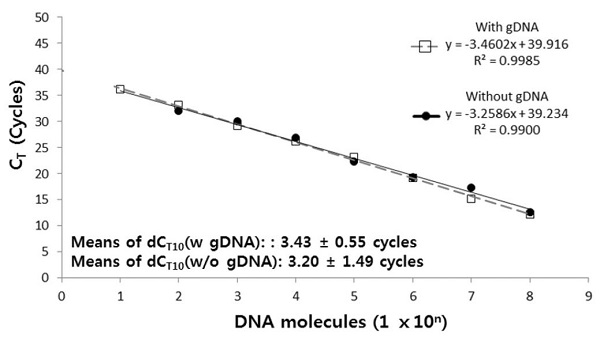
Regression equation based on SBPV molecules and CT using SBPV-specific Ultra-rapid PCR. dCT indicates difference between each CT values (cycles) of PCRs using 10-fold diluted initial templates. Means of dCT10 is 3.20±1.49 cycles. Quantitative measurement was possible from 1.0×108 molecules to 1.0×102 molecules. At the detection limit in the presence of bumblebee gDNA, means of dCT10 is 3.43 ±0.55 cycles. Quantitative measurement was possible from 1.0×108 molecules to 1.0×101 molecules.
한편, 재조합 DNA인 pSBPV-329를 사용한 초고속 PCR에서 연속 희석된 주형 DNA의 양에 따라 각 CT값이 측정되었으며, 이에 따른 회귀식(Regression equation)은 y=-3.2586x + 39.234 (y=초기 SBPV의 분자수)로, 이 때 회귀 상수(Regression coefficient)는 R2=0.9900이었다.
또한, 정확한 정량을 위한 정량 가능 범위는 주형 DNA가 1.0×102 분자 이상 1.0×108 분자까지라 판단하였으며, 이를 정량가능범위로 정하였다(Fig. 4).
뒤영벌 gDNA 존재하에 SBPV 특이 초고속 PCR의 검출한계 및 정량 범위
뒤영벌 gDNA 존재하에 SBPV 특이 초고속 PCR의 안정적 시간 조건(각 회전 중 변성, 혼성, 중합시간 각 1초, 2초, 1초)에서 SBPV 유전자의 최소 검출한계를 확인하였다. 100ng의 뒤영벌 gDNA와 함께 SBPV 특이 유전자 재조합 DNA 1.0×108를 연속 희석하여, 1.0×100 분자까지 초고속 PCR의 주형으로 사용하여 검출한계를 측정하였다(Fig. 5).
Detection limit of SBPV-specific Ultra-rapid real-time PCR with 10-fold serial diluted specific DNAs with gDNA of bumblebee. The detection limit of SBPV specific genes with gDNA of bumblebee was estimated using SBPV specific Ultra-rapid real-time PCR under the minimum times of thermocycling (1 sec of denaturation, 2 sec of annealing, 1 sec of polymerization in each cycle). As initial template, until 1.0×101 molecules of pSBPV-329 was also amplified. Tms of all PCR products were measured as 80.47±1.32°C, except PCR products from 1.0×100 molecule and non template.
100ng 뒤영벌 gDNA의 존재하에서도 SBPV 특이 유전자는, 1.0×101 분자의 초기 주형까지, 성공적으로 증폭되었다. 초기주형의 분자수를 10배 희석시킨 PCR의 CT 값 차이인, dCT10을 각기 계산하였으며, 1.0×108 초기 주형 분자에서 1.0×101 분자까지 계산된 각 dCT10의 평균 dCT10 값은 약 3.43 회전으로 계산되었다. 이는 뒤영벌 gDNA의 첨가없이 수행된 같은 초고속 PCR결과(Fig. 3 및 Fig. 4)와 매우 유사한 값으로, SBPV-특이 초고속 PCR은 초기 주형에 100ng 뒤영벌 gDNA를 사용한 것과 정량 및 정성의 면에서 무관하며, 간섭이 없었음을 보여주었다.
증류수 만을 초기 주형 대신 사용한 SBPV 특이 초고속 PCR은 34 회전 부근에서 CT line을 넘는 증폭이 관찰되었으나(N; non-template, Fig. 5), 융점 분석을 통해 SBPV의 유효범위인 80.47°C와 큰 차이를 보이는 78.49°C로 측정되어 특이적 증폭이 아님을 쉽게 확인할 수 있었다. 1.0×100 분자의 SBPV 특이주형에서도 증폭함을 확인하였으나 융점 분석을 통해 SBPV의 유효범위인 80.47°C와 큰차이를 보이는 73.87°C로 측정되어 특이적 증폭이 아님을 쉽게 확인할 수 있었다.
뒤영벌 gDNA 100ng의 존재하에, 연속 희석된 재조합 DNA pSBPV-329를 사용한 SBPV 특이 초고속 PCR에서 각 CT 값이 측정되었으며, 이에 따른 회귀식(Regression equation)은 y=-3.4602x+39.916 (y=초기 SBPV의 분자수)로, 회귀 상수(Regression coefficient)는 R2=0.9985이었다(Fig. 4).
SBPV 감염 의심 뒤영벌 사체에서의 SBPV 특이 초고속 RT-PCR을 통한 검출
SBPV 특이 초고속 PCR의 현장 적용성을 검증하기 위하여, SBPV 감염이 추정되는 국내외 생산 뒤영벌 시료들로부터 total RNA를 분리하였으며, 해당 뒤영벌 한 마리로부터 회수한 total RNA 100ng을 사용하여 SBPV 특이 초고속 RT-PCR을 수행하였다. 결과로써, SBPV 감염이 추정되던 시료들 중 일부, 벨기에 수입 뒤영벌, 그리고 국내 생산 1개 시료로부터 SBPV 존재에 대한 양성반응을 판정할 수 있었다(Fig. 6).
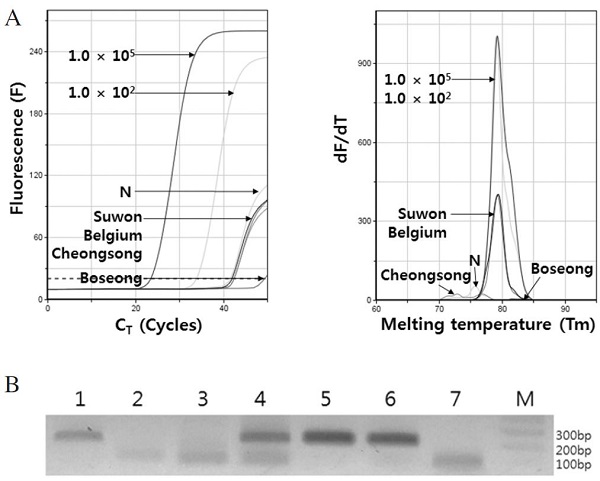
SBPV specific UR-RT PCR with various bumblebee samples. UR-PCR was performed with 100ng total RNA from each sample as inital templates. The Reverse transcription condition was 50°C, 1 min and the PCR condtion was 95°C, 15 sec of pre-denaturation, 95°C, 1 sec of denaturation, 59°C, 2 sec of annealing, 72°C, 1 sec of polymerization in each cycle for 50 cycles. SBPV specific gene was amplified from the samples of Belgium and Suwon. Panel A is the fluorescent graph of amplification and the peak graph of Tm of each amplicon. Panel B is amplified DNAs on the agorose gel. Lane 1 is sample from Suwon. Lane 2 is from Boseong. Lane 3 is Cheongsong. Lane 4 is Belgium sample. Lane 5, 6 are 1.0×105 molecules and 1.0 ×102 molecules of pSBPV-329, respectively. Lane 7 is nontemplate.
SBPV 특이 초고속 PCR 결과, 수원(Suwon)과 벨기에(Belgium)에서 생산된 뒤영벌 시료에서 SBPV 특이 유전자 증폭이 관찰되었으며, 각 산물의 융점분석에서 SBPV 특이 유전자의 증폭으로 판단되었다. 측정된 SBPV의 Tm 값은 수원 79.48°C, 벨기에 79.15로, SBPV의 양성 증폭의 유효범위(80.47±1.32°C) 안에 해당함으로 SBPV 유전자의 증폭으로 판정한 것이다(Table 2).
뒤영벌 사체에 포함되어 있는 SBPV 양은 Fig. 5의 회귀식에 기반하여 계산한 결과, 수원은 4.76×103 분자/마리, 벨기에는 4.82×103 분자/마리로 계산되었으며, 상기의 결과는 SBPV가 국내에서 판매되고 있는 뒤영벌의 사체에 실제로 존재함을 보여주는 것이다.
본 PCR법을 뒤영벌 사육 현장에서 보다 쉽게 사용하기 위해서는, 성봉 또는 유충으로부터 보다 간소화된 핵산 추출법의 개발이 필요로 하며, 이러한 간편 핵산 추출법과 본 초고속 RT-PCR법이 연합될 수 있다면, 바로 현장에서 유전자 검사를 빠르게 수행하여 SBPV의 감염여부를 수 분 내에 판정할 수 있을 것이다. 더 나아가 뒤영벌의 다른 바이러스에 대하여 현장성을 보안한 신속한 검출법 개발로 연구가 확장될 것으로 기대하며 뒤영벌 수출입을 위한 검역 기준을 이용하여 집단적 발병 방지에 기여할 수 있기를 기대한다.
적 요
Slow Bee Paralysis Virus (SBPV)는 꿀벌 그리고 뒤영벌의 병원성 바이러스로써, 벌의 앞발을 마비시킴으로 폐사를 야기한다. 본 연구는, 뒤영벌에서 빠르게 SBPV 검출하고자 SBPV 특이 초고속 PCR법을 개발한 것을 보고하는 것이다.
SBPV 특이 초고속 PCR은 제반 조건을 최적화한 후, 1.0×108 분자의 SBPV 특이 유전자의 존재를 3분 35초 만에 인식할 수 있었으며, 최소 1.0×101 분자의 DNA까지 정량적으로 측정할 수 있었다. 한편, 국내외에서 생산된 뒤영벌을 시료로 SBPV 특이 초고속 역전사 PCR을 시행하였으며, 수입된 뒤영벌과 국내 생산 뒤영벌 1종에서 SBPV가 존재함을 보일 수 있었다. 국내 처음으로 뒤영벌에서의 SBPV의 존재를 확인한 것이다.
SBPV 특이 유전자 초고속 역전사 PCR법은 실험실 내에서 뿐만 아니라 현장에서 정량적 검출을 하기에 유용한 방법이 될 것이며 더 나아가 뒤영벌 수출입을 위한 검역 기준을 이용하여 집단적 발병 방지에 기여하기를 기대한다.
Acknowledgments
본 연구는 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 수출전략기술개발사업(과제번호 115067-02), 첨단생산기술개발사업(과제번호 115058-02, 과제번호 115102-03), 농생명산업기술개발사업(과제번호 312027-03)과 2017학년도 경기대학교 대학원 연구원장학생 장학금 지원에 의하여 수행되었습니다.
인 용 문 헌
- 김정민, 임수진, Truong A Tai, 홍기정, 윤병수, (2017), 초고속 유전자 증폭법을 이용한 벌집꼬마밑빠진벌레(Aethina tumida)의 신속한 검출 기법 개발, Journal of Apiculture, 32(2), p119-131.
- 민상현, 김정민, 임수진, 김병희, 이칠우, 윤병수, (2017), 뒤영벌 병원체 11종에 대한 실시간 중합효소 연쇄반응 검출법 개발, Journal of Apiculture, 32(2), p99-109.
- 민상현, 왕지희, 임수진, 이칠우, 윤병수, (2016), 초고속 역전사 실시간 중합효소 연쇄반응을 이용한 한국산 낭충봉아부패병 바이러스의 가장 빠른 검출법, Journal of Apiculture, 31(2), p121-131.
- 왕지희, 민상현, 임수진, 윤병수, (2016), 꿀벌의 석고병, 백묵병 현장 진단을 위한 신속 Ascosphaera apis 및 Aspergillus flavus 검출법 개발, Journal of Apiculture, 31(1), p31-39.
- 임수진, 김정민, 이칠우, 윤병수, (2017), 꿀벌 6종 주요 병원체에 대한 초고속 다중 PCR 검출법의 개발, Journal of Apiculture, 32(1), p27-39.
- 최용수, 이만영, 홍인표, 김남숙, 김혜경, 변규호, 윤형주, (2010), 뒤영벌류(Bombus terrestris와 Bombus ignitus)에서 꿀벌바이러스 진단, Journal of Apiculture, 25(4), p259-266.
- 최용수, 이명렬, 이만영, 김혜경, 이광길, 이경용, 윤형주, (2009), 국내 최초 서양뒤영벌(Bombus terriestris)에서의 Deformed Wing Virus (DWV) 진단, Journal of Apiculture, 24(1), p23-30.
-
Bailey, L., R. D. Woods, (1974), Three previously undescribed viruses from the honey bee, J. Gen. Virol., 25, p175-186.
[https://doi.org/10.1099/0022-1317-25-2-175]

- Dino, P. M., M. A. Furst, J. Caspar, P. Theodorou, M. J. F. Brown, and R. J. Paxton, (2015), A sting in the spit: widespread cross-infection of multiple RNA viruses across wild and managed bees, Journal of Animal Ecology, 84, p615-624.
-
Han, S. H., D. B. Lee, D. W. Lee, E. H. Kim, and B. S. Yoon, (2008), Ultra-rapid real-time PCR for the detection of Paenibacillus larvae, the causative agent of American Foulbrood (AFB), J. Invertebr. Pathol., 99, p8-13.
[https://doi.org/10.1016/j.jip.2008.04.010]