봉변을 이용한 꿀벌 바이러스성 병원체의 다중 초고속 PCR법
Abstract
Hive debris is a kind of micro-environment of beehive including excreta, mucus, epidermis of bee and carcass. Hive debris might also include almost invaded pathogens and particle of hive parasite and injurious insect against honeybee Multi-Ultra-Rapid PCR (8MUR-PCR) against eight viral pathogens in honeybee, including black queen cell virus, deformed wing virus, sacbrood virus, korean sacbrood virus, acute bee paralysis virus, chronic bee paralysis virus, slow bee paralysis virus and israeli acute paralysis virus, were optimized for one PCR condition. Using several hive debris from Suwon province, 8MUR-PCR was applied and detection-results were analyzed. In many cases, viral pathogens were found more accurately from hive debris than from bee carcass itself, even both originated from same bee-population. Based on this results, hive debris might be better than pathogenic samples for the general survey of public hygiene in single population.
Keywords:
Ultra-Rapid PCR, Honeybee pathogens, Hive debris, Detection sample, Environmental DNA (eDNA)서 론
봉변(Hive debris)은 벌통 바닥에서 발견되는 다양한 봉군의 부산물들이 한데 모인 벌통 내 작은 미소환경(micro-environment)이다. 봉변 내에는 꿀벌의 배설물 외에도 소비의 밀랍, 꿀, 꿀벌의 사체 등이 포함되어 있으며, 꿀벌 응애(Varroa detructor)와 작은벌집밑빠진벌레(Aethina tumida; SHB)와 같은 벌집 해충의 사체 또는 파편도 존재한다 (Cepero et al., 2014; Koeniger et al., 2002; Spiewok and Neumann 2006).
환경 DNA(environmental DNA; eDNA)란 일반적으로 표적 생물체를 분리하지 않고 환경 시료로부터 추출한 DNA를 의미한다(Lodge et al., 2012). 수생 생태계 상에서 유기체 채집 없이 종을 탐지하는 방법으로 eDNA가 사용되어 왔으며(Goldberg et al., 2016), 조류(bird) 종 탐지 방법으로써 eDNA를 이용하여 자연 연못으로부터 5종의 조류 종을 eDNA를 통해 검출해낸 바 있으며, 동일하게 흑해의 침입성 저서 물고기인 둥근 망둥어(Neogobius melanostomus)를 eDNA를 통해 검출한 바 있다(Nevers et al., 2018; Ushio et al., 2018).
봉변에서 미국부저병(american foulbrood; AFB)의 원인균인 Paenibacillus larvae를 검출하고, 이를 배양을 통해 봉변 속 존재 여부를 확인한 연구가 있었으며(Ryba et al., 2009; Eva and Ane, 2013), SHB 또한 봉변에서 그 존재 여부를 가릴 수 있는 시료로 사용되었다(Ward et al., 2007; Kim et al., 2017). 봉변은 흡착지(sticky paper)를 사용하는 꿀벌 응애의 검출법과 더불어 봉변은 꿀벌응애 존재여부를 가리는 시료로 제시되기도 하였다(Asha et al., 2015).
한 봉군의 병원체 존재 여부를 판정하는데 있어서 검사 시료로써 어떤 것을 선택하는 것은 매우 중요한 사항이 될 것이다. 꿀벌이 사망하는 경우 병원체의 감염 및 동사, 자연사가 주된 원인으로 작용할 것이다. 이때, 어떤 사체를 선별하는가에 따라 병원체의 감염에 의한 사멸인지 그렇지 않은지에 대한 판단 기준이 달라질 수 있으므로 어떤 꿀벌 또는 유충을 검사시료로 선택하는데 따라 병원체 존재 여부의 결과는 큰 편차를 보일 수 있다.
봉변은 벌통 내에 있는 거의 모든 사체의 최소 일부분을 담고 있을 뿐만 아니라 중장관에 서식하는 장내 병원체 또한 봉변에 섞여들어 갈 수 있어, 봉군 내의 생명체의 유전정보를 취합할 수 있는 통합 정보제공체로 기능할 수 있을 것이다. 선행 연구에서 봉변이 유전자 검출의 시료로써 유용함을 보여주었고, 또한 이를 재료로 SHB의 특이 DNA만을 선택적으로 증폭시키는 SHB 특이 초고속 PCR 검출법을 제시한 바 있다(Kim et al., 2017).
따라서, 본 연구에서는 환경에서 분해되기 쉬운 RNA virus를 표적으로 검출 가능성을 확인하고자 하였으며, 총 8개의 RNA virus (Black Queen Cell Virus; BQCV, Deformed Wing Virus; DWV, Sacbrood Virus; SBV, korean Sacbrood Virus; kSBV, Acute Bee Paralysis Virus; ABPV, Chronic Bee Paralysis Virus; CBPV, Slow Bee Paralysis Virus; SBPV and Israeli Acute Paralysis Virus; IAPV)에 대해 벌의 개별 사체로부터 추출한 핵산과 봉변으로부터 추출한 핵산으로부터 검출 양상을 확인하며, 봉변이 봉군의 병원체 검출 양상을 대변할 수 있는 시료로써의 가능성을 확인하고자 하였다.
재료 및 방법
폐사 벌통으로부터의 봉변 수집
봉변 및 벌 사체 시료는 2018년 2월, 3월, 5월 3번에 걸쳐 경기도 용인시 양봉장 한 곳, 경기도 수원시 광교양봉장 한 곳에서 원인 미상으로 폐사된 총 6통에서 각기 수집되었다. 봉변 및 벌 사체는 각기 50ml conical tube를 이용하여 별도 수집하였으며, 각 시료는 수집즉시 -20°C 냉동 보관하였다.
꿀벌 병원체 검출을 위한 primer의 설계
총 8종의 각기 다른 RNA virus의 병원체의 특이 유전자 증폭을 위하여 병원체 검출 특이 primer를 새로이 설계 또는 선행논문을 검증하여 확보하였다(Table 1).
특이 PCR증폭을 위한 primer의 염기서열은 NCBI(National Center for Biotechnology Information)에 기재된 해당 병원체의 염기서열 정보를 통하여 각 병원체에 대해 특이적인 검출이 가능하게끔 설계하였다. 각각의 primer들은 본 연구에 사용된 하나의 Rapi:chip™(Genesystem Co., Korea)에서 8개의 PCR이 독립적으로 그리고 동시에 수행되도록 PCR 온도 조건을 상호 조정하며 설계하였다.
봉변이 꿀벌의 병원체를 대변한다는 것은 꿀벌(Apis mellifera)의 유전자 또한 봉변에 포함되어 있다는 것을 의미하게 된다. 병원체 감염 및 비 감염 봉군에서 eDNA의 추출이 원활하게 진행되었음을 확인하고자 대조군으로써 증폭반응을 동시에 실시하였다. A. mellifera의 mitochondria DNA (mtDNA) 상에 존재하는 Cytochrome Oxidase Subunit Ⅰ (COI) 유전자를 표적으로 특이 primer를 추가 설계하였다.
8종 바이러스성 병원체 동시 검출을 위한 PCR 조건의 최적화
봉변으로부터 8종 바이러스성 병원체의 동시 검출을 실시하고자 각각의 병원체 특이 primer의 최적 annealing 온도를 재 측정하였다. PCR 증폭 시간의 단축을 위해서는 열의 상승 하강하는 시간인 ramping time을 줄이는 것이 가장 중요한 관점이 될 것이다. 때문에 초고속 PCR에서 혼성온도는 보다 높은 온도에서 진행함으로써 증폭에 걸리는 시간을 최소화하기에 보다 효율적이며, 높은 온도의 최적 혼성온도는 초고속 PCR에 더욱 유리할 것으로 판단하였다.
8종의 PCR은 각기 온도구배 PCR을 통하여 혼성온도 55±10°C의 범위에서 최적 온도를 선별하였으며, 이 PCR에서 각 병원체의 특이 염기서열을 탑재한 1ng 재조합 DNA(약 108분자)를 주형으로 사용하였다. 각 병원체에 대한 특이 primer쌍은 모두 최종농도 0.5μM로 조정하였으며, 10μl의 2X greenstar qPCR master mix (Bioneer Co., Korea)를 이용하여, 최종 20μl PCR 반응액으로 조성하여 수행하였다.
온도구배 PCR은 Exicycler™ quantitative thermal block (Bioneer Co.)를 사용하였으며, 초기변성 95°C, 5분, 변성 95°C, 30초, 혼성 55°C±10°C, 30초, 신장 72°C, 30초 조건하에서 30회전 PCR 증폭을 수행하였다.
현장 적용을 위한 초고속 핵산 추출법
현장에서 병원체의 특이 유전자 존재를 빠르게 검출하기 위하여 PCR 증폭 시간의 단축과 함께 현장에서 짧은 시간 내에 핵산을 추출하는 것은 매우 중요할 것이다.
본 연구에서는 봉변으로부터 핵산을 추출하기 위한 최적화된 방법을 찾고자 다음의 세가지 방법에 의한 핵산 추출을 수행하였다. 먼저 봉변 100mg을 소형 지퍼백(70×115mm)에 넣고 1ml 3차 증류수를 넣은 후 손으로 비벼 곱게 분쇄한 진탕용액을 각 100μl씩 3개의 200μl 튜브에 분주하였으며, 다음 세 가지 방법을 이용한 핵산 추출법을 수행하였다. 즉, 1) 액체질소(Liquid Nitrogen; LN2)를 이용하여 1분간 급속 냉각을 진행하고, 이후 1분 상온 해동을 하는 급속냉각법과, 2) 100°C, 1분간 정치 후 1분 냉각를 통한 고온정치법을 수행하였으며, 양 시료들은 각기 13,000rpm, 1min 원심분리하여 상등액만을 새로운 원심분리관에 옮기었다. 또한, 3) iNtRON Biotechnology사의 Viral DNA/RNA extraction kit를 사용하였으며, 핵산의 각 추출양을 비교하기 위하여, 상기의 과정에서 동일한 100μl의 진탕용액을 사용하여 DNA/RNA를 순수분리 하였다. 이때 최종 47μl의 핵산이 포함된 용액을 얻었으며, 53μl D.W.를 첨가하여 총량을 100μl로 각각 맞추었다. 이후 증폭양상 비교를 통해 각 방법에 따른 핵산의 추출양상을 비교 평가하였다.
봉변에서의 Apis mellifera mtDNA COI 유전자의 검출
봉변에서 추출된 핵산으로부터 A. mellifera의 mtDNA COI 유전자 증폭의 가능함은 봉변의 구성물이 봉군으로부터 유래되었음을 의미하며, 봉변으로부터 핵산의 추출이 이루어졌음을 의미한다. mtDNA는 환형의 DNA이며, 하나의 세포에 다량의 mitochondria를 가지고 있기에 봉변으로부터 검출하는eDNA의 관점에서 유리한 것으로 판단되었다.
증폭 반응은 GENECHECHKER™ (Genesystem Co. Ltd., Korea) 기기를 이용하여 초고속 PCR 환경에 맞추었으며, 표준 검체로써 108 분자의 재조합 DNA를 사용하여 비교분석하였으며, 1μM 최종농도의 primer 쌍, 5μl 2X Rapi master mix (Genesystem Co. Ltd.)를 이용하여 최종 10μl PCR 반응액으로 조성하여 수행하였다.
초고속 PCR 조건은 초기변성 95°C, 15초, 변성 95°C, 1초, 혼성 55°C, 3초, 신장 72°C, 3초 조건하에서 50회전 PCR 증폭을 수행하였다.
봉변 및 꿀벌 시료로부터 주요 8종 virus성 병원체의 검출
병원체에 의한 사멸이 의심된 봉군으로부터 봉변 및 꿀벌 성체로부터 핵산을 추출하였으며, 대조군으로써 각 병원체의 특이 유전자를 형질전환하여 획득 또는 확보한 각 병원체의 재조합 plasmid DNA(108분자)를 주형으로 초고속 PCR을 수행하였다. 각 시료의 PCR 증폭 양상은 초고속 정량 PCR을 통한 증폭 곡선 및 융점 곡선을 통하여 특이적, 비 특이적 증폭을 확인하였으며, 추가된 전기영동을 통하여 특이 유전자의 증폭 여부를 확인하였다. 1μM 최종농도의 8종 virus성 병원체에 대한 primer 쌍, 5μl 2X RT Rapi master mix (Genesystem Co., Korea)를 이용하여 최종 10μl PCR 반응액으로 조성하여 수행하였다.
초고속 RT-PCR (reverse transcription PCR) 조건은 역전사 반응 50°C, 1분, 초기변성 95°C, 15초, 변성 95°C, 1초, 혼성 55°C, 3초, 신장 72°C, 3초 조건하에서 50회전 PCR 증폭을 수행하였다.
결과 및 고찰
8종 바이러스성 병원체의 동시 검출을 위한 PCR 조건의 최적화
8종의 PCR 반응을 독립적으로 또한 동시에 진행하기 위한 초고속 PCR의 최적 혼성 온도를 측정하였다. 각 병원체 검출에 대한 PCR의 혼성 온도는 45°C에서 65°C의 온도구배 PCR로 진행하였으며, 실시간 PCR의 결과와 함께 전기영동상의 PCR 증폭산물의 생성 여부에 따라 각 PCR이 증폭 가능한 혼성온도의 범위를 구획하였다.
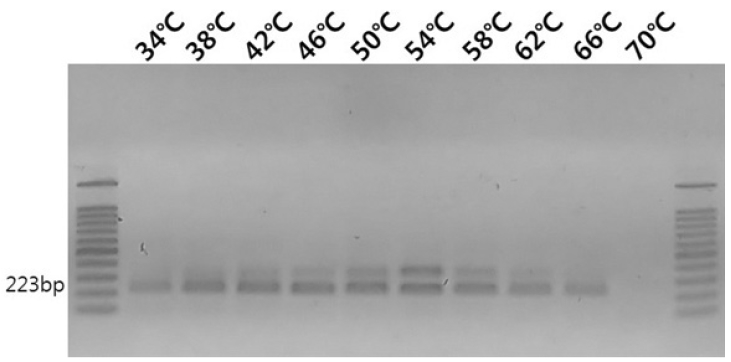
BQCV 특이 유전자 표적 primer의 경우 45°C부터 62°C까지 증폭 양상을 보였으며, DWV, kSBV, IAPV, CBPV는 45°C에서 65°C, SBV의 경우 45°C에서 60°C, ABPV는 47°C에서 65°C, SBPV는 45°C에서 57°C까지 증폭양상을 보였으며, 각각 49°C (BQCV), 59°C(DWV), 50°C (SBV), 54°C (kSBV), 54°C (IAPV), 56°C(CBPV), 53°C (ABPV), 51°C (SBPV)에서 가장 높은 증폭양상을 보였다(Fig. 1).
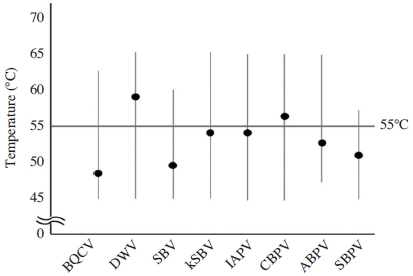
Optimal annealing temperature of qPCR against 8 viral pathogens of Apis mellifera. Gradient PCR was performed from 45°C to 65°C, for each viral pathogens. The highest efficiencies were shown in 49°C (BQCV), 59°C (DWV), 50°C (SBV), 54°C (kSBV), 54°C (IAPV), 56°C (CBPV), 53°C (ABPV), 51°C (SBPV). Considering the stability of 8 different PCRs, the optimum annealing temperature was finally determined at 55°C for all 8 PCRs.
온도 구배 PCR에서 혼성온도의 증가 혹은 감소함에 따라 높은 온도와 낮은 온도에서는 불안정한 증폭 또는 증폭되지 않는 양상을 보였으며(Fig. S1), primer의 염기서열, 결합 능력에 따라 가장 증폭이 일어나는 혼성 온도가 다르게 확인되었다.
8종의 바이러스성 병원체의 특이 유전자의 증폭양상의 경우 증폭 가능한 온도범위가 SBPV에서 가장 낮은 온도인 57°C를 보였다.
57°C의 온도에서도 모든 virus성 병원체의 특이 유전자가 증폭 가능하나, 온도범위의 양 끝의 경우 primer의 결합 및 증폭 효율에 악영향을 미치기에 보다 낮은 온도에서 증폭에 불리함을 보이지 않는 55°C의 혼성 온도를 최적 혼성 온도로 최종 결정하였다.
현장 적용을 위한 초고속 핵산 추출법
추출된 핵산의 양을 확인하고자 IAPV의 특이 유전자 및 특이 primer를 이용하여 정량 그래프를 작성하였으며, 각 반응의 조건에 맞춰 그래프를 조정하여, 각 검출법에 따른 초기 분자량을 역산하여 계산하였다(자료 미제시).
Viral DNA/RNA extraction kit를 이용하여 핵산을 추출하고 IAPV 유전자의 증폭양상을 확인한 경우 100배 희석한 주형의 경우 1.59×108 분자의 핵산이 존재하는 것으로 계산되었다. 100°C 정치를 통한 고온정치법의 경우 100배 희석 주형에서 1.42×107 분자의 핵산이 존재하는 것으로 계산되었고, LN2를 이용한 급속냉각법의 결과로 100배 희석 주형에서 4.22×107 분자의 초기 핵산이 존재하는 것으로 계산되었다(Fig. 2).
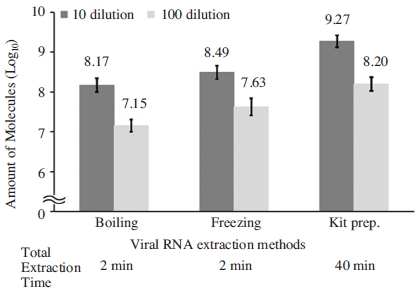
Comparison among three extraction methods of nucleic acid from hive debris. Nucleic acids were extracted by three extraction methods. Quantity of IAPV template using Viral DNA/RNA extraction kit was estimated 10 times more than quantities by other two extraction methods (boiling and freezing).
동일한 희석 주형 하에서 비교하였을 때, 100배의 희석 주형의 경우 Viral DNA/RNA extraction kit를 통하여 핵산을 추출했을 때보다, 고온정치법이 11.20배, 급속 냉동법이 3.77배 낮은 분자 수의 핵산이 추출되는 것으로 확인되었으며, 급속 냉동법 및 고온 정치법을 이용하는 핵산 추출법이 추출되는 양에서 불리한 면을 보이는 것으로 확인되었다. 그러나 시간적인 측면에서 고온정치법의 경우 단순 100°C, 1분 정치이기에 끓이는 시간을 제외한 용액을 식히는 데 1분, 총 2분의 핵산추출 시간이 필요하였으며, 급속 냉동법은 LN2를 이용한 급속 냉각 및 해동에 각 1분씩 총 2분의 시간이 소요되었다. 반면, kit의 경우 전처리 과정을 제외하고 40분이라는 비교적 긴 시간을 필요로 하였다.
꿀벌 병원체의 핵산의 개수가 102 분자 이하의 적은 양이 존재한다는 것은 봉군 내 병원체의 감염이 만연한 상황이 아닌 병원체 유입의 초기 단계로 보이며, 병원체의 감염 여부를 판단하고자 할 때, kit를 이용한 핵산 추출에 비해 급속 냉동법을 통한 핵산 추출방법이 현장성 및 신속성 측면에서 적용에 가장 유용한 방법으로 판단되었다.
봉변에서의 꿀벌 유전자의 검출
봉변에서 검출되는 유전자가 봉군으로부터 유래되었음을 확인하고자 꿀벌의 유전자가 봉변에서 검출되는 지를 확인하고자 꿀벌의 mtDNA의 COI 유전자의 증폭을 실시하였다. 동일 봉변시료에서 핵산을 추출하고, 초고속 PCR로 증폭하였으며, 수원시 및 용인시로부터 확보한 봉변의 시료 모두에서 증폭양상을 보임을 확인하였으며, PCR 증폭산물의 융점분석을 통하여 대조군으로 증폭한 108 분자의 COI 재조합 DNA의 증폭과 비교하였을 때, 양성 Tm값의 오차범위(±0.5°C)에 포함되어 있으며, 동일한 증폭산물이 형성되었음을 확인하였다(Fig. 3). 이는 추가 전기영동을 통해서 특이적 밴드의 증폭을 확인하였다(자료 미제시).
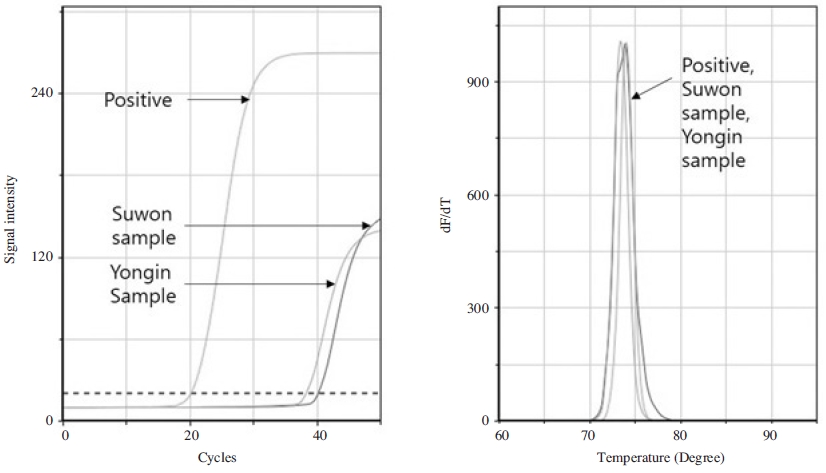
Amplification of COI genes for Apis mellifera using ultra rapid qPCR from hive debris in Suwon and Yongin. COI genes of A. mellifera from hive debris were amplified using ultra rapid PCR. A. mellifera COI recombinant DNA of 108 molecules was amplified together to confirm amplification pattern. Amplification reactions were observed in both Suwon and Yongin samples, it was confirmed that specific amplification occurred by melting point analysis.
mtDNA는 한 세포 내 다수가 존재하며, 환형의 유전체를 갖고 있기에 보존성에서 유리한 것으로 사료되며, 100mg의 소량의 봉변에 꿀벌의 사체 일부만 섞어있어도 능히 검출 가능함을 보여주고 있다.
봉변 및 꿀벌사체에서 증폭된 8종 Virus성 병원체 특이 유전자의 검출 양상
경기도 용인시와 수원시의 봉변 시료 및 꿀벌의 사체 시료로부터 바이러스성 병원체의 존재 유무를 확인하고자 각 바이러스성 병원체 특이 primer를 이용하여 8종 다중 초고속 PCR 증폭을 실시하였다.
경기도 용인시의 봉변 및 꿀벌 사체로부터 유전자를 추출하여 병원체 유전자를 증폭한 결과 DWV와 SBV, IAPV의 특이 유전자가 특이 증폭됨을 확인하였다.
각 증폭 양상에서 꿀벌 사체 시료와 봉변 양쪽에서 모두 특이 PCR산물의 증폭반응을 확인하였으며, 또한 전기영동 상에서도 각 특이 PCR 산물인 223bp(DWV), 214bp (IAPV)를 확인할 수 있었다.
이들은 초고속 PCR에서 대조군으로 사용한 각 특이 유전자의 재조합 DNA 주형 108 분자의 증폭 양상과 비교하였으며, 모든 봉변 시료에 DWV와 IAPV 초기 주형이 꿀벌 사체의 그것보다 더 많은 것으로 측정되었다. 즉 DWV의 각 Ct값은 35.09cycles(봉변) 그리고 47.19cycles(꿀벌사체)로 계산되었으며, IAPV의 각 Ct값은 26.18cycles(봉변) 그리고 35.22cycles(꿀벌사체)로 계산되었다. 이 양적 차이는 전기영동상에서 산물의 강약으로도 볼 수 있었다(Fig. 4; Fig. 5).
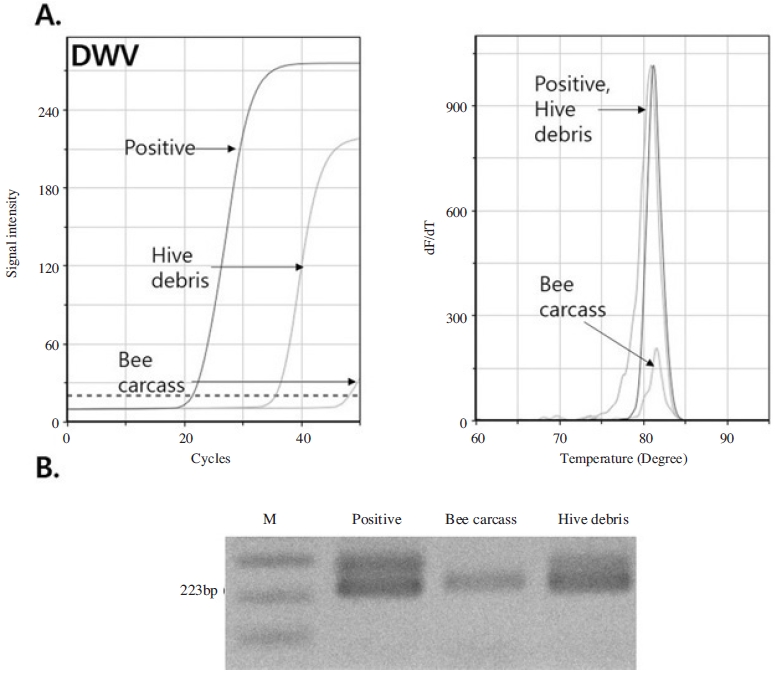
Specific amplification of ultra rapid PCR against Deformed Wing Virus from bee carcass and hive debris in Yongin. Specific gene of Deformed Wing Virus was amplified by ultra-rapid PCR. Panel A. Amplification curve and melting analysis of ultra rapid PCR showed positive reaction in all 3 different templates. Panel B. Specific amplicon of DWV on electrophoresis.
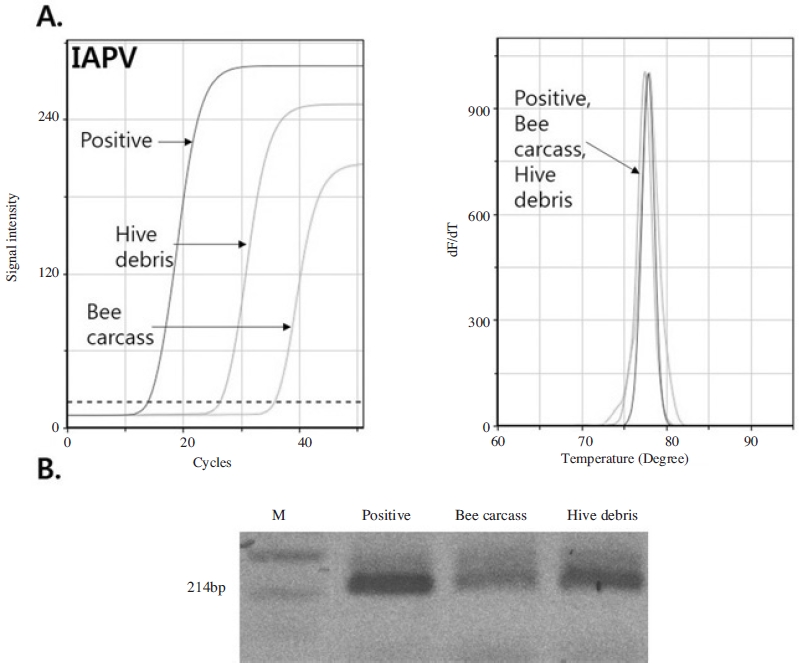
Specific amplification of ultra rapid PCR against Israeli Acute bee Paralysis Virus from bee carcass and hive debris in Yongin. Specific gene of Israeli Acute bee Paralysis Virus was amplified by ultra-rapid PCR. Panel A. Amplification curve and melting analysis of ultra rapid PCR showed positive reaction in all 3 different templates. Panel B. Specific amplicon of IAPV on electrophoresis.
SBPV의 특이 유전자 증폭에서 초고속 PCR 상에서는 재조합 DNA를 반응한 대조군과 벌 사체, 봉변의 유전자 모두에서 증폭반응이 일어난 것으로 모니터링 상에서 확인되었으나, 융점분석을 실시하였을 때, 꿀벌 사체의 경우 그 Tm (melting temperature)값이 재조합 DNA를 증폭한 Tm값과 4.21°C 차이를 보이며(positive: 81.39°C, bee carcass: 77.18°C), 이는 특이 유전자의 증폭이 아닌 비 특이적인 증폭이 일어난 것으로 확인되었다. 이는 전기영동 상에서도 100bp 마커보다 아래서 증폭양상이 확인되었으며, SBPV의 특이 유전자가 존재하지 않는 것으로 확인되었다.
하지만, 봉변 시료의 경우 Tm값의 차이에서 1.05°C의 차이를 보이고 있으며(positive: 81.39°C, hive debris: 79.44°C), 이는 순수 Virus의 RNA 핵산만을 추출한 것이 아닌 봉변에 포함된 A. mellifera의 유전자 및 박테리아성, 진균성 병원체의 유전자 또한 존재 가능하며, 나아가 꿀벌 해충인 꿀벌 응애의 유전자가 모두 포함될 수 있을 것이다. 이에 mismatch 되는 유전자가 비특이적으로 증폭 된 것으로 보이며, 전기영동 상에서도 특이적 밴드(224bp) 및 비 특이적 밴드가 확인되었다(Fig. 6).
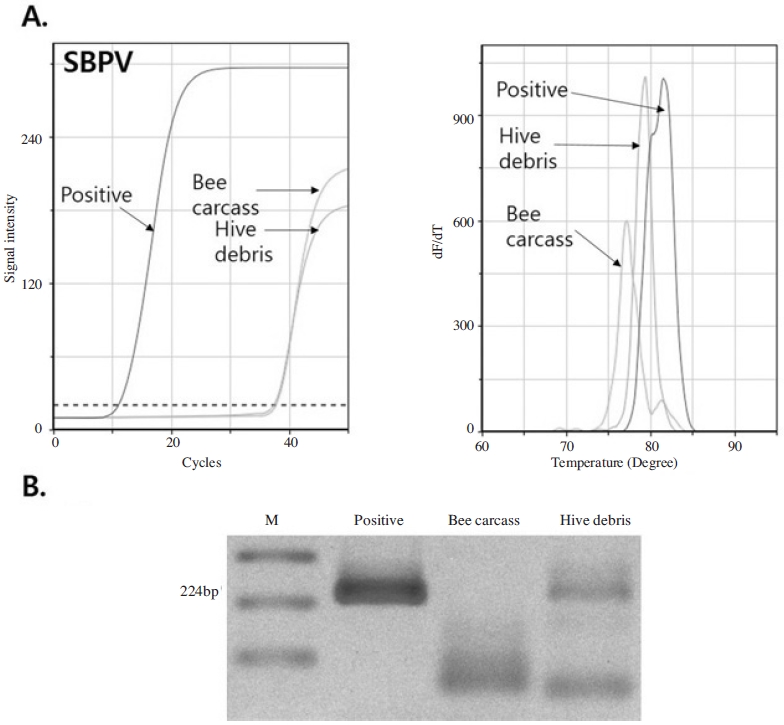
Specific amplification of ultra rapid PCR against Slow Bee Paralysis Virus from bee carcass and hive debris in Yongin. SBPV-like amplicon was amplified by ultra-rapid PCR. Panel A. Amplification curves from 3 different templates showed differences of Cts. In melting analysis 3 amplicons were measured at different Tms. Panel B. Specific amplicons of SBPV on electrophoresis. SBPV-specific 224-bp long amplicon was not recognized in lane of Bee carcass.
이러한 증폭의 불안정성은 nested PCR 및 3군데의 다른 특이적 유전자 부위를 증폭하는 3 point multiplex PCR 등에 의해 2차 판별이 필요한 경우로 판단된다.
허나 일차적으로 전기영동 상에서 예상 증폭 크기인 224bp에서 밴드를 형성함을 확인할 수 있었으며, 이는 꿀벌 사체에서는 SBPV 특이 유전자의 부재로 인하여 증폭 반응을 확인 할 수 없었던 반면, 봉변의 시료에서는 SBPV의 특이 유전자가 증폭되었으며, 이는 봉변이 병원체 검출 시료로써 꿀벌 사체보다 유용할 수 있음을 내포하는 결과로 보여진다.
BQCV, CBPV, ABPV의 경우 봉변 및 꿀벌 사체에서 특이 유전자 검출 양상이 확인되지 않았으며, 전기영동 상에서도 동일한 결과로 확인되었다(Fig. 7).
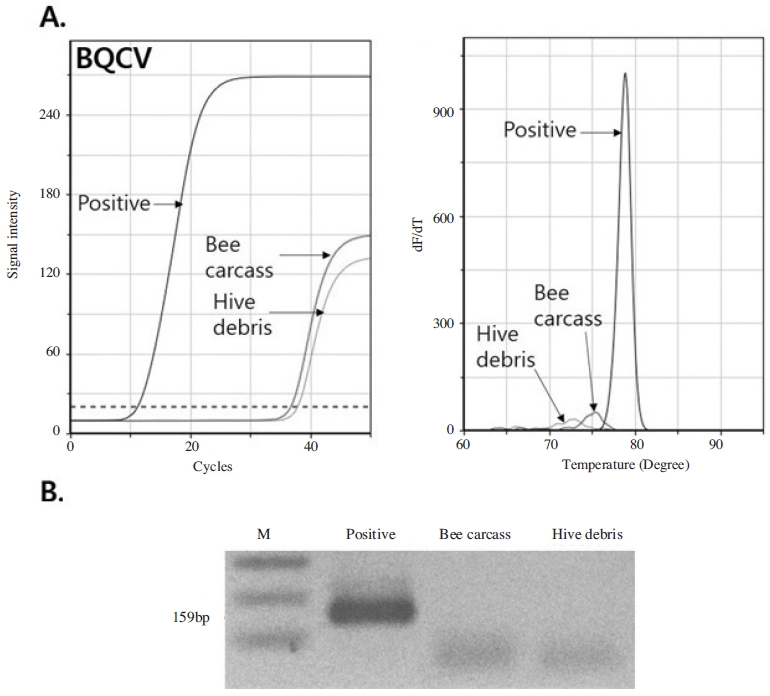
Specific amplification of ultra rapid PCR against Black Queen Cell Virus from bee carcass and hive debris in Yongin. BQCV-like amplicon was amplified by ultra-rapid PCR. Panel A. Amplification curves from 3 different templates showed differences of Cts. In melting analysis 3 amplicons were measured at different Tms. Panel B. BQCV-like amplicons on electrophoresis. BQCV-specific 159-bp long amplicon was not recognized in lane of Bee carcass or hive debris.
경기도 용인시 및 수원시의 꿀벌 사체, 봉변 시료는 반복실험에 의하여 병원체 검출을 실시하였다. 전체 반응 횟수 중 바이러스성 병원체 검출에서 양성 반응을 보인 횟수를 기재하였으며, 양성 검출 횟수를 총 반응 횟수로 나누어 백분율(%)로 표기하였다.
용인시의 시료의 경우 BQCV, CBPV, ABPV의 특이 유전자의 증폭에서 벌 사체와 봉변시료 모두 양성 검출이 이루어지지 않았으며, kSBV의 경우 특이적으로 4마리의 벌 사체 중 1마리의 벌 사체에서만 검출이 확인되었다. 허나 DWV, SBV, IAPV, SBPV의 특이 유전자의 증폭에서 사체 벌의 경우 선택되는 벌 시료에 따라 바이러스의 감염 정도가 다르며, 비 감염 시료가 존재하는 것으로 확인되었으나, 봉변 시료의 경우 서로 다른 위치에서 증폭하였음에도 불구하고 모두 양성반응으로 나타남이 확인되었다(Table 2).

Total result of amplification against 8 viral pathogens using ultra rapid PCR from bee carcass and hive debris samples in Yongin samples
수원시 시료의 경우 BQCV, CBPV의 특이 유전자는 사체 벌 및 봉변 시료 모두에서 검출되지 않았으며, SBV, kSBV, ABPV의 특이 유전자의 경우 사체 벌 일부에서만 검출이 되었으며, 봉변 시료에서는 검출되지 아니하였다. 허나 DWV, IAPV의 경우 봉변 시료에서는 모든 반응에서 양성 검출로 확인되었으나, 사체 벌의 경우 선택된 서로 다른 벌 중 절반 이하의 사체 벌 시료에서만 검출된 것으로 확인되었다. 또한 SBPV의 경우 두 시료 모두에서 병원체의 특이 유전자 검출이 이루어졌으나 일부 시료에서만 검출된 것으로 확인되었다(Table 3).

Total result of amplification against 8 viral pathogens using ultra rapid PCR from bee carcass and hive debris samples in Suwon
꿀벌의 병원체 검출 및 진단에서 감염된 유전자의 특이적인 증폭은 결과에 신뢰성을 부여할 것이다. 그러나 사체 꿀벌의 경우 선택되는 꿀벌에 따라서 병원체의 존재 여부가 서로 다르며, 병원체에 의해 사멸한 봉군이 아닌 일반적인 봉군에 대한 병원체의 검사에서 이러한 결과는 커다란 검사결과의 편차를 보일 수 있으며, 병원체의 진단의 신뢰성을 떨어뜨리는 결과로 이어질 수 있다.
물론, 감염이 분명한 폐사 벌통에서 감염된 사체를 이용하여 병원체 검출 및 감염여부를 진단하는 것은 바이러스성 병원체를 포함한 꿀벌 병원체의 양적인 면에서 더욱 유리한 시료일 것이다. 허나 병원체 감염이 만연하지 않은 상황에서 살아있는 성체벌및유충을 선택한다는 것은 자칫 건강한 벌 및 유충으로부터 병원체의 유무를 판단하고자 하는 무의미함이 있을 수 있다. 이에 봉변에서의 병원체 유전자의 검출은 봉군의 여러 환경을 모두 아우를 수 있는 하나의 공통분모가 될 것이며, 감염된 성체벌 및 유충의 선별 없이 봉군의 병원체 감염 현상을 간편하게 모니터링 할 수 있는 방법이 될 것이다.
본 연구는 이러한 단점을 보완하고자 벌통 내에서 병원체의 감염양상을 대변할 수 있는 시료로써 봉변의 사용 가능성을 제시하고자 하였다. 병원체 감염여부 판별을 위해 선택된 벌 시료의 불확실한 병원체 특이 유전자 증폭의 위험성을 제시하며, 벌통 내부에서 공통분모로써 병원체 감염 양상을 대변할 수 있는 시료로써, 봉변을 새로이 제안하며, 벌통 내에서 병원체 감염을 포함한 하나의 미소환경으로써 우리에게 중요한 정보를 제공해 줄 수 있음을 기대한다.
봉변이 우리에게 주는 정보는 병원체의 존재 여부를 넘어서 꿀벌 응애와 벌집꼬마밑빠진벌레, 현재 국내 보고사례는 없으나 꿀벌 기생해충으로 알려진 기문 응애 등의 존재 여부를 분자적으로 확인할 수 있는 시료로써 이용 가능할 것이며, 육안으로 측정이 불가능한 봉군 내 꿀벌 응애의 개체 수 측정 또한 봉변을 통한 분자적 계수가 가능할 수 있음을 강조하고자 한다.
봉변의 미소환경적 시료로써 사용가능함을 확인하고 정확한 병원체 환경을 대변하기 위하여 봉변 시료를 이용한 병원체의 검출법 개발, 개선하는 것은 우리의 다음 목표가 될 것이며, 이를 통하여 봉군 내 다양한 변화 양상을 보다 쉽게 모니터링이 가능하게 될 것이다.
본 연구에서 제안된 봉군 내 병원체 감염 환경을 대변하는 시료로써의 봉변의 사용은 병원체 감염 확인을 위해 선택되는 시료에 따른 불분명한 증폭 양상을 포함할 수 있음을 보여주고 있으며, 병원체 검출의 신뢰성을 더욱 증대시킬 수 있는 시료라 할 것이다. 본 연구에서 제안하는 병원체 검출 시료로써 봉변의 사용이 병원체 검출 및 제어에 널리 사용되길 기대하는 바이다.
Acknowledgments
본 연구는 2018학년도 농촌진흥청 연구사업(세부과제번호: PJ01408002) 지원에 의하여 수행되었으며, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(115102-03)과 중소기업청에서 지원하는 2017년도 산학연협력 기술개발사업(C0563751)에 의한 연구 결과물임을 밝힙니다. 또한 2018학년도 경기대학교 대학원 연구원 장학생 장학금 지원에 의하여 수행되었습니다.
References
- Asha, R. Gulati, S. K. Sharma, and D. R. Aneja, (2015), Comparative evaluation of sticky paper and hive debris as sampling methods for population assessment of Varroa destructor in Apis mellifera colonies, Indian Journal of Agricultural Research, 49(6), p503-508.
-
Cepero, A., M. Higes, A. M. Salvador, A. Meana, and R. M. Hernandez, (2014), A two year national surveillance for Aethina tumida reflects its absence in Spain, BMC Research Notes, 7, p878.
[https://doi.org/10.1186/1756-0500-7-878]

- Eva, F., and A. T. Laugen, (2014), Prognostic value of using bee and hive debris samples for the detection of American foulbrood disease in honey bee colonies, Apidologie, 45, p10-20.
- Goldberg, C. S., C. R. Turner, K. Deiner, K. E. Klymus, P. F. Thomsen, M. A. Murphy, S. F. Spear, A. McKee, S. J. O. McCance, R. S. Cornman, M. B. Laramie, A. R. Mahon, R. F. Lance, D. S. Pilliod, K. M. Strickler, L. P. Waits, A. K. Fremier, T. Takahara, J. E. Herder, and P. Taberlet, Critical considerations for the application of environmental DNA methods to detect aquatic species, Methods in Ecology and Evolution, 7, p1299-1307.
-
Kim, J. M., S. J. Lim, T. A. Tai, K. J. Hong, and B. S. Yoon, (2017), Development of Rapid Detection System for Small Hive Beetle (Aethina tumida) by using Ultra-Rapid PCR, Journal of Apiculture, 32(2), p119-131.
[https://doi.org/10.17519/apiculture.2017.06.32.2.119]

-
Koeniger, G., N. Koeniger, D. L. Anderson, C. Lekprayoon, and S. Tingek, (2002), Mites from debris and sealed brood cells of Apis dorsata colonies in Sabah (Borneo) Malaysia, including a new haplotype of Varroa jacobsoni, Apidologie, 33, p15-24.
[https://doi.org/10.1051/apido:2001005]

-
Lodge, D. M., M. L., C. R. Turner, C. L. Jerde, M. A. Barnes, L. Chadderton, S. P. Egan, J. L. Feder, A. R. Mahon, and M. E. Pfrender, (2012), Conservation in a cup of water: estimating biodiversity and population abundance from environmental DNA, Molecular Ecology, 21, p2555-2558.
[https://doi.org/10.1111/j.1365-294x.2012.05600.x]

-
Masayuki, U., M. Koichi, S. Tetsuya, N. Isao, T. Masamichi, I. Wataru, and M. Masaki, (2018), Demonstration of the potential of environmental DNA as a tool for the detection of avian species, Scientific Reports, 8, p4493.
[https://doi.org/10.1038/s41598-018-22817-5]

-
Min, S. H., J. H. Wang, S. J. Lim, C. W. Lee, and B. S. Yoon, (2016), Development of Quantitative Real-time Recombinase Polymerase Amplification (qRT-RPA) Method for Quantitative Detection against Pathogenic Virus in Honeybee, Journal of Apiculture, 31(2), p147-156.
[https://doi.org/10.17519/apiculture.2016.06.31.2.147]

-
Min, S. H., J. M. Kim, S. J. Lim, B. H. Kim, C. W. Lee, and B. S. Yoon, (2017), Development of real-time PCR Detections against 11 Pathogens of Bombus Species, Journal of Apiculture, 32(2), p99-109.
[https://doi.org/10.17519/apiculture.2017.06.32.2.99]

-
Nevers, M. B., M. N. Byappanahalli, C. C. Morris, D. Shively, K. P. Kelly, A. M. Spoljaric, J. Dickey, and E. F. Roseman, (2018), Environmental DNA (eDNA): A tool for quantifying the abundant but elusive round goby (Neogobius melanostomus), Plos One, 13(1), e0191720.
[https://doi.org/10.1371/journal.pone.0191720]

-
Ryba, S., D. Titera, M. Haklova, and P. Stopka, (2009), A PCR method of detection American Foulbrood (Paenibacillus larvae) in winter beehive wax debris, Veterinary Microbiol, 139, p193-196.
[https://doi.org/10.1016/j.vetmic.2009.05.009]

-
Spiewok, S., and P. Neumann, (2006), Cryptic low-level reproduction of small hive beetles in honey bee colonies, Journal of Apicultural Research and Bee World, 45(1), p47-48.
[https://doi.org/10.1080/00218839.2006.11101313]

-
Wang, J. H., D. B. Lee, S. J. Ku, M. C. Peak, S. H. Min, S. J. Lim, C. W. Lee, and B. S. Yoon, (2016), Development of a Detection Method against 11 Major Pathogens of Honey Bee using Amplification of Multiplex PCR and Specific DNA-chip, Journal of Apiculture, 31(2), p133-146.
[https://doi.org/10.17519/apiculture.2016.06.31.2.133]

-
Ward, L., M. Brown, P. Neumann, S. Wilkins, J. Pettis, and N. Boonhan, (2007), A DNA method for screening hive debris for the presence of small hive beetle (Aethina tumida), Apidologie, 38(3), p272-280.
[https://doi.org/10.1051/apido:2007004]