
Acute Bee Paralysis Virus (ABPV)의 정확한 검출을 위한 Nested 초고속 PCR법 개발
Abstract
Acute bee paralysis virus (ABPV) is an infectious disease causing paralysis in healthy honeybee colonies. Because ABPV is highly homologous with relative viruses, it is difficult to distinguish ABPV from especially IAPV or KBV by molecular diagnosis. In this study, for accurate and specific detection of ABPV, a secondary nested PCR detection method following the primary detection PCR against ABPV was developed. It could be able to detect from less than 100 molecules of ABPV. The detection time was also minimized from RNA extraction to PCR. Using ultra-rapid ABPV-specific detection and nested PCR, the presence of ABPV was detected from infected bee samples within 50 minutes. It might be expected that this kind of detection would be helpful for accurate detection and monitoring of ABPV in environment.
Keywords:
Acute bee paralysis virus (ABPV), Nested Ultra-Rapid PCR, Field detection서 론
꿀벌의 병원성 바이러스들은 양봉에서 큰 영향을 미치고 있으나, 지난 수십 년간 그 실체를 파악하는데 연구가 집중되어 왔다(McMahon et al., 2018). 꿀벌에 질병을 일으키는 바이러스는 전 세계적으로 Deformed wing virus(날개불구병 바이러스, DWV), Acute bee paralysis virus(급성 꿀벌마비병 바이러스, ABPV), Apis iridescent virus, Chronic bee paralysis virus(만성 꿀벌마비병 바이러스, CBPV), Black queen cell virus(여왕벌흑색병 바이러스, BQCV), Cloudy wing virus (CWV), Kashmir bee virus (KBV), Sacbrood virus(낭충봉아부패병, SBV), Israeli acute bee paralysis virus(이스라엘 급성 꿀벌마비병 바이러스, IAPV) 등 18종 이상이 알려져 있다(유 등, 2007; 최 등, 2007). 한편, 국내에서 이 등(2005)에 의해 국내산 꿀벌로부터 DWV의 존재가 최초로 확인된 이래로, ABPV, BQCV, CBPV, IAPV, Kakugo virus (KV), KBV, kSBV, SBV 등 9종 이상의 꿀벌 바이러스가 존재함이 보고되었다(유 등, 2007; 최 등, 2007; 이 등, 2012; 민 등, 2016; 왕 등, 2016).
이들 꿀벌 바이러스들은 모두 양성 단일가닥(Positive Single-strand)의 RNA-genome을 가지며, 생활환 중 DNA stage가 없고 바이러스 분류학상 Picornavirales에 속한다(Chen and Siede, 2007; de Miranda et al., 2010a; b).
급성 꿀벌마비병 바이러스(ABPV)는 처음 발견 당시에 꿀벌(Apis mellifera)의 불분명한 감염의 원인으로 설명되었다(Bailey et al., 1963). 이후 ABPV의 존재는 전 세계 여러 국가에서 보고되었고(Carpana et al., 1991; Faucon et al., 1992; Hung et al., 1999), 건강한 군집에서 자주 발견되는 벌의 일반적인 감염인자로 간주되었다(Bakony et al., 2002). 이 바이러스는 꿀벌 응애(Varroa destructor)가 침입한 꿀벌 군집의 갑작스러운 붕괴의 원인이 된다고 추정되었다(Bakony et al., 2002).
ABPV는 같은 과인 Dicistroviridae에 속하는 KBV 및 IAPV와 특히 생물학적 및 유전적 변이의 중요한 요소가 밀접하게 연관되어 또한 실제로 이들 바이러스 간의 염기서열 상동성은 95% 이상이기에, 꿀벌 바이러스 검출에 주로 사용되는 Polymerase Chain Reaction(PCR)법을 기반으로 한 유전자 검사법에서 많은 오진 사례를 보이고 있다(민 등, 2016). 이런 이유로 이들, ABPV, KBV, IAPV를 하나의 복합체(AKI-complex)로 표현되기도 하였다(de Miranda et al., 2010a).
한편, PCR법은 일반적으로 100 분자 이하의 주형 DNA에 대하여 특이 증폭이 어려운 것으로 알려져 있다. 꿀벌바이러스의 감염을 초기에 진단하기 위하여, 100 분자 이하의 목적 유전자를 증폭시키는 방법이 요구되고 있으며, 2번의 연속된 PCR을 수행하는 nested PCR법이 이 문제를 극복할 수 있는 대안으로 제시되었다.
Nested PCR법은 목적유전자에 대한 1차 PCR 증폭을 수행한 후, 형광 또는 염색으로 인지할 수 없는 1차 PCR에서 생산된 극소량 증폭산물을 주형으로, 독립적인 2차 PCR을 수행하는 것이다. 이때, 2차 PCR을 위한 nested primer는 1차 PCR의 primer (detection primer)와 중첩되지 않도록 그 내부에 위치하게 설계하여, 보다 특이성을 높이게 된다.
본 연구는 ABPV 특이 진단에 nested PCR법을 적용시킴으로, 시료 중 극소량의 ABPV를 검출할 수 있는 ABPV 특이 검출법을 고안하고자 하였으며, 동시에 근연종인 IAPV 및 KBV와 분명한 구분이 될 수 있도록 하였을 뿐 아니라, 초고속 PCR을 적용함으로써 양봉 현장에서도 즉석 검사가 가능하게 하였다. 이로써 환경 내 극소량으로 존재할 수 있는 ABPV를 정확히 모니터링 할 수 있는 새로운 도구로 활용되기를 기대한다.
재료 및 방법
공시재료
질병에 의해 치사된 것으로 의심되는 성체 꿀벌(Apis mellifera)시료는 경기도 수원, 경기도 용인, 강원도 강릉, 경북 의성, 전남 여수, 울산, 제주로부터 수집되었으며, 수집된 시료들은 -20°C에서 보관하며 본 연구에 사용하였다.
ABPV 특이 유전자의 인공합성 및 Molecular Cloning
ABPV는 국내에서 확진된 사례가 매우 드물고, 정확한 ABPV 유전체를 확보하는 것은 지극히 어려운 일이었다. 정확한 ABPV 특이 유전자를 확보하기 위하여 유전자 인공합성을 수행하였으며, Genbank에 보고된 ABPV 유전자(Accession No. AF150629)의 RNA dependent RNA polymerase (RdRp) 유전자 중 근연종 바이러스인 KBV 및 IAPV의 RdRp와 분명히 구분될 수 있는 부위를 대상으로 인공합성 유전자를 설계하였다(Fig. 1).
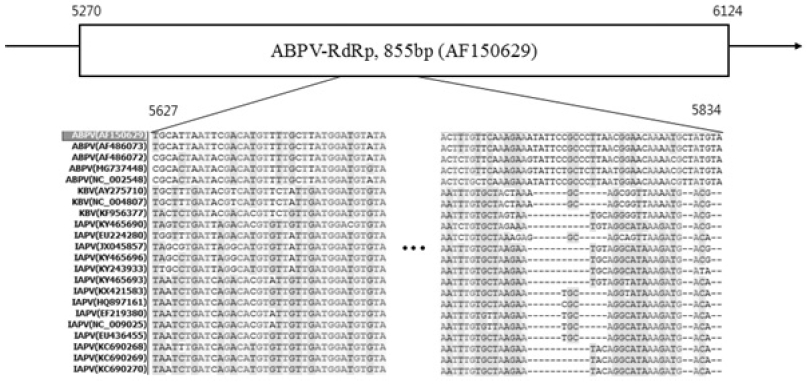
Alignment among 17 sequences from RdRp in ABPV, KBV and IAPV. From full genome sequences of 4 ABPV, 3 KBV and 23 IAPV, 17 RdRp genes were selected and aligned. In the RdRp gene of ABPV, region that clearly differed from KBV and IAPV were selected and artificially synthesized.
ABPV의 RdRp 특이 유전자는 208 염기였으며, 추후 활용을 위한 제한효소 BamHI과 HindIII의 절단자리 18 염기를 부가하여 총 226 염기 크기로 설계하였고, 이는 ABPV-226이라 명명하였다. ABPV-226의 제작을 위하여 6개의 long-nucleotide를 설계하였고 주문 제작한 후(Bionics, Korea), 인공합성을 수행하였다(Table 1).
인공합성의 방법을 약술하면, ABPV-RdRp-F1/R1을 사용하여 104 염기 크기의 1차 주형을 생산하였으며, 이는 ABPV-RdRp-F1/R1의 각 최종농도 0.25μM, TOPsimple™ DryMIX-HOT (Enzynomics, Korea)를 넣어 각 반응 당 20μl의 PCR 반응액이 되도록 조성하였고, 1차 주형 생산을 위한 template-less PCR의 조건은 초기변성 95°C 5분 후, 변성 95°C 30초, 혼성 50°C 30초, 신장 72°C 30초를 1회전으로 M총 35회전을 진행하고, 추가신장 72°C 5분을 진행하였다.
생산된 1차 주형(104 bp)은 1/1,000로 희석하여, ABPV-RdRp-F2/R2를 사용한 2차 주형의 제작에 사용하였으며, primer를 제외하고 PCR의 조성 및 조건은 1차 주형의 제작과 동일하게 수행하였다.
생산된 2차 주형(181 bp)은 1/1,000로 희석하여, ABPV-RdRp-F3/R3를 사용한 최종 3차 주형의 제작에 사용하였으며, primer를 제외하고 PCR의 조성 및 조건은 1차 주형의 제작과 동일하게 수행하였다.
각 주형이 제작되었을 때 1.5% agarose 전기영동을 수행하여 정확한 합성여부를 분석하였다. 최종 주형을 합성한 후, 인공 합성된 ABPV 특이 유전자(ABPV-226)를 탑재한 재조합 DNA를 제작하였으며, 이 ABPV 특이 재조합 DNA는 pABPV-RdRp226라 명명하였다. 탑재된 염기서열의 sequencing 분석은 전문회사에 의뢰하였으며(Bionics, Korea), 목적했던 ABPV-226임을 확인하였다.
ABPV 특이 유전자 검출을 위한 특이 primer 쌍들
ABPV의 특이 유전자를 검출하기 위한 특이 primer 쌍들을 설계하였다. ABPV 검출용으로 적합한 특이 primer의 선발을 위하여, NCBI (National center for biotechnology information)의 Primer-BLAST 프로그램을 사용하였으며, KBV와 IAPV의 오검출 없이 ABPV만을 특이적으로 검출할 수 있는 detection primer 쌍(ABPV-RdRp-DF/DR) 및 nested primer 쌍(ABPV-NT-F/R)을 설계할 수 있었다(Table 2).
ABPV 특이 detection 및 nested primer 쌍들의 최적 농도
ABPV 특이 detection PCR 및 nested PCR의 조성에서 detection primer와 nested primer의 최적농도를 구하기 위하여, 각 ABPV 특이 primer 쌍들의 최종농도를 500nM (5pmole/μl), 1.0μM (10pmole/μl), 2.0μM(20pmole/μl), 4.0μM (40pmole/μl)으로 조성하여 초고속 PCR을 수행하였다.
각 반응 당 재조합 DNA pABPV-RdRp226 1.0×108 분자를 주형으로 사용하였고, 2×Rapimix (Genesystem Co., Korea)에 각 농도의 primer 쌍을 넣어 반응액 당 총 10μl가 되도록 조성하여 초고속 PCR을 수행하였다. 이때 detection primer를 사용한 초고속 PCR 조건은 초기 변성 95°C 30초 후, 변성 95°C 2초, 혼성 55°C 2초, 신장 72°C 2초를 1회전으로 하여 총 50회전이었으며, nested primer를 사용한 초고속 PCR 조건은 초기변성 95°C 30초 후, 변성 95°C 2초, 혼성 45°C 2초, 신장 72°C 2초를 1회전으로 총 50회전이었다.
ABPV 특이 detection 및 nested PCR의 최적 혼성온도
ABPV의 신속, 정확한 검출을 위하여, 초고속 ABPV 특이 detection PCR 및 초고속 ABPV 특이 nested PCR의 최적 혼성온도 조건을 구하였다. 초고속 PCR을 위하여 30회전의 PCR을 12분 이내에 마칠 수 있는 Genechecker™ (Genesystem Co., Korea)를 사용하였으나, 먼저 온도구배 PCR (Temperature-gradient PCR)이 가능한 PCR 기기인 PTC-200 Peltier thermal cycler (MJ Research, USA)를 사용하여 50±10°C의 온도범위에 서각 PCR의 혼성가능 온도를 추론하였다.
각 반응 당 재조합 DNA pABPV-RdRp226 1.0×108 분자를 주형으로 사용하였고, 각각의 특이 primer쌍의 최종농도 1.0μM, TOPsimple™ DryMIX-HOT를 사용하여 최종 20μl PCR 반응액이 되도록 조성하였다.
결과는 1.5% agarose 전기영동으로 분석하였으며, 각 DNA 증폭산물의 density를 Image J 프로그램(https://imagej.nih.gov/ij)으로 측정하여 각 primer 쌍의 혼성가능 온도를 추론하였고, 이 결과를 초고속 PCR에 적용하여 검증하였다.
Primer ABPV-RdRp-DF/DR을 사용한 초고속 ABPV 특이 detection PCR은 혼성온도 50°C, 52°C, 54°C, 56°C에서 수행하였으며, 각 반응 당 재조합 DNA pABPV-RdRp226 1.0×108 분자부터 1.0×100 분자까지를 주형으로 사용하였고, 특이 primer의 최종농도는 1.0μM, 2×RapiMix를 사용하여 최종 10μl PCR 반응액이 되도록 조성하였다. PCR 조건은 초기변성 95°C 30초이후, 변성 95°C 2초, 혼성 각 50°C, 52°C 54°C, 56°C에서 2초, 신장72°C 2초를 1회전으로 하여 총 50회전 수행하였다.
Primer ABPV-NT-F/R을 사용한 초고속 ABPV 특이 nested PCR은 46°C, 48°C, 50°C, 52°C에서 수행하였으며, PCR의 조성은 nested primer를 사용한 것을 제외하면 ABPV 특이 detection PCR과 동일하게 조성하였다. PCR 조건은 초기변성 95°C 30초 이후, 변성 95°C 2초, 혼성 각 46°C, 48°C, 50°C, 52°C에서 2초, 신장 72°C 2초를 1회전으로 하여 총 50회전 수행하였다.
ABPV 특이 detection 및 nested PCR의 최단 검출시간 및 검출 한계 측정
ABPV 특이 detection PCR 및 nested PCR의 최적조건 하에서 회전 중 각 단계의 시간을 감소시켜 총 PCR시간의 단축을 시도하였다.
최적조건을 확립한 2쌍의 ABPV 특이 primer는 재조합 DNA pABPV-RdRp226을 1.0×108 분자부터 1.0×100 분자까지 연속 희석한 것을 초고속 PCR의 주형으로 사용하였으며, 2×RapiMix와 최종농도 4.0μM의 primer를 사용하여 총 10μl PCR 반응액으로 조성하였다. 초고속 PCR 증폭 반응은 총 50회전을 진행하였는데 회전의 각 단계의 시간을 순차적으로 2초에서 1초로 감소시키면서 초고속 PCR에 필요한 최단 증폭시간과 검출한계를 동시에 측정하였다.
최적조건을 최적화하고 최단 검출시간을 측정한 뒤, 이에 따른 검출한계와 정량범위를 측정하였으며 Ct (Threshold Cycles)와 주형 분자간의 회귀식(regression equation) 및 회귀상수(regression coefficient, R2)를 확인하였다.
공시시료로부터 ABPV 특이 유전자 검출
꿀벌시료에서 RNA의 추출은, 먼저 꿀벌에서 날개를 제거하여 1.5ml 원심분리관에 넣고 100μl D.W.를 부가한 후, 액체질소(LN2)에 잠기게 하여 1분간 동결한 것을 상온에서 10분간 정치하여 녹이고, 1.5ml 원심분리관 전용 homogenizer를 사용하여 분쇄하였다. 분쇄액은 1분간 13,000rpm에서 원심분리하여, 그 상등액 1.0μl를 ABPV 특이 PCR의 초기기질로 사용하였다.
ABPV 특이 detection primer를 사용한 1차 PCR은 2×RT RapiMix (Genesystem Co., Korea), 최종농도 4.0μM의 primer를 넣어 반응 당 10ul가 되도록 조성하였다. 조건은 역전사과정 50°C 1분을 진행하고, 초기변성 95°C 20초후, 변성 95°C 1초, 혼성 52°C 2초, 신장 72°C 2초를 1회전으로 하여 총 50회전의 초고속 PCR을 시행하였다.
이후 증폭이 확인되지 않은 1.0×101 분자와 1.0×100 분자의 증폭산물을 각각 1/1,000로 희석하여 초고속 nested PCR의 주형으로 사용하였으며, 2×RapiMix, 최종농도 4.0μM의 primer를 넣어 반응 당 10ul PCR 반응액이 되도록 조성하였다. 이때, 조건은 초기 변성 95°C 20초 후, 변성 95°C 2초, 혼성 48°C 2초, 신장 72°C 2초를 1회전으로 하여 총 50회전으로 nested 초고속 PCR을 수행하였다.
결과는 Ct값과 Tm (mid-point of melting temperature) 값의 비교 및 분석을 통하여 어느 수준의 ABPV가 감염되었으며, 바이러스 존재 입증 및 감염정도를 판단할 수 있는지를 측정, 계산하였다.
결과 및 고찰
ABPV 특이 유전자 인공합성
ABPV 특이 long-nucleotide들을 순차적으로 사용하여 최종적으로 226 bp의 ABPV RdRp 유전자의 합성여부를 확인하였다. 이때, F1/R1에 의한 증폭산물은 104 bp 크기를 가지고, 이를 주형으로 사용하여 F2/R2에 의해 신장시켰을 때는 181 bp, 최종적으로 F3/R3에 의해 신장됐을 때는 226 bp 크기를 가지는 것을 확인하였다(Fig. 2).
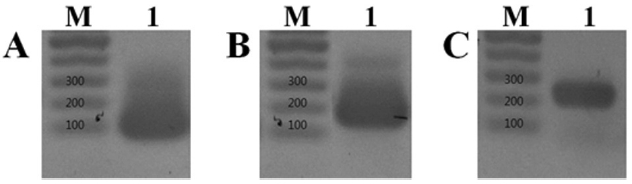
Artificially synthesized ABPV RdRp specific DNA. Using six ABPV-specific long-nucleotides sequentially, the artificial synthesis were successfully performed. Panel A, 104 bp long DNA fragment with long-nucleotide F1/R1; Panel B, 181 bp DNA with F2/R2; Panel C, 226 bp DNA with long-nucleotide F3/R3.
인공합성된 ABPV RdRp 유전자(ABPV-226)는 염기서열의 분석을 진행하였으며, 제한효소 인식부위 18 염기를 제외한 208 염기서열이 ABPV (Genbank Accession No. AF150639)의 RdRp 유전자 부위와 100% 일치함을 확인하였다. 이 208 염기서열상에서 ABPV 특이 검출용 primer 쌍과 ABPV 특이 nested primer 쌍의 위치를 제시하였다(Fig. 3).
ABPV 특이 detection 및 nested primer 쌍들의 최적 농도
초고속 PCR법에 적용시키기 위한 두 쌍의 ABPV 특이 primer의 최적 농도를 확립하기 위하여 500nM, 1.0μM, 2.0μM, 4μM의 최종농도로 조성하여 초고속 PCR을 진행하였다(Table 3).
최적의 primer 농도는 측정된 각 농도별 Ct값 및 최종형광값 그리고 melting graph에서 비특이적 증폭 양상을 보이지 않는 조건에서 결정하였다.
pABPV-RdRp226 1.0×108 분자를 주형으로 사용했을 때, 2쌍의 ABPV 특이 primer는 모두 4.0μM에서 가장 빠른 Ct값과 높은 최종형광값을 가지는 것을 확인할 수 있어 이를 두 ABPV 특이 primer 쌍의 최적조건으로 정립하였다.
ABPV 특이 detection 및 nested PCR의 최적 혼성온도
ABPV 특이 검출용 primer를 사용한 ABPV 특이 detection PCR은 40°C부터 60°C까지의 혼성온도를 사용한 온도구배 PCR을 수행하였다. 결과로, 실험된 전체 혼성온도에서 비교적 고른 증폭산물이 관찰되었으며, 특히 50~56°C의 범위에서 많은 PCR산물이 생산되었다(자료 미제시).
이 결과를 토대로 초고속 ABPV 특이 detection PCR은 혼성온도 50°C, 52°C, 54°C, 56°C에서 Ct값 및 최종 형광값, 정량성을 기준으로 최적의 혼성온도를 구하였다
ABPV 특이 detection PCR은 혼성온도가 증가함에 따라 Ct값도 증가하는 경향을 보였으며, 50°C에서 가장 빠른 Ct값을 가지는 것을 확인할 수 있었다. 그러나 검출한계 측면에서는 혼성온도 52°C에서 가장 우수했으며(1.0×102 분자까지 증폭), 그 외의 혼성온도는 비교적 낮은 검출한계를 가지는 것을 확인하였다. 최종형광값은 모든 혼성온도에서 큰 차이가 없음을 확인하였다. 따라서 52°C를 ABPV 특이 detection PCR의 최적 혼성온도로 결정하였다. 이때 증폭된 모든 산물의 Tm값은 78.89±0.24°C로 측정되었다(Fig. 4, Table S1).
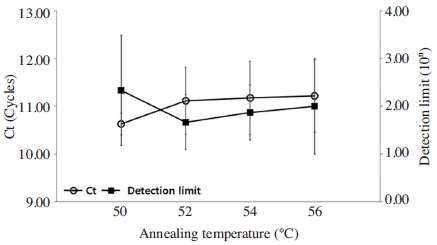
Optimization of annealing temperature in ABPV-specific detection PCR. Ultra-rapid ABPV-specific detection PCR was performed at each annealing temperature. The annealing temperature range of detection primer was set at 50°C, 52°C, 54°C and 56°C 1.0×108 molecules of pABPV-RdRp226 was used as template in ABPV-specific detection PCR. The fastest Ct value was found on detection PCR at 50°C, however, the detection limit of 52°C was more sensitive. Therefore, 52°C was determined as the optimal annealing temperature in ABPV-specific detection PCR.
ABPV 특이 nested primer를 사용한 ABPV 특이 nested PCR 또한 40°C부터 60°C까지의 혼성온도를 사용한 온도구배 PCR을 수행하였다. 이 또한 전체 온도에서 비교적 고른 증폭산물이 관찰되었으나, 특히 46~50°C의 온도 범위에서 보다 많은 PCR 산물이 생산되었다(자료 미제시).
이 결과를 토대로 초고속 ABPV 특이 nested PCR에서 혼성온도 46°C, 48°C, 50°C, 52°C에서 Ct값 및 최종 형광값, 정량성을 기준으로 최적의 혼성온도를 구하였다.
ABPV 특이 nested PCR은 혼성온도 48°C에서 가장 빠른 Ct값을 가지면서 동시에 우수한 검출한계(1.0×102 분자까지 증폭)를 가짐을 판정하였으며, 48°C를 ABPV 특이 nested PCR의 최적 혼성온도로 결정하였다. 이때 증폭된 모든 산물의 Tm값은 78.79±0.22°C로 측정되었다(Fig. 5, Table S2).
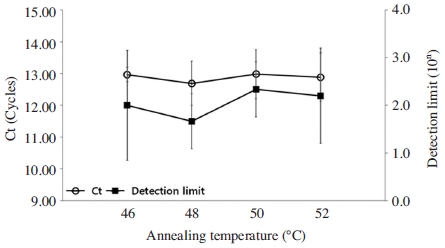
Optimization of annealing temperature at ABPV-specific nested PCR. Ultra-rapid ABPV-specific nested PCR using nested primer was performed at each annealing temperature (46°C, 48°C, 50°C and 52°C) with 1.0×108 molecules of pABPV-RdRp226. PCR at 48°C show fastest Ct value and more sensitive detection limit.
ABPV 특이 detection PCR은 primer 최적농도 각 4μM(최종농도), 혼성온도 52°C에서, 그리고 ABPV 특이 nested PCR은 primer 최적농도 4μM(최종농도), 혼성온도 48°C에서 빠른 Ct값과 높은 검출한계를 가지는 것을 확인함으로써, 각 primer 쌍에 의한 초고속 PCR에서 최적조건으로 사용하였다.
ABPV 특이 primer 쌍에 의한 최단 검출시간 및 검출한계 측정
두 쌍의 ABPV 특이 primer의 민감도를 평가하기 위하여 결정된 최적 혼성온도 및 primer 농도에서 재조합 DNA 주형을 1.0×108 분자부터 1.0×100 분자까지 연속 희석한 것을 초고속 PCR의 주형으로 사용하였으며, 이에 대한 검출한계 및 민감성을 측정하였다. 또한 검출시간의 단축을 위하여 PCR의 각 회전 당 변성, 혼성, 신장 단계를 각각 2초에서 1초로 단축시키며 실시하였으며, 신속하고 높은 검출한계를 가지는 조건을 확인하였다(Fig. 6, Fig. 7).
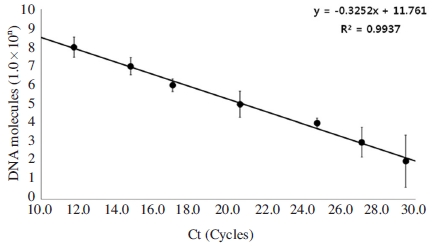
Detection limit in ABPV-specific detection PCR. Ultra-rapid ABPV-specific detection PCR using detection primer was performed at optimal condition. pABPV-RdRp226 was used as template in PCR that were diluted from 1.0×108 to 1.0× 100 molecules/PCR, respectively. It was calculated regression equation (y=-0.3252x+11.761) and regression coefficient (R2=0.9937).
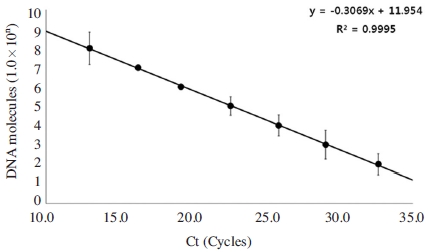
Detection limit at ABPV-specific nested PCR. Ultra-rapid ABPV-specific nested PCR using nested primer was performed at optimal condition. pABPV-RdRp226 was used as template in PCR that were diluted from 1.0×108 to 1.0× 100 molecules/PCR, respectively. It shows regression equation (y=-0.3069x+11.954) and regression coefficient (R2=0.9995).
ABPV 특이 detection primer를 사용한 초고속 PCR에서, 회전 당 각 단계는 1초, 2초, 2초의 시간조건에서 가장 빠른 Ct값과 우수한 검출한계를 가지는 것으로 측정되었으며, 이 는 1.0×102 분자 이상의 정량적 증폭이 가능함을 보여주었다. Ct값과 주형 분자 수를 사용한 회귀식은 y=-0.3252X+11.761로 계산되었으며, 이에 따른 회귀상수는 R2=0.9937로 나타나 높은 정량성을 보여주었다. Tm값은 78.80±0.31°C로 측정되었다(Table S3).
ABPV 특이 nested primer를 사용한 초고속 정량 PCR에서는 회전의 각 단계를 2초, 2초, 2초의 조건에서 수행했을 때 가장 빠른 Ct값과 높은 민감도를 갖는 것을 확인하였으며, 이 조건에서 1.0×102 분자 이상이 정량적으로 증폭이 가능함을 확인하였다. Ct값과 주형 분자 수를 사용한 회귀식은 y=-0.3069X+11.954로 계산되었으며, 이에 따른 회귀상수는 R2=0.9995로 나타나 높은 정량성을 보여주었다. Tm값은 79.98±0.21°C로 측정되었다(Table S3).
ABPV 특이 Nested PCR의 민감성
ABPV 특이 detection primer를 사용한 1차 PCR은 1.0×108 분자부터 1.0×102 분자까지 정량적으로 증폭됨을 Ct값과 Tm값을 통하여 확인하였으며, 1.5% agarose gel에서 특이적인 밴드를 가지는 것을 확인하였다(Fig. 8).
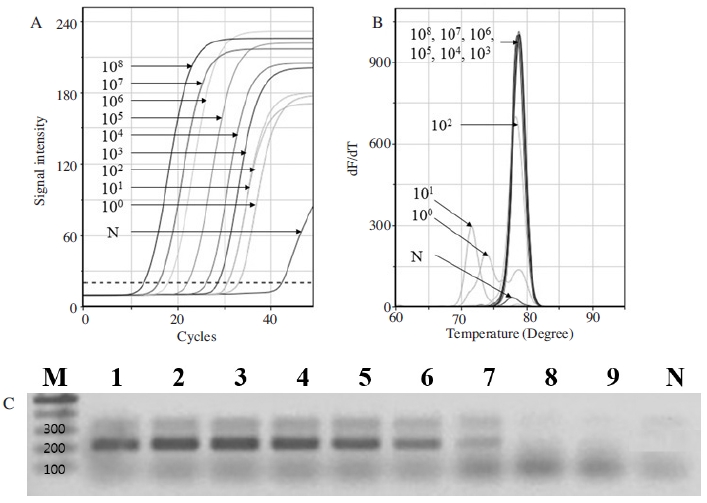
Quantitative analysis using ABPV-specific detection UR-PCR. Ultra-rapid ABPV-specific detection PCR using detection primer was performed at optimal condition. pABPV-RdRp226 was used as template in PCR that were diluted from 1.0×108 to 1.0×100 molecules/PCR, respectively. Panel A, amplification curve that can be confirmed that 1.0×108 to 1.0×102 molecules are quantitatively detected. Panel B, melting curve. Tm values of PCR products using 1.0×108 to 1.0×102 molecules were identical value, however, Tms using 1.0×101 and 100 molecules were differently estimated. Panel C, 1.5% agarose gel. Lane 1 to 9 are PCR products using 1.0×108 to 1.0×100 molecules, respectively. 208 bp long PCR product was not seen on lane 8 (1.0×101) and 9 (1.0×100) only.
또한, ABPV 특이 nested primer를 사용한 2차 PCR에서도 1.0×108 분자부터 1.0×102 분자까지 정량적으로 증폭되는 것을 Ct값과 Tm값을 통하여 확인하였으며, agarose gel에서도 특이적인 밴드를 가지는 것을 확인하였다(결과 미제시).
ABPV 특이 detection PCR에서, 그리고 ABPV 특이 nested primer를 사용한 2차 PCR에서도 1.0×102 분자 이상의 ABPV 특이 초기 주형에 대하여 특이 증폭이 확인되었으나, 1.0×101 분자와 1.0×100 분자의 초기 주형에 대하여 Ct값은 측정되지만, Tm값에서 각 7.33°C, 5.04°C의 차이를 보여 특이 증폭이 아닌 것으로 판정할 수 밖에 없었고, agarose gel의 크기 분석에서도 특이적인 208 bp의 PCR산물을 관찰할 수 없었다.
초기주형 1.0×101 분자 및 1.0×100 분자를 사용한 ABPV 특이 detection PCR의 증폭산물들은, 극미량의 주형이 1차 PCR에 의해 증폭은 되었으나, 증폭된 PCR산물이 너무 미량이라, 형광 또는 염색으로 측정되지 못하였다는 가정 하에 상기 증폭산물들을 1/10부터 1/108까지 희석하여 각기 ABPV 특이 nested PCR의 주형으로 사용하는 재 증폭을 추구하게 되었다.
결과로써, ABPV 특이 detection PCR에서 특이 증폭이 확인되지 않았던 초기 주형 1.0×101 분자에 대한 PCR은, 그 PCR 산물을 희석하여 재차 주형으로 사용한 ABPV 특이 nested PCR에서 ABPV 특이 증폭이 분명히 확인되었음은 물론, ABPV 특이 nested PCR은 ABPV 특이 1.0×101 분자를 1012 분자이상으로 증폭한 것으로 측정되었다(Fig. 9, Table S4).
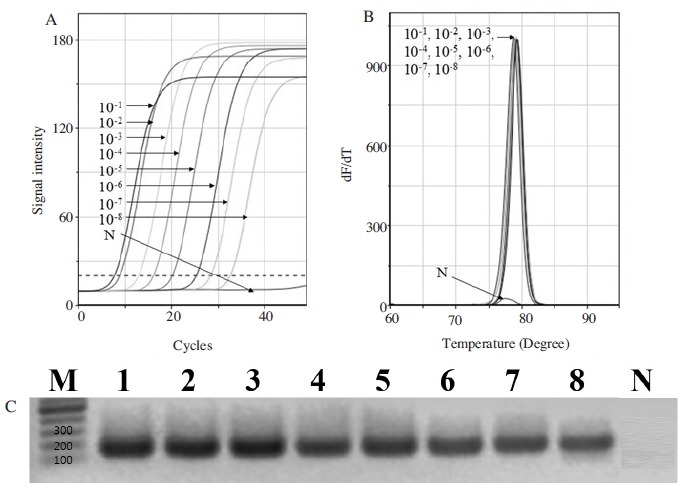
ABPV-specific nested PCRs of diluted PCR products using ABPV-specific detection PCR with 101 molecules as a initial template. Ultra-rapid ABPV-specific nested PCR was performed at optimal condition with diluted primary PCR products. Panel A, amplification curve according each diluted rate. Panel B, Tm values were identical except negative control. Panel C, 1.5% agarose electrophoresis. Lane 1 to 8 are PCR products of 10-1 to 10-8 using ABPV-specific nested primer, respectively.
또한 결과로써, ABPV 특이 detection PCR에서 특이 증폭이 확인되지 않았던 초기 주형 1.0×100 분자에 대한 PCR은 그 PCR 산물을 희석하여 재차 주형으로 사용한 ABPV 특이 nested PCR에서 ABPV 특이 증폭이 또한 분명히 확인되었음은 물론, ABPV 특이 nested PCR은 ABPV 특이 1.0×100 분자를 1011 분자이상으로 증폭한 것으로 측정되었다(Fig. 10, Table S4).
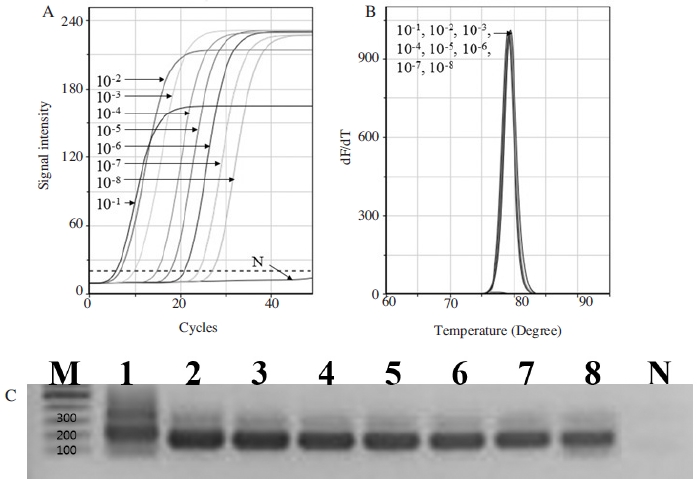
ABPV-specific nested PCRs of diluted PCR products using ABPV-specific detection PCR with 100 molecules as a initial template. Ultra-rapid ABPV-specific nested PCR was performed at optimal condition with diluted primary PCR products. Panel A, amplification curve according each diluted rate. Panel B, Tm values were identical except negative control. Panel C, 1.5% agarose electrophoresis. Lane 1 to 8 are PCR products of 10-1 to 10-8 using ABPV-specific nested primer, respectively.
ABPV 특이 detection PCR에서 특이 증폭이 인정되지 않았던 수십(101) 또는 수(100) 분자에 대한 검출은 이 PCR 증폭산물을 사용한 2차, 즉 ABPV 특이 nested PCR에 의해 ABPV 특이 증폭이 인정되었다. 이는 수 개의 분자뿐이어도 ABPV의 존재를 확인할 수 있음을 의미한다.
국내 꿀벌 시료로부터 ABPV 특이 유전자의 검출
국내 8개 지역(수원, 용인 2곳, 강릉, 의성, 여수, 울산, 제주)에서 수집된 ABPV 감염 의심 꿀벌시료로부터 total RNA를 추출하여 ABPV 특이 detection PCR의 주형으로 사용하였다.
결과로써, ABPV 특이 detection PCR에서 용인(Y1, Y2), 강릉(G), 여수(YS), 울산(U)의 5개 시료에서 PCR 산물의 증폭을 관찰되었으나, 용인1(Y1)과 강릉(G) 시료에서만 표준 DNA (pABPV-RdRp226)와 유사한 Tm값을 보였으며, 전기영동에 의한 PCR 산물의 크기분석에서 Y1의 PCR 산물만이 표준 DNA의 PCR 산물(208 bp)과 유사한 크기를 보였다.
한편, ABPV 특이 detection PCR 결과에서 ABPV 특이 유전자가 증폭되었다고 판단된 Y1과 G의 Ct값을 회귀식 y=-0.3252x+11.761 (Fig. 6)에 대입하였으며, 용인1 (Y1, 1/100)의 ABPV 유전자 양은 7.8×102 분자, 강릉(G)은 4.1×101 분자로 계산되었다(Table 4).
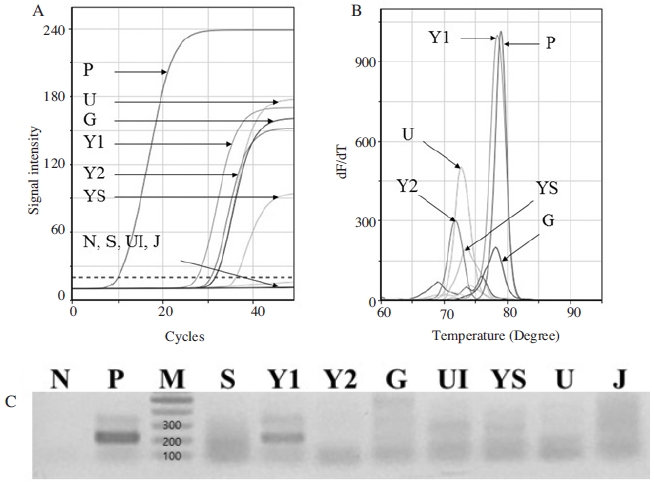
ABPV-detection using ABPV-specific detection PCR with 8 samples. RNA extracted from each honeybee sample was used as initial template, and performed by ABPV-specific detection PCR. Samples were collected from S (Suwon), Y1 (Yongin1), Y2 (Yongin2), G (Gangneung), UI (Uiseong), U (Ulsan) and J (Jeju). ABPV-specific gene was only detected from Y1. Panel A, amplification curves. Panel B, Tm values of Y1 and G were similar to Positive (79.15°C). Panel C, Only Y1 amplified PCR products was identical size with Positive (208 bp).
ABPV 특이 detection PCR에 의한 결과로써, 국내 8개 꿀벌 시료에 대한 검사는 단지 용인1 시료만 ABPV 양성으로, 강릉의 경우 의양성으로, 나머지 6개 시료는 음성으로 판단되었다.
한편, 본 연구의 결과(Fig. 9, 10)는 초미세량으로 존재하는 ABPV에 대하여 과연 ABPV 특이 detection PCR에 의한 1회의 PCR만으로 검출한계가 충분한가 하는 의심이 있었으며, 이에 ABPV 특이 detection PCR에 의한 PCR증폭산물들을, 각기 1,000배씩 희석하여 ABPV 특이 nested PCR의 초기 주형으로 사용하여 2차 PCR을 수행하였다.
결과로, ABPV-specific detection PCR 결과에서 ABPV 특이 유전자가 검출된 것으로 판정된 Y1은 ABPV-specific nested PCR에서 Tm값(79.15°C)이 표준 DNA의 PCR 산물의 Tm값(79.15°C)과 동일함이 재차 확인되었을 뿐만 아니라, 1차 PCR (ABPV-specific detection PCR)에서 의양성이라 판정하였던 강릉(G)과 음성으로 판정하였던 수원(S), 의성(UI), 울산(U), 제주(J)에서 ABPV양성의 결과를 보여 주었으며, 이는 PCR의 정규 증폭, Tm값 일치성, 전기영동에 의한 증폭산물 크기 비교(166 bp)의 결과로 판정된 것이다(Fig. 12).
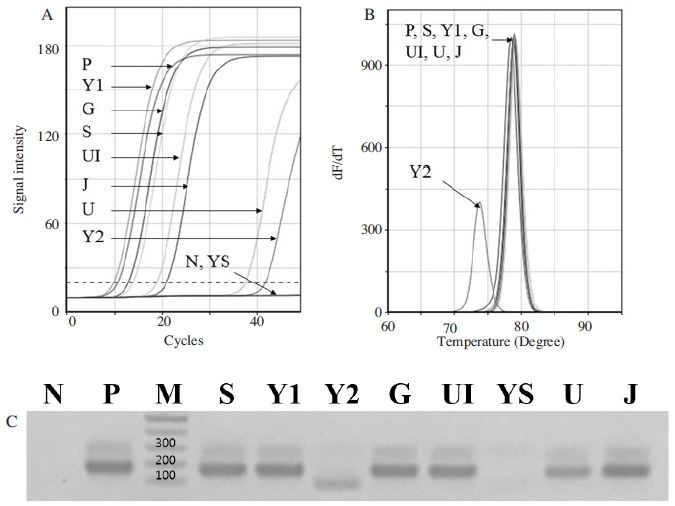
Secondary ABPV-detection using ABPV-specific nested PCR with 8 samples. 1/1,000 diluted PCR products amplified by ABPV-specific detection PCR was used as an initial template, respectively, for ABPV-specific nested PCR. Panel A, amplifications curves. Panel B, all Tms except Y2 and YS are identical with positive one. Panel C, all amplified PCR products except Y2 and YS are identical size with positive one. ABPV-specific gene was detected from lane S, Y1, G, UI, U and J.
본 실험의 결과는 ABPV 특이 detection PCR에 의한 한번의 PCR 결과만으로 시료 중 극미량 ABPV의 존재 여부를 판단하는 것은 부족할 수 있으며, 극미량 존재하는 ABPV의 경우에는 ABPV 특이 nested PCR과 같은 2차 PCR의 수행이 보다 정확한 판정을 하는데 요긴한 것임을 보여주고 있다.
따라서 상기 8개 시료에 대한 최종 결과는 6개 시료(용인1, 강릉, 수원, 의성, 울산, 제주)에 대하여, 각기 양적인 차이는 있으나, ABPV 양성으로, 2개 시료(용인2, 여수)는 음성으로 판정되었다.
결 론
ABPV의 신속하고 정확한 검출을 위하여 ABPV 특이 detection PCR과 ABPV 특이 nested PCR법을 고안하였다.
1회의 PCR로 수행되는 ABPV 특이 detection PCR은 100 분자 이상의 ABPV 특이 초기 주형이 있을 경우, 우수한 정확성과 신속성을 보였으나, 그 이하의 수십 혹은 수 분자의 ABPV 특이 주형이 있을 경우는 ABPV가 존재함을 보여주지 못하였다. 이런 검출한계는 환경 시료로부터 해당 바이러스를 모니터링하는데 한계를 보일 수 있을 뿐 아니라, 병원성 바이러스의 검출여부(존재 확인)로 질병을 진단하는 유전자 진단에서 오진을 일으키게 된다.
본 연구에서 제안하는 ABPV 특이 nested PCR법은 ABPV 특이 detection PCR (1차 PCR)의 PCR산물을 초기 주형으로 하여 재차 PCR하는 것으로, 수개 분자 이하의 극미량 ABPV라도 검출(존재 확인)할 수 있음을 입증할 수 있었다.
실례로써, 전국적으로 수집된 8개 ABPV 의심 꿀벌시료에서 ABPV 특이 detection PCR법으로 단 1개 시료(Y1)만 ABPV의 존재를 확인할 수 있었으며, 나머지 7개에 대하여는 ABPV 의심 또는 불검출로 판정할 수 있었다. 그러나 ABPV 특이 detection PCR 산물을 희석하여, ABPV 특이 nested PCR법을 적용한 결과, Y1시료를 포함한 6개(Y1, S, G, UI, U, J)시료에서 ABPV 존재를 확인할 수 있었으며, 나머지 2개 시료(Y2, YS)에 대하여는 ABPV 불검출로 판정할 수 있었다.
본 연구는 수 개의 분자와 같이 극미량 바이러스도 검출 가능함을 보여 주었을 뿐 아니라, ABPV 특이 nested PCR로 ABPV 특이 유전자 증폭을 재차 확인함으로 ABPV를 근연종인 KBV나 IAPV와 정확히 구분 할 수 있는 방법이 될 수 있을 것이다.
본 연구에서 고안된 ABPV 특이 detection PCR과 ABPV 특이 nested PCR법은 ABPV에 국한된 것이 아니며, 극미량으로 존재하는 다른 바이러스들의 다양한 연구에 확장 적용되기를 기대하는 바이다.
Acknowledgments
본 연구는 2018학년도 경기대학교 대학원 연구원 장학생 장학금 지원과 2018학년도 농촌진흥청 연구사업(세부과제번호: PJ01408002) 지원에 의하여 수행되었으며, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(11510203)과 중소기업청에서 지원하는 2017년도 산학연협력 기술개발사업(C0563751)의 연구수행으로 인한 결과물임을 밝힙니다.
References
-
민상현, 왕지희, 임수진, 이칠우, 윤병수, (2016), 꿀벌 병원성 바이러스의 정량적 검출을 위한 정량 실시간 재조합효소-중합효소 증폭법의 개발, Journal of Apiculture, 31(2), p147-156.
[https://doi.org/10.17519/apiculture.2016.06.31.2.147]

-
왕지희, 이도부, 구수진, 백문철, 민상현, 임수진, 이칠우, 윤병수, (2016), 다중 PCR증폭과 특이 DNA-chip을 활용한 꿀벌 주요 11종 병원체의 검출법 개발, Journal of Apiculture, 31(2), p133-146.
[https://doi.org/10.17519/apiculture.2016.06.31.2.133]

- 유미선, 이동우, 김일욱, 김동수, 권순환, 임윤규, 윤병수, (2007), 국내 양봉장에서 Kashmir bee virus(KBV)의 검출과 이의 분자생물학적 고찰, Journal of Apiculture, 22(1), p33-42.
- 이중구, 이보람, 윤병수, (2012), Loop-mediated isothermal amplification(LAMP) 법을 이용한 Kakugo virus의 새로운 검출법 개발, Journal of Apiculture, 27(4), p267-274.
- 이혜민, 이도부, 한상훈, 이명렬, 임윤규, 윤병수, (2005), 한국산 꿀벌에서 Deformed wing virus의 발견과 real-time PCR 검출법의 원리, Journal of apiculture, 20(2), p85-94.
- 최용수, 이명렬, 이만영, (2007), 국내 꿀벌에서의 Israle acute paralysis virus(IAPV) 진단, Journal of apiculture, 22(2), p159-165.
-
Bailey, L., Gilbbs, A. J., Woods, R.D., (1963), Two viruses from adult honey bees (Apis mellifera Linnaeus), Journal of virology, 21(3), p390-395.
[https://doi.org/10.1016/0042-6822(63)90200-9]

-
Bakony, Tamas, Farkas Robert, Szendroi Amdrea, Dobos-Kovacs Mihaly, and Rusvai Mikols, (2002), Detection of acute bee paralysis virus by RT-PCR in honey bee and Varroa destructor field samples: rapid screening of representative Hungarian apiaries, Apidologie, 33, p63-74.
[https://doi.org/10.1051/apido:2001004]

- Carpana, E., Vecchi, M. A., Lavazza, A., Bassi, S., Dottori, M., and Ritter, W., (1991), Prevalence of acute paralysis virus (APV) and other viral infections in honeybees in Italy, Proc. Int. Symp. on Bee Pathology, Ghent, Belgium, p155-165.
- Chen, Yan Ping, and Siede Reinhold, Honey bee virus, (2007), Advances in virus research, 70, p33-80.
-
de Miranda, Joachim R., Cordoni Guido, and Budge Giles, (2010), The acute bee paralysis virus-Kashmir bee virus-Israeli acute paralysis virus complex, Journal of Invertebrate Pathology, 103, pS30-S47.
[https://doi.org/10.1016/j.jip.2009.06.014]

-
de Miranda, Joachim R., Dainat, Bejamin, Locke, Barbara, Cordoni, Guido, Berthoud, Helene, Gauthier, Laurent, Neumann, Peter, Budge, Giles E., Ball, Brenda V., and Stoltz , Don B., (2010), Genetic characterization of slow bee paralysis virus of the honeybee (Apis mellifera L.), Journal of General Virology, 91, p2524-2530.
[https://doi.org/10.1099/vir.0.022434-0]

- Faucon, J. P., Vitu, C., Russo, P., and Vignoni, M., (1992), Diagnosis of acute paralysis: application to the epidemiology of honey bee viral diseases in France in 1990, Apidologie, 23, p139-146.
-
Hung, A.C.F., and Shimanuki, H., (1999), A scientific note on the detection of Kashmir bee virus in individual honeybees and Varroa jacobsoni mites, Apidologie, 30, p353-354.
[https://doi.org/10.1051/apido:19990414]

-
McMahon, Dino P., Wilfert, Lena, Paxton, Robert J., and Brown, Mark J. F., (2018), Emerging viruses in bees: From molecules to ecology, Advances in virus research, 101, p251-291.
[https://doi.org/10.1016/bs.aivir.2018.02.008]

Appendix
SUPPLEMENTARY DATA

Ct, Tm, final fluorescence and detection limit of ABPV-specific detection PCRs at each annealing temperature

Ct, Tm and quantitative analysis of ABPV-specific nested PCR with diluted PCR products of ABPV-specific detection PCR with 101 or 100 molecules as initial template
