
사탕수수 설탕 및 사양꿀에서 사탕수수 (Saccharum officinarum) 고유 유전자의 검출
Abstract
Sugar cane-specific gene could be successfully amplified with DNAs isolated from sugar or sugar-honey using Saccharum officinarum-specific Ultra-Rapid or conventional PCR. Specificity of PCR products from sugar or sugar-honey was verified by nested PCR and DNA sequencing. This PCR could be applied to a quantitative analysis for honey-evaluation. In our knowledge, it is first report that sugar cane-specific sequence could be detected from sugar-honey or sugar itself, and that sugar-honey could be evaluated by genetic examination.
Keywords:
Honey, Saccharum officinarum, Sugar cane, Sugar-honey, Sugar, Ultra-rapid PCR서 론
벌꿀에 대한 국제 규격인 Codex 및 EU규정 등에서 벌꿀은 ‘꿀벌들이꽃꿀, 수액 등 자연물을 채집하여 벌집에 저장한 벌집꿀과 이것에서 채밀한 벌꿀’로 정의하고 있으며(AOAC, 1995; EU Commission, 2015), 다른 물질이 첨가되지 않은 순수한 것을 말한다.
꿀벌에게 설탕을 먹여 제조한 사양꿀은 천연꿀과 구별이 쉽지 않고, 생산비용은 상대적으로 매우 저렴하여 상품으로써 천연꿀의 존립을 크게 위태롭게 하고 있을 뿐 아니라, 벌꿀 산업 전체에 대한 신뢰를 크게 떨어뜨리고 있다(Yoo et al., 2010).
우리나라 식품공전에서는 천연꿀 뿐 아니라 설탕물을 주어 꿀벌이 수집한 사양꿀까지 벌꿀류에 포함하여 설정하고 있으며(MFDS, 2014), 이는 벌꿀 구성성분의 이화학적 특성에 근거한 평가를 주로 하여 순수 벌꿀 여부의 확인에 어려움을 주고 있다. 사양꿀은 영양적인 면이나 가격적 면에서 천연꿀과 크게 차이나지만 외관상 구분이 잘 되지 않기에 분별할 수 있는 방법이 필요한 실정이다(Kim et al., 2017).
국내에서도 사탕수수 설탕을 꿀벌에게 먹여 제조된 사양꿀이 공식적으로 판매되고 있으며, 사양꿀은 탄소동위원소비를 측정하는 EA-IRMS (Elemental Analysis-Isotope Ratio Mass Spectrometry)법에 의해 천연꿀과 쉽게 구분될 수 있으나(Padovan et al., 2003), 사양꿀과 천연꿀이 혼합된 불량꿀의 경우, 계속적인 논란을 야기하고 있다. 또한 탄소동위원소비 분석법은 고가의 기기가 요구되는 점, 검출에 걸리는 시간, 그리고 사탕무 같은 C4 식물군의 당류 구분이 어렵다는 점 등의 문제점이 있어, 사양꿀의 판별에 보다 확실한 대안이 요구되고 있다(Kim et al., 2017).
근래까지, 고도로 정제된 백색 설탕에 잔류된 사탕수수 유전자는 있을 수 없으며, 더욱이 이 설탕물로 제조한 사양꿀에는 사탕수수 유전자가 잔류될 수 없다는 통설은, 사양꿀에 대한 유전자검사를 시도 조차 어렵게 하여왔다.
일반적으로 설탕의 정제는 1ppb (part per billion) 수준으로 수행되고 있으며(CODEX, 2001), ppb의 수준이란 설탕 1gram 당 1ng의 양을 의미한다. 한편, 1ng의 DNA는 3kb DNA가 108개(분자)가 존재하는 양으로, 현재 진보된 PCR 기술로는 101 분자의 DNA도 정량 검출할 수 있기에(김 등, 2017), 초극미량의 사탕수수 유전자가 설탕에 잔류된다면 이를 특이 PCR로 증폭하는 것은 능히 가능하리라 사료할 수 있었다.
한편, 당밀을 꿀벌의 사료로 사용한 생산된 벌꿀에서 당밀 고유 유전자를 PCR로 증폭시켜 이를 입증한 바 있었으며(Sobrino-Gregorio et al., 2018), 설탕처럼 고도의 정제를 거치지 않았다 하더라도 밀원의 유전자가 생산된 벌꿀에서 검출되는 것은, 화분의 혼입을 제외하고도 충분히 가능함을 보여 주었다(Laube et al., 2010).
사양꿀의 제조에서 가장 쉽게 사용되는 당원은 사탕수수에서 생산된 설탕이라 할 것이다.벌꿀에서 사탕수수 고유의 유전자 발견된다면, 이는 부주의하게 월동사양시킨 사양꿀이 천연벌꿀과 혼합되는 경우와 의도적으로 설탕물을 사양시킨 사양꿀이라 할 것이다.
본 연구는 고도로 정제된 설탕과 그 설탕으로 만들어진 사양꿀에 잔류할 수 있는 초미량의 사탕수수 고유 유전자의 존재를 입증하고, 더 나아가, 이 고유 유전자를 이용한 유전자 검사법을 새로운 사양꿀판별법으로 제시하고자 수행하였다.
목적 유전자로는 사탕수수(Sugar Cane, Saccharum officinarum)의 엽록체에 위치한 maturase K (matK, Genbank accession No. LN849913) 유전자를 사용하였다. 사탕수수 고유의 유전자(matK)는 사탕수수의 씨(재래종 Sugar cane seeds; 판매처, 가람원; 2018.6)로부터 확보하였다. 사탕수수 씨에서 추출한 DNA로부터 matK 유전자를 증폭시켰으며, 이 사탕수수 고유의 유전자를 탑재한 재조합 DNA pCane-Cp-mat를 제작하여 염기서열을 결정한 후, 사탕수수 고유의 분자 표준물질로 사용하였다(Table 1).
본 연구에서 사용된 설탕 시료의 DNA는, 시판되고 있는 사탕수수 설탕(백설 하얀설탕; 원당 100%; 가는 백설탕; 2018. 4. 13 제조)으로 제조한 80%(w/v) 용액에서, CTAB (hexadecyltrimethyl ammonium bromide)법 및 DNA-binding column (Qiagen, Germany)을 연결시켜 추출하였다. 당액 10ml에서 20μl의 DNA용액을 얻었으며, 이중 1μl를 PCR에 주형으로 사용하였다. PCR 반응액은 1μl primer 용액(각 10 pmole), 1μl DNA 주형, 5μl 2x Rapi:Detect Master mix (Genesystem Co., Ltd., Korea), 2μl 증류수를 각각 혼합하여, 총 10μl/reaction로 조성하였다. 초고속 PCR은 초기변성 95°C 30초 이후에, 변성 95°C, 3초 - 혼성 55°C, 3초 - 중합 72°C, 3초를 1 cycle로 하여, 총 50 cycle을 GENECHECKERTM(Genesystem Co., Ltd., Korea)를 사용하여 수행하였다.
결과적으로, 설탕에 잔류된 사탕수수 고유 유전자는 본 초고속 PCR에서 성공적으로 증폭시킬 수 있었으며, 이는 사탕수수 고유의 유전자를 탑재한 재조합 DNA pCane-Cp-mat로 증폭된 PCR산물과 Tm값, PCR산물의 크기에서 일치하여, 동일한 DNA서열이 증폭된 것으로 판정되었다(Fig. 1).
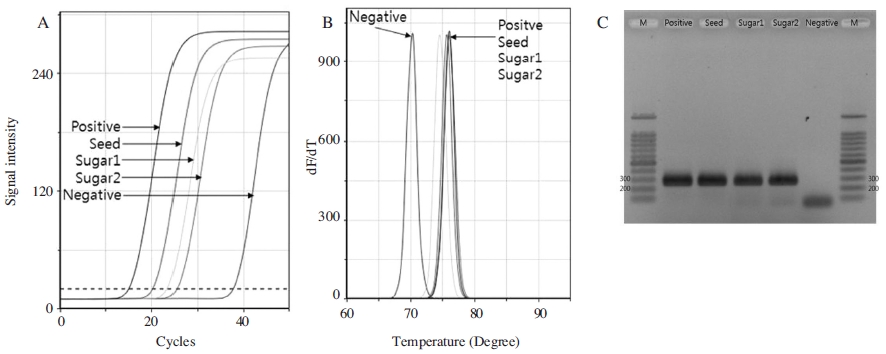
Amplification of S. officinarum-specific matK gene with DNA from sugar. Panel A. Fluorescence graph of S. officinarum-specific PCRs with DNAs from sugar cane seed (Seed), pCane-Cp-mat (Positive), and samples 1, 2 (Sugar 1, 2) of sugar using primer, Cane-cp-dF/dR. Panel B. Melting temperature analysis. Tms of PCR products from sugar are estimated on 74.58 and 75.88°C, respectively. Panel C. Agarose gel electrophoresis of PCR products. Lane M is DNA size marker. Expected size of S. officinarum-specific PCR product is 271 bp-long.
사양꿀 시료로는 시판되고 있는 사양꿀(종근당건강, 사양벌꿀 90%, 잡화꿀 10%, 탄소동위원소 비 -12‰)를 선택하였으며, 꿀 10ml를 사용하여 DNA의 추출, PCR의 조건 및 조성은 설탕시료의 경우와 동일하게 하였으며, 재조합 DNA pCane-Cp-mat을 사탕수수의 분자적 표준물질로 사용하였다.
사양꿀에 잔류된 사탕수수 고유의 유전자도 본 초고속 PCR방법을 사용하여 성공적으로 증폭을 시킬 수 있었다. 이는 사탕수수 고유의 유전자를 탑재한 재조합 DNA pCane-Cp-mat로 증폭된 PCR산물과 Tm값 비교와 PCR 산물의 크기에서 일치하여, 동일한 DNA 서열이 증폭된 것으로 판정되었다(Fig. 2).
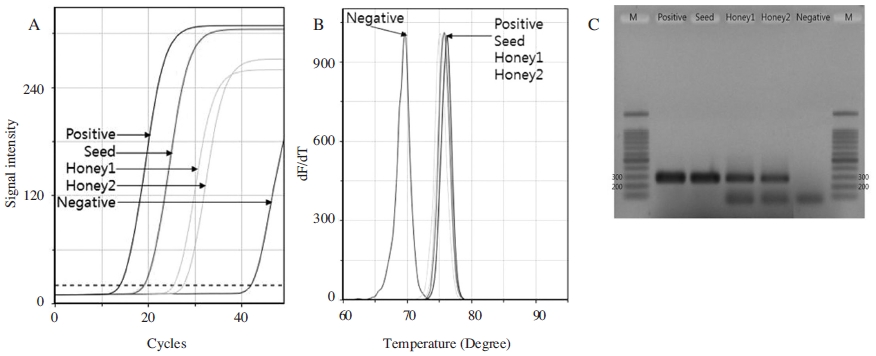
Amplification of S. officinarum-specific matK gene with DNA from sugar-honey. Panel A. Fluorescence graph of S. officinarum-specific PCRs with DNAs from sugar cane seed (Seed), pCane-Cp-mat (Positive), and samples 1, 2 (Honey 1, 2) of sugar-honey using primer, Cane-cp-dF/dR. Panel B. Melting temperature analysis. Tms of PCR products from sugar-honey are estimated on 75.23 and 75.56°C, respectively. Panel C. Agarose gel electrophoresis of PCR products. Lane M is DNA size marker. Expected size of S. officinarum-specific PCR product is 271 bp-long.
사양꿀 및 설탕에서 증폭된 PCR산물이 사탕수수 고유의 matK 염기서열인지 재확인하기 위하여 nested primer를 사용한 재 PCR 증폭을 수행하였다. 즉, detection primer를 사용한 1차 PCR 산물들을 각기 증류수로 1,000배 희석하여 nested PCR의 주형으로 사용하였으며, nested primer의 사용을 제외한 PCR 조성 및 조건은 1차 PCR과 동일하였다.
결과적으로, 사양꿀과 설탕물에서 추출된 DNA를 matK 특이 PCR로 증폭시킨 1차 PCR 산물들은 각기 nested PCR을 통해 정확히 증폭되었으며, 분자적 표준물질로 사용한 pCane-Cp-mat의 PCR산물과 Tm값 및 DNA 크기에서 일치됨을 보여주어, 생산된 PCR산물들은 모두 사탕수수 고유의 matK 유전자임이 확인되었다(Fig. 3).
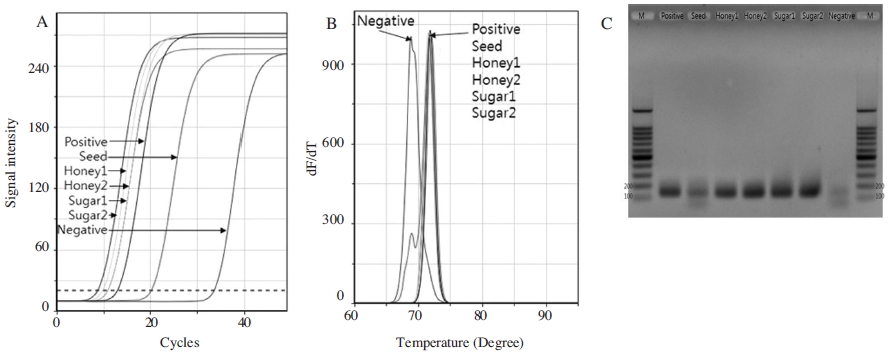
Amplification of S. officinarum-specific gene with DNA from sugar-honey and sugar by using nested PCR. Panel A. Fluorescence graph of S. officinarum-specific nested PCRs with DNAs from sugar cane seed (Seed), pCane-Cp-mat (Positive), and first PCR product of sugar-honey (Honey 1, 2) and sugar (Sugar 1, 2) using primer, Cane-cp-nF/nR. Panel B. Melting temperature analysis. Panel C. Agarose gel electrophoresis of nested PCR products. Lane M is DNA size marker. Expected size of S. officinarum-specific nested PCR product is 137 bp-long.
사양꿀 및 설탕에서 증폭된 PCR산물이 사탕수수 고유의 matK 염기서열을 가졌는지 보다 분명히 하기 위하여 각 PCR산물들에 대한 염기서열 결정을 수행하였다.
결정된 각 PCR산물들의 염기서열들은 정렬분석(alignment analysis)하였으며, 사탕수수, 사양꿀, 설탕, 분자적 표준물질(pCane-Cp-mat)에서 증폭된 모든 PCR산물은, 의심의 여지없이 모두 사탕수수 고유의 matK 염기서열로 판정되었다(Fig. 4).
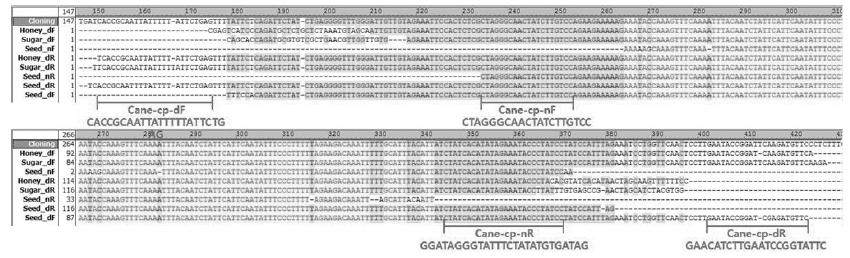
Alignment for sequences of PCR products from each sample. DNA sequences of the PCR products from seed, sugar-honey and sugar were aligned. All sequences were identical with matK (Genbank accession No. LN849913).
한편, 본 연구에서 사용된 초고속 PCR은 PCR을 보다 빠르게 수행하고자 하는 특수한 PCR방법이다. 그러므로 본 연구에서 제안하는 사탕수수 특이 matK PCR (S. officinarum-specific matK PCR)법이 현재 널리 사용되고 있는 일반 PCR기기에 바로 적용될 수 있는지 확인하고자, 일반 PCR기기를 사용한 사탕수수 특이 matK PCR을 수행하였다. 이를 위하여, 일반 PCR 기기인 Bioneer사의 Exicycler™ 96을 사용하였으며, PCR의 조성은 AccuPowerⓇ 2X GreenStar™ qPCR Master Mix (Bioneer, Korea)을 기본으로, 초고속 PCR의 경우와 동일한 primer들을 사용하였다(Table 1). 일반 PCR의 조건은 초기변성 95°C 5분 이후에, 변성 95°C, 30초 - 혼성 55°C, 30초 - 중합 72°C, 30초를 1cycle로, 총 40 cycle을 수행하였다.
결과로서, 사탕수수의 씨, 사양꿀, 설탕물에 추출된 DNA는 일반 PCR 조건에서도 성공적으로 증폭시킬 수 있었으며, 이는 사탕수수 고유의 유전자를 탑재한 재조합 DNA pCane-Cp-mat로 증폭된 PCR산물과 Tm값 및 PCR 산물의 크기에서 일치하여 동일한 DNA 서열이 증폭된 것으로 판정되었다. 이는 사탕수수 특이 matK PCR법이 어떤 기기를 사용하였을 때에도 설탕 및 사양꿀에 잔류하는 사탕수수의 고유 유전자를 증폭시킬 수 있음을 의미하는 것이다(Fig. 5).
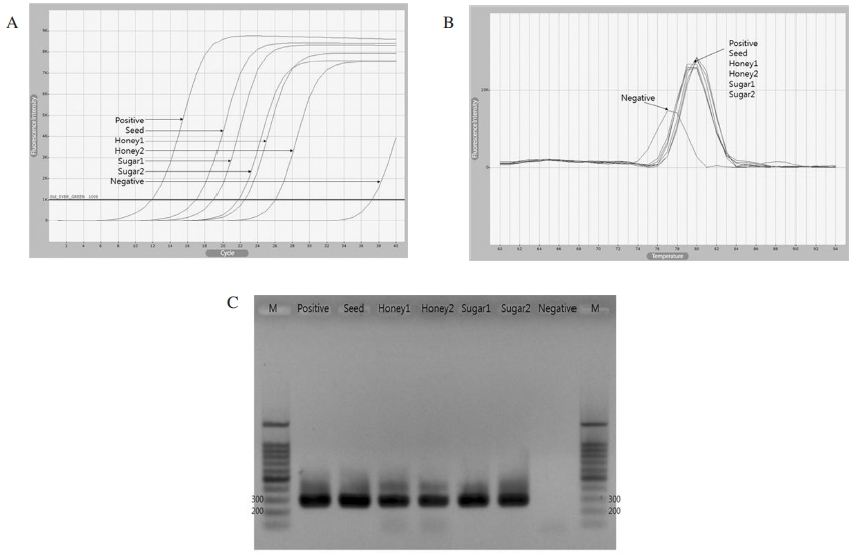
Amplification of S. officinarum-specific matK gene with DNA from seed, sugar-honey and sugar using conventional PCR. Panel A. Fluorescence graph of S. officinarum-specific PCRs with DNAs from sugar cane seed (Seed), pCane-Cp-mat (Positive), sugar-honey (Honey 1, 2) and sugar (Sugar 1, 2) using primer, Cane-cp-dF/dR. Panel B. Melting temperature analysis. Panel C. Agarose gel electrophoresis of PCR products. Lane M is DNA size marker. Expected size of S. officinarum-specific PCR product is 271 bp-long.
결론적으로, 설탕사양으로 생산한 사양꿀에 잔류하는 사탕수수 고유의 유전자를 PCR 방법으로 검출할 수 있다는 사실을 본 연구는 입증할 수 있었다. 이는 우리가 아는바 최초의 보고이며, 이 사실은 사탕수수 설탕사양꿀 뿐 아니라, 옥수수 시럽(Corn syrup)등을 사용한 사양꿀 등에서 해당 고유 유전자를 검출할 수 있는 연구들로 파생될 수 있을 것이며, 천연벌꿀이 아닌 사양의 형태로 제조되는 모든 사양꿀의 밀원을 쉽게 판별할 수 있는 유전자 검사법으로 범위를 확장시킬 수 있을 것이다. 본 연구의 결과가 천연벌꿀의 위치를 공고히 하는데 널리 사용될 수 있을 것으로 기대된다.
Acknowledgments
본 연구는 2018학년도 농촌진흥청 연구사업(세부과제번호: PJ01408002) 지원에 의하여 수행되었으며, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(115102-03)과 중소기업청에서 지원하는 2017년도 산학연협력 기술개발사업(No. C0563751)의 연구수행으로 인한 결과물임을 밝힙니다.
References
-
김정민, 임수진, Truong A Tai, 홍기정, 윤병수, (2017), 초고속 유전자 증폭법을 이용한 벌집꼬마밑빠진벌레(Aethina tumida)의 신속한 검출 기법 개발, J. Apiculture, 32, p119-131.
[https://doi.org/10.17519/apiculture.2017.06.32.2.119]

- AOAC, (1995), Official Method of Analysis of AOAC Intl, 20th ed., Method 998-12, Association of Official Analytical Chemists, Arlington, VA, USA.
- CODEX, (2001), CODEX STANDARD FOR SUGARS, CODEX STAN 212-1999.
- European Commission, (2015), Official controls and enforcement, Honey 2015-16, Brussels, Belgium.
-
Kim, S. G., I. P. Hong, S. O. Woo, H. R. Jang, J. S. Jang, and S. M. Han, (2017a), Chemical Composition of Korean Natural Honeys and Sugar Fed Honeys, The Korean Journal of Food and Nutrition, 30, p112-119.
[https://doi.org/10.9799/ksfan.2017.30.1.112 ]

-
Laube, I., H. Hird, P. Brodmann, S. Ullmann, M. Schöne-Michling, J. Chisholm, and H. Broll, (2010), Development of primer and probe sets for the detection of plant species in honey, Food Chemistry, 118, p979-986.
[https://doi.org/10.1016/j.foodchem.2008.09.063]

- Lim, H. Y., S. H. Han, and B. S. Yoon, (2014), Manual of Methods for Molecular Biology Ⅳ:, p95-97.
- MFDS, (2014), Food Standard Code, Ministry of Food and Drug Safety, Cheongju, Korea.
-
Padovan, G. J., D. De Jong, L. P. Rodrigues, and J. S. Marchini, (2003), Detection of adulteration of commercial honey samples by the 13C/12C isotopic ratio, Food Chemistry, 82, p633-636.
[https://doi.org/10.1016/s0308-8146(02)00504-6]

-
Sobrino-Gregorio, L., S. Vilanova, J. Prohens, and I. Escriche, (2018), Detection of honey adulteration by conventional and real-time PCR, Food Control, 95, p57-62.
[https://doi.org/10.1016/j.foodcont.2018.07.037]

- Yoo, E. C., Y. K. Kong, and B. S. Yoon, (2010), A Study on the Improved Analysis-methods to Determine Adulterated Honeys, Korean Journal of Apiculture, 25, p63-76.
