
국내 꿀벌에서 Lotmaria passim 검출
Abstract
Apis mellifera, also known as the western honey bee, is one of the most important pollinators of flowering plants, including many agricultural crops. In Korea, the number of honey bee pollinations have been fluctuating in recent years. Several honey bee parasites have been presumed as probable causes of decline. Lotmaria passim is the most virulent trypanosome parasite which usually infects the A. mellifera intestine. After first report in 1911, L. passim widely present in South America and Europe including Uruguay, Argentina, Chile and Spain. However, the presence of this virulent trypanosome is not reported in Korea before. The aim of the present work was to provide the evidence of detection of the L. passim from A. mellifera in Korean agriculture. A. mellifera from three sites; Gwangju, Yangpyung and Toechon was ground with liquid nitrogen and mRNA was extracted. Quantitative real-time PCR (qPCR) was used to investigate the presence of L. passim nucleic acid. The amplification products were also sequenced and analyzed. L. passim was detected from honey bee in Yangpyung. PCR products were sequenced and the nucleotide sequence was aligned to the complete L. passim sequence from NCBI database, and the sequence were revealed 100% identity. The honey bee parasitic trypanosome L. passim was found in the Korea honey bee first time and should be considered as a potential contributor of honey bee infectious disease in Korea.
Keywords:
Apis mellifera, Lotmaria passim, Trypanosome, Korean agriculture서 론
Apis mellifera 또는 A. mellifera라 불리는 이 꿀벌은 유럽, 아프리카, 중동 및 북아메리카를 비롯한 전 세계의 많은 지역에서 발견되는 꿀벌 종 중 하나이다 (Castelli et al., 2019). 이 A. mellifera는 사람들에게 꿀, 로열 젤리와 밀랍 등을 제공하는 것뿐만 아니라 꽃에 꽃가루를 전파하여 수분 활동을 돕는 것을 통해 국제경제 및 생태계에서 중요한 역할을 하고 있다 (Winter et al., 2006). 이 때문에, A. mellifera는 현대 농업에서 꽃가루 매개자로서 매우 중요한 역할을 맡고 있는 곤충이다. 하지만 A. mellifera는 최근 몇 십년간 전 세계적으로 위협적인 상황에 직면하고 있다. Varroa destructor 등의 여러 병해충, 농약 오염, 기후 변화 등의 요인으로 인해 꿀벌의 수가 감소하고 있다. 최근 일부 연구에서는 Trypanosome이 꿀벌의 주요 감염원 중 하나로 지목되고 있으며 그중 Lotmaria passim이 가장 많이 감염되었고 또한 병원성이 강하다고 밝혀지고 있다 (Runckel et al., 2011). Trypanosome은 원생생물로 동물, 곤충 심지어 식물까지 감염시킨다. 또한 이 원생생물은 벌목 (Hymenoptera)의 꿀벌이나 말벌도 감염시킬 수 있다 (Merzlyak et al., 2001). 이 중 꿀벌의 경우, 원생생물은 꿀벌의 소화관 내부에 번식하며 (Buendía-Abad et al., 2022), 꿀벌의 면역 체계를 약화시켜 꿀벌의 건강에 직·간접적인 영향을 미칠 수 있고 심할 경우 봉군붕괴현상 (CCD: Colony Collapse Disorder)을 유발하여 벌 군집에 막대한 피해를 줄 수도 있다 (Brown et al., 2003; Ravoet et al., 2013; Arismendi et al., 2022; Bartolomé et al., 2022). L. passim에 의한 감염 보고가 지속적으로 늘어나고 있는 추세이다 (Willians et al., 2019). 본 연구에서는, 한국의 세 지역 (양평, 퇴촌, 광주)에서 채집한 A. mellifera에서 원생생물인 L. passim이 검출되는지 확인하고자 하였다.
재료 및 방법
1. 꿀벌 시료 준비 및 Total RNA 추출
본 연구에서 원생생물 진단을 위하여 사용된 꿀벌 시료는 2022년, 국내 세 지역의 서로 다른 여섯 곳의 양봉농가에서 정상적인 꿀벌과 벌통 주변에서 폐사한 꿀벌을 별도로 채집하여 살아있는 상태를 유지한 후 한 마리의 성충 전부를 Total RNA의 분리에 사용하였으며, 실험에 사용된 시료들은 액체질소에 처리 후 -70℃에서 보관하였다. 각 sample의 Total RNA는 꿀벌 시료를 액체질소로 동결시킨 상태에서 mortar and pestle을 이용하여 파쇄한 후 0.75 mL의 Modified chloroform/phenol (TRIZOLTM, Invitrogen, Carlsbad, CA)과 Chloroform (Sigma-Aldrich, St. Louis, MO)을 0.2 mL 첨가하여 Cell lysis를 진행하였다. 이후, 15분간 12,000 g에서 원심분리를 진행하여 나온 Supernatant를 새 튜브에 옮긴 후 Isopropanol (Daejung, South Korea) 0.5 mL를 첨가하였다. 10분간 12,000 g에서 원심분리를 진행한 후, 원심분리 후 나온 상층액을 버리고, 침전물을 75% 에탄올 1 mL와 섞은 후 5분간 7,500 g에서 원심분리를 진행하였다. 에탄올을 완벽하게 제거한 후, 50 μL의 RNase-free water에 elution하였다.
2. cDNA 합성
추출한 Total RNA의 농도는 260 nm/280 nm 흡광도 값의 비율로 결정하였다. 분리한 Total RNA는 20 ng/μL로 Random hexamer와 HelixCriptTM Thermo Reverse Transcriptase kit (NanoHelix, South Korea)에 의한 역전사 효소반응 PCR (RT-PCR)을 통해 cDNA를 합성하였다.
3. Real-time PCR 분석
cDNA는 Gene-specific primer와 Control primer를 이용한 PCR 반응의 주형으로 사용하였다. 꿀벌에 감염된 L. passim 검출을 위한 Specific primer pairs는 Lp-gF/R, TrypanF1/R1 (Arismendi et al., 2016), qCrF/R (Revainera et al., 2020)과 CytbF/R (Ravoet et al., 2015)이 사용되었으며 Control primer로는 Am_RPL8F/R (Collins et al., 2004)을 이용하였다 (Table 1). PCR 증폭반응은 IncloneTM Excel Speed-Pfu polymerase kit (NanoHelix, South Korea)를 사용하였으며, Pre-denaturation 95℃ 10분, Denaturation 95℃ 30초, Annealing은 Primer set에 따라 (45℃/55℃/60℃) 30초, Polymerization 72℃ 1분으로 35 Cycle을 반복하고 72℃에서 5분간 Final extension을 수행하였다. PCR 산물은 0.7 μL/mL Ethidium bromide가 함유된 1% agarose gel로 전기영동 후, UV light transilluminator를 사용하여 확인하였다.
4. 계통도 분석
다른 지역의 L. passim과의 연관성을 추측하기 위해서, 증폭된 DNA를 HiGeneTM Gel&PCR Purification System (Biofact, South Korea)을 이용하여 정제하고 pGEM-T Easy vector (TA vector) (Promega, South Korea)에 Ligation한 후 DH5α에 transformation하였다. Plasmid를 추출하여 Sequencing을 진행하였다. Sample과 다른 지역의 L. passim Sequences의 유사도는 Basic Local Alignment Search Tool (BLAST, NCBI)을 통하여 확인하였다. 이후, 계통도는 MEGA X를 통해 Neighbor-joining method를 이용하여 만들었다 (Larkin et al., 2007; Yamamoto et al., 2023). Spain (Accession number MT5586091, KJ704266, MT559012, KJ704256, and MT558108), Uruguay (Accession number KX953207, KX953208, and KX953209), Chile (Accession number KT252557 and KT252561), Belgium (Accession number KM066212), USA (Accession number KJ713349), China (Accession number AB745489), Japan (Accession number AB716357) 그리고 Korea (Accession number OP913373)에서 보고된 L. passim에 대하여 계통도를 작성하였다. 다른 지역의 L. passim과의 연관성을 추측하기 위해서,
결과 및 고찰
각각의 세 지역에서 선택한 총 여섯 곳의 농가의 samples에 대하여 L. passim-specific primers를 이용하여 Real-time PCR을 진행한 결과 mRNA expression fold change의 값이 양평의 sample에서 높게 나오는 것을 확인할 수 있었다 (Fig. 1). L. passim-specific primers의 특이적 결합성의 입증을 위해 다양한 primer를 사용한 후 1% agarose gel에 PCR primer를 TAE buffer에 30분간 전기영동하여 나온 amplicon을 sequence하여 확인하였고 그 결과 Lp-gF/R이 L. passim에 특이적으로 결합하는 것을 확인하였다. 이후 양평 1의 Sample (GAPDH, OP913373)을 Lp-gF/R primer pairs를 이용하여 PCR로 DNA를 증폭시킨 결과 계통도에서 벨기에의 Trypanosome L. passim strain (KM066212)은 양평 1의 GAPDH와 100% 일치하는 것을 확인할 수 있었다 (Fig. 2). 따라서, L. passim이 현재 국내의 꿀벌들에게 감염되고 있다는 것을 확인할 수 있다. 또한 현재 L. passim은 일본 및 동남아시아에서도 감염이 확인되고 있는 상황이기 때문에 국내의 꿀벌에게도 충분히 발견될 수 있는 상황이다. 최근 연구에서, 겨울철의 꿀벌 관련 생산량이 매년 지속적으로 줄고 있음을 알 수 있다 (Jung and Cho, 2015). 그러나, 겨울철의 꿀벌 개체수의 지속적인 감소에 대한 명확한 원인은 아직 밝혀지지 않았다. 그러므로 새로운 연구를 통해 L. passim이 꿀벌 군집에 미치는 영향 혹은 다른 미생물이나 바이러스와의 상호작용을 통해 미치는 영향을 확인할 필요성이 있다.
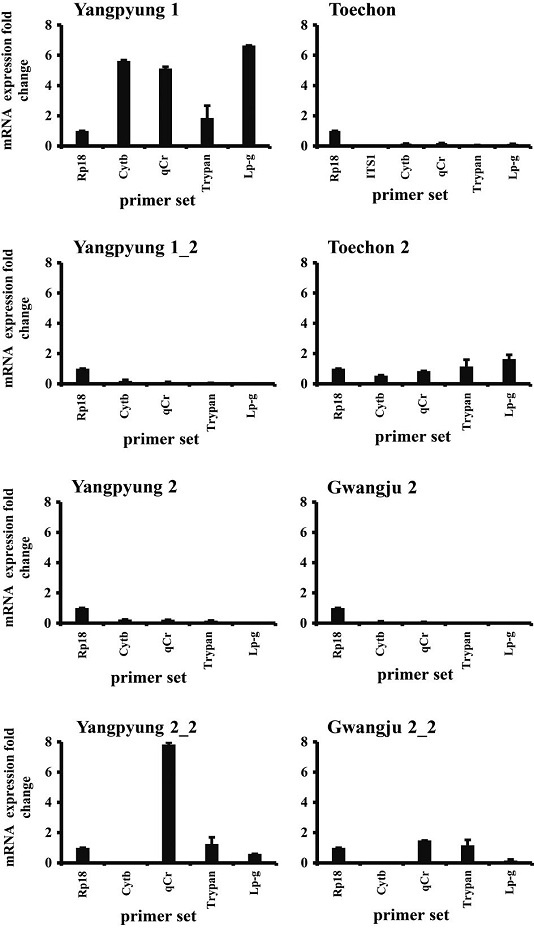
qPCR of L. passim primer with honeybee. Honeybee samples from Yangpyung, Gwangju and Toechon were ground with liquid nitrogen and mRNA was extracted. qPCR was used to investigate the presence of trypanosome parasite, L. passim. The statistical test used a T-test.
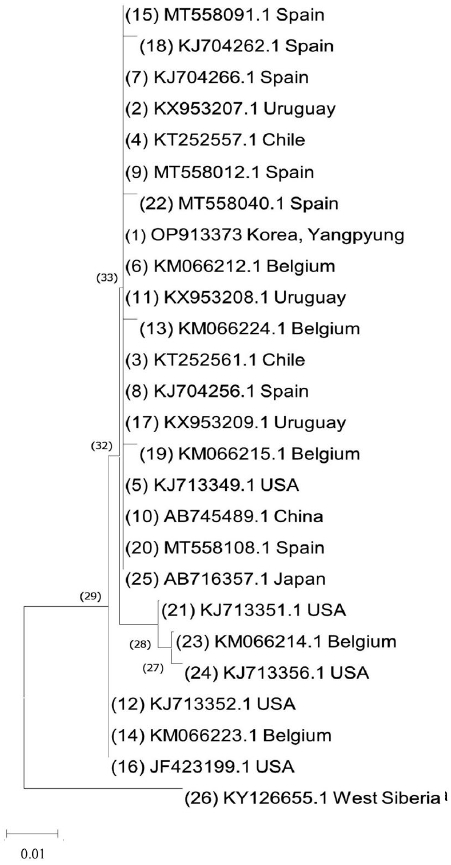
Fig. 2. Sequencing of samples Yangpyung 1 gene with L. passim primer. Yangpyung 1 was sequenced after agarose gel electrophoresis. After PCR by L. passim primer, Yangpyung 1 were purified by agarose gel electrophoresis and sequenced for alignment. The result of sequencing, Yangpyung 1 has 100% identity with L. passim.
적 요
본 연구는 한국에서 처음으로 양봉장 또는 꿀벌에 L. passim이 있음을 확인한 결과이다. 아직 한국에서는 L. passim의 감염이 보고된 바가 없었으나, 현재 L. passim은 전 세계적으로 감염 보고가 늘어나고 있다. 꿀벌을 수입하고 있는 국내 사정으로는 이제는 Trypanosome이 존재함을 인지하고 이에 대한 감염과 꿀벌에 미치는 영향에 대한 연구가 필요하다 (Ribani et al., 2021). 이 병원체로 인한 새로운 스트레스가 꿀벌 건강의 역학에 영향을 미칠 수 있음은 분명하다.
Acknowledgments
본 연구는 꿀벌 sample을 기증하신 농가의 도움과 농촌진흥청 (PJ015763)의 지원으로 수행된 결과이며 이에 깊은 감사를 드립니다.
References
-
Arismendi, N., M. P. Castro, M. Vargas, C. Zapata and G. Riveros. 2022. The trypanosome Lotmaria passim prevails in honey bees of different ages and stages of development. J. Apic. Res. 61(1): 63-69.
[https://doi.org/10.1080/00218839.2020.1828239]

-
Arismendi, N., A. Bruna, N. Zapata and M. Vargas. 2016. PCR-specific detection of recently described Lotmaria passim (Trypanosomatidae) in Chilean apiaries. J. Invertebr. Pathol. 134: 1-5.
[https://doi.org/10.1016/j.jip.2015.12.008]

-
Bartolomé, C., M. Buendía-Abad, C. Ornosa, P. De la Rúa, R. Martín-Hernández, M. Higes and X. Maside. 2022. Bee Trypanosomes: First Steps in the Analysis of the Genetic Variation and Population Structure of Lotmaria passim, Crithidia bombi and Crithidia mellificae. Microb. Ecol. 84(3): 856-867.
[https://doi.org/10.1007/s00248-021-01882-w]

-
Brown, M. J., R. Schmid-Hempel and P. Schmid-Hempel. 2003. Strong context-dependent virulence in a host-parasite system: reconciling genetic evidence with theory. J. Anim. Ecol. 72(6): 994-1002.
[https://doi.org/10.1046/j.1365-2656.2003.00770.x]

-
Buendía-Abad, M., P. García-Palencia, L. M. de Pablos, J. M. Alunda, A. Osuna, R. Martín-Hernández and M. Higes. 2022. First description of Lotmaria passim and Crithidia mellificae haptomonad stages in the honeybee hindgut. Int. J. Parasitol. 52(1): 65-75.
[https://doi.org/10.1016/j.ijpara.2021.06.005]

-
Castelli, L., B. Branchiccela, C. Invernizzi, I. Tomasco, M. Basualdo, M. Rodriguez, P. Zunino and K. Antúnez. 2019. Detection of Lotmaria passim in Africanized and European Honeybees from Uruguay, Argentina and Chile. J. Invertebr. Pathol. 160: 95-97.
[https://doi.org/10.1016/j.jip.2018.11.004]

-
Collins, A. M., V. Williams, J. D. Evans. 2004. Sperm storage and antioxidative enzyme expression in the honey bee, Apis mellifera. Insect Mole. Biol. 13(2): 141-146.
[https://doi.org/10.1016/j.jip.2016.07.001]

-
Jung, C. and S. K. Cho. 2015. Relationship between honeybee population and honey production in Korea: A historical trend analysis. J. Apic. 30(1): 7-12.
[https://doi.org/10.1111/j.0962-1075.2004.00469.x]

-
Larkin, M. A., G. Blackshields, N. P. Brown, R. Chenna, P. A. McGettigan, H. McWilliam, F. Valentin, I. M. Wallace, A. Wilm, R. Lopez, J. D. Thompson, T. J. Gibson, and D. G. Higgins. 2007. Clustal W and Clustal X version 2.0. Bioinformatics 23(21): 2947-2948.
[https://doi.org/10.1093/bioinformatics/btm404]

-
Merzlyak, E., V. Yurchenko, A. A. Kolesnikov, K. Alexandrov, S. A. Podlipaev and D. A. Maslov. 2001. Diversity and phylogeny of insect trypanosomes based on small subunit rRNA genes: polyphyly of Leptomonas and Blastocrithidia. J. Eukaryot. Microbiol. 48(2): 161-169.
[https://doi.org/10.1111/j.1550-7408.2001.tb00298.x]

-
Ravoet, J., J. Maharramov, I. Meeus, L. De Smet, T. Wenseleers, G. Smagghe and D. C. de Graaf. 2013. Comprehensive bee pathogen screening in Belgium reveals Crithidia mellificae as a new contributory factor to winter mortality. PLoS One 8(8): e72443.
[https://doi.org/10.1371/journal.pone.0072443]

-
Ravoet, J., R. S. Schwarz, T. Descamps, O. Yañez, C. O. Tozkar, R. Martin-Hernandez, C. Bartolomé, L. De Smet, M. Higes, T. Wenseleers, R. Schmid-Hempel, P. Neumann, T. Kadowaki, J. D. Evans and D. C. de Graaf. 2015. Differential diagnosis of the honey bee trypanosomes Crithidia mellificae and Lotmaria passim. J. Invertebr. Pathol. 130: 21-27.
[https://doi.org/10.1016/j.jip.2015.06.007]

-
Revainera, P. D., S. Quintana, G. Fernández de Landa, F. Meroi Arcerito, M. Lucía, A. H. Abrahamovich, S. Plischuk, M. J. Eguaras and M. D. Maggi. 2020. Phoretic mites on South American bumblebees (Bombus spp.) as parasite carriers: a historical input. Apidologie 51: 455-464. https://link.springer.com/article/10.1007/s13592-020-00733-w, .
[https://doi.org/10.1007/s13592-020-00733-w]

-
Ribani, A., V. J. Utzeri, V. Taurisano, R. Galuppi and L. Fontanesi. 2021. Analysis of honey environmental DNA indicates that the honey bee (Apis mellifera L.) trypanosome parasite Lotmaria passim is widespread in the apiaries of the North of Italy. J. Invertebr. Pathol. 184: 107628.
[https://doi.org/10.1016/j.jip.2021.107628]

-
Runckel, C., M. L. Flenniken, J. C. Engel, J. G. Ruby, D. Ganem, R. Andino and J. L. DeRisi. 2011. Temporal analysis of the honey bee microbiome reveals four novel viruses and seasonal prevalence of known viruses, Nosema, and Crithidia. PLoS One 6(6): e20656.
[https://doi.org/10.1371/journal.pone.0020656]

-
Stevanovic, J., R. S. Schwarz, B. Vejnovic, J. D. Evans, R. E. Irwin, U. Glavinic and Z. Stanimirovic. 2016. Species-specific diagnostics of Apis mellifera trypanosomatids: A nine-year survey (2007-2015) for trypanosomatids and microsporidians in Serbian honey bees. J. Invertebr. Pathol. 139: 6-11.
[https://doi.org/10.1016/j.jip.2016.07.001]

-
Williams, M. K. F., A. D. Tripodi and A. L. Szalanski. 2019. Molecular survey for the Honeybee (Apis mellifera L.) trypanosome parasites Crithidia mellificae and Lotmaria passim. J. Apic. Res. 58(4): 553-558.
[https://doi.org/10.1080/00218839.2019.1568956]

- Winter, K., L. Adams, R. Thorp, D. Inouye, L. Day, J. Ascher and S. Buchmann. 2006. Importation of non-native bumble bees into North America: potential consequences of using Bombus terrestris and other non-native bumble bees for greenhouse crop pollination in Canada, Mexico, and the United States. [Google Scholar]
-
Yamamoto, S., S. Nakamura, K. Nakayama, K. Kusakisako, K. Watanabe, H. Ikadai and T. Tanabe. 2023. Molecular detection of Lotmaria passim in honeybees in Japan. Parasitol. Int. 93: 102711.
[https://doi.org/10.1016/j.parint.2022.102711]

