초고속 역전사 실시간 중합효소 연쇄반응을 이용한 한국산 낭충봉아부패병 바이러스의 가장 빠른 검출법
초록
꿀벌의 병원성 바이러스인 낭충봉아부패병 바이러스의 변종으로, 국내에서 한국산 낭충봉아부패병 바이러스(Korean Sacbrood Virus; kSBV)가 처음 인식된 것은 2010년이었다. 2010~2015년의 기간에 국내 토종벌의 거의 90% 이상이 이 바이러스의 감염에 의하여 큰 피해를 보았으니, 아직 이 바이러스의 감염을 차단하거나 통제 하는 방법, 왜 토종벌에만 감염이 되는지에 대하여 알려진 바는 거의 없다. 그렇기 때문에 빠르고 쉬운 검출방법 개발이 매우 중요하다 할 것이다.
본 연구에서는 가장 빠르고 쉬운 한국산 낭충봉아부패병 바이러스 검출방법을 고안한 것으로, 초고속 실시간 PCR(URRT-PCR)과 cDNA 합성과정을 최적화하여, 한국산 낭충봉아부패병 바이러스에 감염된 유충으로 부터 한국산 낭충봉아부패병 바이러스 검출에 소요되는 시간을 최소화한 것이다. 최적화된 한국산 낭충봉아부패병 바이러스-특이 초고속 역전사 실시간 PCR법을 적용하면, 낭충봉아부패병 바이러스의 검염여부는 7분 12초 만에 판정할 수 있었으며, 최소 2.5×101 kSBV의 DNA복사체도 정량적으로 측정할 수 있었다. kSBV-URRTRT PCR법은 실험실 뿐 아니라 양봉현장에서 한국산 낭충봉아부패병 바이러스 감염을 정량적으로 검출해 내는 유용한 방법이 될 것으로 기대한다.
Abstract
New strain of Sacbrood virus as pathogen of honeybee, named korean Sacbrood virus (kSBV) was firstly recognized in Korea on 2010. During the time 2010-2015 near 90% populations of Apis cerana in Korea were heavy damaged by infection of kSBV. It is not clear yet, how to prevent it or how to control it and why infect only to A. cerana. These are why this development of rapid and easy detection method is important. In this study, the fastest and easy detection method against kSBV was designed. To optimize Ultra-Rapid Real-Time PCR (URRT-PCR) and cDNA generation the kSBV-detection time from kSBV-infected larvae could be minimized. Using optimized kSBV-specific ultra-rapid reverse transcription real-time PCR (kSBV-URRTRT-PCR), minimum detection time for kSBV-infection was completed inner 6 minutes 12 seconds from kSBV-infected larvae quantitatively. Alternatively 2.5×101 copies of cDNA from kSBV-genome could be detected using developed method. kSBV-URRTRT PCR might be helpful to detect kSBV quantitatively in laboratory and also in apiary field.
Keywords:
Korean sacbrood virus, URRT-PCR, Reverse transcription, Rapid detection, Honeybee키워드:
한국산 낭충봉아부패병 바이러스, 초고속 역전사 실시간 PCR, 역전사반응, 빠른검출법, 꿀벌서 론
낭충봉아부패병 바이러스(Sacbrood virus; SBV)는 1913년에 처음 보고되었으며, 꿀벌 유충에 질병을 야기하는 심각한 바이러스 중 하나로 1964년에 그 특징이 규명되었다(Bailey, 1964). 낭충봉아부패병 바이러스는 분류학적으로 iflavirus 속에 속하며 양성가닥 RNA(positive strand RNA)를 가진 picornavirus이다(Ghosh et al., 1999). SBV는 꿀벌 유충에서 발병되며, 감염된 유충의 증상은 주머니(Sac)를 형성하며, 노란색에서 검은 갈색으로 점차 부패 되면서 번데기로 변태 되지 못하고 죽게 된다(Bailey et al., 1975). 일반적으로 서양종 꿀벌(Apis mellifera)에서 검출되면 발견 지역에 관계없이 낭충봉아부패병 바이러스라 통칭하나, 동양종 꿀벌(Apis cerana)에서 검출된 낭충봉아부패병 바이러스는 지역에 따라 염기서열 차이를 보이기 때문에 검출된 지역에 따른 명명을 사용하여, 중국에서 보고된 것은 중국산 낭충봉아부패병 바이러스(Chinese sacbrood virus; cSBV) 라 불리며(Ma et al., 2011), 한국산 Apis cerana에서 검출된 것은 한국산 낭충봉아부패병 바이러스(Korean sacbrood virus; kSBV)로 명명되었다(Han et al., 2011).
현재 낭충봉아부패병 바이러스에 대한 치료법으로, 그 효능이 입증된 것을 찾기는 매우 지난하기에, 꿀벌에서 낭충봉아부패병 바이러스의 빠른 검출 및 진단을 통하여 감염을 인지하고, 우선 이 감염체로부터 감염되지 않은 꿀벌을 격리시키는 것이 유력한 대처방법이라 할 것이다.
꿀벌에서 병원체를 검출해 내는 방법 중 현재 병원체의 유전자 검사법이 주를 이루고 있다. PCR 검사법은 가장 민감하고 특이성을 가진 방법으로 알려져 있으며, 실시간 PCR법 (Real-time PCR; RT-PCR)은 정량적, 정성적 분석이 가능하여 보다 널리 사용되고 있다.
최근 극소량의 반응액을 사용하는, 이른바 chip에 기초한 PCR방법은 종전 일반 PCR 기기(thermal cycler)를 사용하던 방법들과 비교하여 많은 장점을 입증하고 있으며, 예로써 GenSpector TMC-1000(Samsung, Korea)은 칩 기반 실시간 중합효소연쇄반응용 PCR기기로써 종전의 기기가 이루지 못한 빠른 온도변환을 가능하게 하였다. 이 기기가 사용하는 유리-실리콘 접합 미세 배열 칩(glass-silicone based micro-chip)은 소량인 6μl의 PCR 반응액을 사용하며, 열전도를 증가시킨 물질들의 적용과 배열로써, PCR 반응액에 유효한 균일 온도를 빠르게 전달해줄수있다(Cho et al., 2006).
이런 장점들을 수용한 초고속 실시간 PCR(Ultrarapid real-time PCR; URRT-PCR)은, PCR의 온도변환을, 100μl의 전용 튜브를 사용하는, 종전의 PCR cycle 보다 획기적으로 빠르게 반응을 진행시킬 수 있었으며, 이 기종을 이용하여 개발된, two-step URRT-PCR은, 설사유발 대장균(Escherichia coli)의 독성 stx2 유전자를 7분 8초만에 검출한 바 있었으며, 또한 Vibrio parahaemolyticus의 tdh 유전자를 7분 10초만에 빠른 검출한 바 있어 초고속 PCR이라 명명되게 되었다(Kim et al., 2010a; Kang et al., 2010b).
본 연구에서는 한국산 낭충봉아부패병 바이러스의 빠른 검출을 위하여, PCR의 온도 조건인 변성, 중합, 신장 단계(step)의 시간을 최소화하고자 하였으며, 유효한 PCR 회전수(cycles)를 최소화 하여 초고속 PCR이 가능하도록 최적화 시켰으며, 아울러, 이런 초고속 온도의 변화에서 가장 잘 기능할 수 있는 내열성 DNA 중합효소(Extreme thermophile DNA polymerase)를 찾고자 하였다. 또한, 한국산 낭충봉아부패병 바이러스가 양성 가닥RNA만을 유전체로 가지고 있기에, 꿀벌 시료로부터 한국산 낭충봉아부패병 바이러스를 검출하기 위하여, 꿀벌시료로부터 전체 RNA 추출 및 cDNA로 합성하는 시간을 유효하게 최소화 하고자 하였다.
따라서, 본 연구에서는 한국산 낭충봉아부패병 바이러스의 빠른 검출을 위하여, 시료로부터 전체RNA 추출 시간을 최소화 하고, reverse transcription 시간을 최소화하고, PCR의 시간도 최소화한, 초고속 역전사 실시간 PCR방법(URRTRT-PCR; Ultra-Rapid Reverse-Transcription Real-Time PCR)을 개발하고자 하였다.
재료 및 방법
시료의 준비(꿀벌 애벌레)
본 연구에 사용된 병성 시료는 대한민국 옥천에서 수집된 kSBV에 감염된 동양종 꿀벌(Apis cerana)의 유충(larvae)들이며, 실험기간 내 분주하여 냉동한 후 사용되었다. 이 시료에서 kSBV의 특이 염기서열이 발견되었으며, 표준 kSBV의 염기서열은 Genbank accession Number HQ322114을 사용하였다.
전체 RNA 추출
감염이 의심되는 동양종 꿀벌 유충 시료를 1.5ml 원심분리관에 각 2마리씩을 넣고 homogenizer로 조직을 분쇄하였다. 여기에 iNtRON사의 R&A-Blue™키트를 사용하여 제작자의 지침에 따라, 전체RNA를 분리하였으며, 최종과정은, 진탕액을 13,000 rpm, 10분, 4°C 원심분리로 상하층을 분리하고, 그 상층액을 취하여 전체 RNA로 사용하였다. 분리된 RNA용액은 분광광도계(spectrophotometer)로 정량하였으며, 분주 후, -70°C에 보관하며 사용하였다.
cDNA 합성(Reverse Transcription)
cDNA 합성은 전체 RNA를 1μg을 사용하였고, oligo dT대신 특이 reverse primer를 사용하였다. dNTP, 100mM DTT, Reaction buffer, MML-V reverse transcriptase(Bioneer Inc, Korea)를 사용하였으며, 반응액을 42°C에 정치시켜 cDNA를 합성하게 하였다. cDNA 제작 시간은 각각 0 min, 1 min, 5 min, 15 min, 30 min으로 다르게 하였다.
Plasmid construction
주형 DNA를 확보하기 위해 kSBV 유전체(Genbank Accession Number HQ322114)를 표준으로 각 primer set들의 DNA fragment를 얻기 위하여 각각 PCR로 증폭하였다. 증폭하여 확보한DNA는 pBX 또는 pGEM-3zf T-Cloning vector를 사용하여 재조합체로 확보하였다. 대장균 숙주는 DH5αF'을 사용하였으며, 재조합체는 각PCR의주형DNA로 사용하였다(Fig. 1, Table 1).
Primer 제작
kSBV (Genbank Accession Number. HQ322114)의 유전체 내부 open reading frame (ORF) 중 capsid protein, peptidase-c3g, RNA-dependent RNA-polymerase (RdRP)를 암호화 하는 위치에 각각 특이 primer쌍을 설계, 제작하였다. 이중 URRT-PCR에 최적화된 특이 primer쌍은 capsid protein 내부에 위치한 것이었다(Fig. 1, Table 1).
초고속 실시간 PCR을 위한 kSBV-primer 쌍의 선별
설계된 kSBV-특이 primer쌍 들은, 각기 비교 PCR을 시행하여 그 기능을 평가하였다. 각 PCR은 해당 primer쌍에 대한 재조합 plasmid를 2.49×105 molecules 사용하였으며, 각 5 pmole primer, 2x SsoFast™ EvaGreen® Supermix (BIORAD)로 합 20μl의 반응액을 조성하였으며, 각기 real-time PCR을 시행하였다. Real-time PCR 조건은 전 변성 94°C, 10 sec를 시행한 후, 변성 94°C, 5 sec, 중합/신장 60°C, 5 sec의 조건으로 50 cycle 진행하였으며, PCR product의 melting 온도 분석은 70°C에서 90°C까지 수행하였다.
Temperature gradient RT-PCR
각 primer set의 최적 중합 온도 조건은 gradient RT-PCR을 수행하여 그 결과로 결정하였다. RT-PCR은 2x SsoFast™ EvaGreenㅁ Supermix (BIORAD)를 사용하였고, forward, reverse primer 각 5 pmole, target DNA를 각 2.49×105 molecules가 사용되었으며 총 20μl volume으로 수행하였다. Gradient real-time PCR은 Exicycler™ Quantitative Thermal Block (Bioneer)로 수행하였고, PCR 조건은 전 변성 94°C 10 sec 이후, 변성 94°C 5 sec, 중합/신장 42~70.67°C 5 sec의 조건으로 40 cycle 진행하였고, PCR 산물의 Melting 온도 분석은 70°C에서 90°C까지 수행하였다.
RT-PCR을 사용한 민감도 확인
kSBV-VP2 primer set의 민감도 확인을 위하여, 우선 pGEM-kSBV-vp2 plasmid를 serial dilution하여 2.49×107-1 분자의 각기 다른 양을 주형을 사용하였다. Real-Time PCR에는 각 5 pmole kSBV-vp2 primer, 2x SsoFast™ EvaGreenㅁ Supermix (BIORAD)을 사용하였다. Real-time PCR 조건은 전 변성 94℃, 10 sec 이후, 변성 94°C, 5 sec, 중합/신장 60°C, 5 sec의 조건으로 50 cycle 진행하였고, PCR 산물의 melting 온도 분석은 70°C에서 90°C까지 수행하였다.
꿀벌 genomic DNA의 오염 하에 specific RT-PCR의 민감도 확인
Contamination test는kSBV-vp2 primer의 민감도 test에 증류수 대신 꿀벌의 genomic DNA 1μg을 사용하였고 나머지 조성과 조건은 동일하게 하였다.
RT-PCR을 위한 각 step 시간의 최소화
RT-PCR의 온도조건 중 변성 94°C는 5 sec, 3 sec, 2 sec로 축소하였으며, 이와 연동하여 중합/신장 60°C도 각 5 sec, 4 sec, 3 sec로 시간을 축소시켜 RT-PCR을 수행하였다. PCR 산물의 melting 온도 분석은 76°C에서 86°C까지 수행하였다.
DNA polymerase (master mix) 변경에 따른 URRT-PCR 속도 변화
URRT-PCR 반응을 더 빠르게 진행 하고자 DNA-polymerase의 기원과 기능 이상이 한 2가지 PCR premix를 사용하였다. 사용된 premix는 2X SsoFast™ EvaGreenㅁ Supermix (BIORAD)와2X fast RT-PCR premix mix이며, 이들을 초고속 PCR에 적용시켜 상호 성능을 비교하였다. v2X SsoFast™ EvaGreenㅁ Supermix (BIORAD)는변성95°C, 5 sec, 중합/신장60°C 5 sec으로 2-step URRT-PCR 40 cycle을 수행하였고, 2X fast RT-PCR premix mix는 변성95°C 5 sec, 중합 60°C 5 sec, 신장 72°C, 5 sec로 3-step URRT-PCR 40 cycle을 수행하였다.
초고속 실시간 PCR을 사용한 변성, 중합 온도의 최적화
kSBV-vp2 primer set의 URRT-PCR을 사용한 온도 조건 최적화는 전 변성은 95°C, 10 sec로 진행 하였고, 이후 최적 변성 온도는 93°C, 94°C, 95°C, 96°C, 97°C 2 sec로 다르게 하고 중합은 60°C로 고정하여 40 cycle을 수행하였다. 최적 중합 온도를 알기 위해 58°C, 59°C, 60°C, 61°C, 62°C 5 sec로 다르게 하였고, 이때, 변성 온도는 95℃로 고정하여 40 cycle을 수행하였다. Melting 온도 분석은 70°C부터 90°C (0.5°C/sec)까지 진행하였다. URRT-PCR은 target DNA로 2.49×107 분자를 사용하였으며, 각 10 pmole kSBV-VP2 primer와 2x fast RT-PCR premix mix를 전체 용량 6μl로 조성하여 수행하였다.
URRT-PCR을 사용한 Primer 민감도 확인
kSBV-vp2 primer set의 민감도 확인을 위하여 pGEM-kSBV-vp2 plasmid를 serial dilution하여 2.49×107-1 분자의 각기 다른 양의 주형을 사용하였다. kSBV-vp2 primer set의 URRT-PCR에 사용한 온도 조건은 전 변성 95°C, 10 sec으로 진행 하였고, 이후, 변성 95°C, 5 sec로 하였으며, 중합 온도는 60°C, 5 sec로 신장 온도는 72°C, 5 sec로 하여 40 cycle을 수행하였다. Melting 온도 분석은 70°C부터 90°C(0.5°C/sec)까지 진행하였다.
URRT-PCR의 각 step 시간 최소화
URRT-PCR 조건을 전 변성 95°C 10 sec으로 하였으며, 이후, 변성 95°C를 2 sec, 또는 1 sec으로 하였고, 중합 60°C를 5 sec, 4 sec, 3 sec으로 신장 온도는 72°C로 하여 PCR을 진행하였다. URRT-PCR은 target DNA 2.49×107 분자를 사용하였으며, 각 10 pmole kSBVvp2 primer와 2x fast RT-PCR premix mix를 전체 용량 6μl로 조성하여 수행하였다.
결 과
Real-Time PCR을 사용한 초고속 PCR용 Primer쌍의 선별
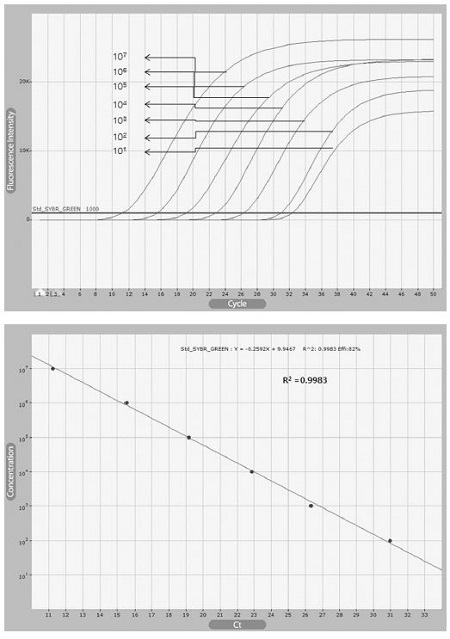
kSBV genome을 기반으로 제작된 각각의 특이 primer sets(Table 1)들로 각각의 target DNA 105, 104 분자를 각기 RT-PCR로 증폭시켰다. 각 primer sets를 target DNA 105 분자에서 확인한 결과, 각각 primer set kSBV-URRT, kSBV-vp1, kSBV-c3-823, kSBV-vp2, kSBV-RdRp은 12.96, 24.54, 22.80, 20.19, 29.37의 threshold cycles (CT value) 값을 보였다(Fig. 2 and Table 2). 한편, kSBV-vp2 primer set는 RT-PCR의 최종 형광값에서 가장 높은 수치를 보였다.
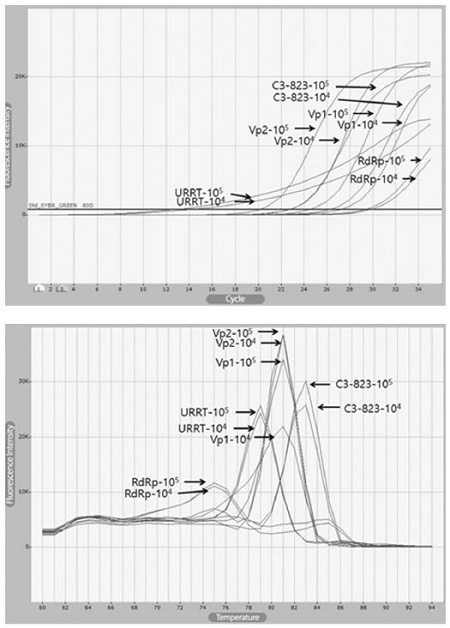
Primer selection using Real-Time PCR and melting analysis of RT-PCR product. Each target gene was amplified by each primer set using Real-time PCR. kSBV-vp2 amplicon was detected as highest fluorescence more than others.
RT-PCR을 사용한 primer 민감도 확인
kSBV-vp2 plasmid의 serial dilution에 따른 kSBV-vp2 primer set의 민감도 확인 결과 101 분자의 target DNA까지 진단할 수 있는 것이 확인되었다. 회귀상수(Regression coefficient), R2 값은 0.9983으로 확인되어 높은 정량성을 보이는 것이 확인되었다(Fig. 3).
RT-PCR 각 step의 시간 축소
kSBV-vp2 plasmid DNA의 target DNA 105 molecules을 사용하여 2-step RT-PCR 조건에서 변성과 중합/신장 시간을 각각 30 sec으로부터 각각 2 sec, 3 sec으로 줄인 결과에서도 PCR 증폭이 그래프로 확인되었다(Fig. 4).
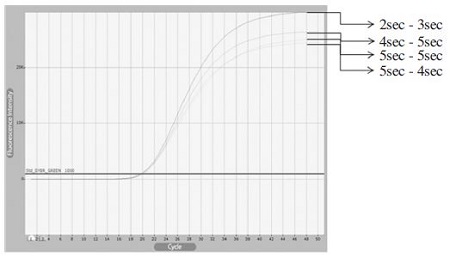
Minimized time of denaturation and annealing/extension conditions were affect to real-time PCR amplicon. In real-time PCR used kSBV-vp2 primer set, even though, denaturation and annealing/extension time were decreased, Ct values were rarely changed. Final fluorescence graph were unrelated with PCR condition times.
cDNA synthesis 시간 축소
kSBV에 감염된 꿀벌 애벌레로부터 추출한 전체 RNA를 1 ng사용하여 cDNA를 제작하였다. MML-V reverse transcriptase (Bioneer)를 사용한cDNA는 42°C에서 각기 1, 2, 3, 4, 5, 15, 30, 60분간 반응시켰으며, 이로써 제작된 각각의 cDNA들은 kSBV-vp2 primer를 사용하여 real-time PCR로 그 양(target DNA의 분자수)을 측정하였다. Real-time PCR 결과 29.56, 29.55, 29.43, 28.75, 28.54, 28.38, 27.57, 26.14의 threshold cycles (CT value) 값을 보였다(Fig. 5, Table 3).
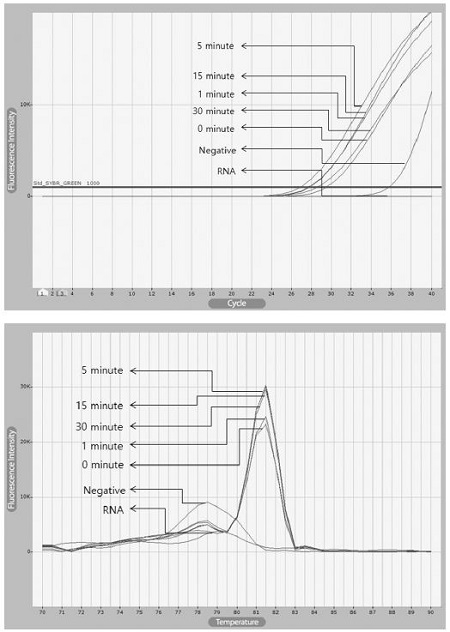
Real-Time PCRs with kSBV-vp2 primer using various cDNAs generated on various time of incubation. cDNA products were synthesized by MMLV reverse transcriptase under the same conditions with different reaction times. They were performed by real-time PCR. In fluorescence graph, Ct values of 30 min reaction time and 1 min reaction time were rarely changed. It was shown that reaction time were not important factor at cDNA synthesis. Actually, created cDNA from 1 min reverse transcription reactions could amplify target gene by real-time PCR.
DNA-polymerase(Master mix)의 변경에 따른 초고속 PCR 시간 변화
URRT-PCR에서 2x SsoFast™ EvaGreenㅁ Supermix와 2X fast RT-PCR premix mix를 각기 사용하고, 동일한 kSBV-vp2 primer set, 동일한 target DNA을 사용하여 그 기능을 비교 분석하였다.
URRT-PCR에서 master mix를 2x fast RT-PCR premix를 사용한 것은 Ct값이 21.55 cycles로, 2x SsoFast™ EvaGreenㅁ Supermix를 사용한 것은 Ct 값이 33.99 cycles로 나타났으며, 그 차이는 전자가 후자에 비하여 12.44 cycles나 더 빠른 것으로 나타났다. 또한 40 회전 최종 형광값도 전자가 후자에 비하여 약 2배 더 높은 것으로 확인되었다(Fig. 6).
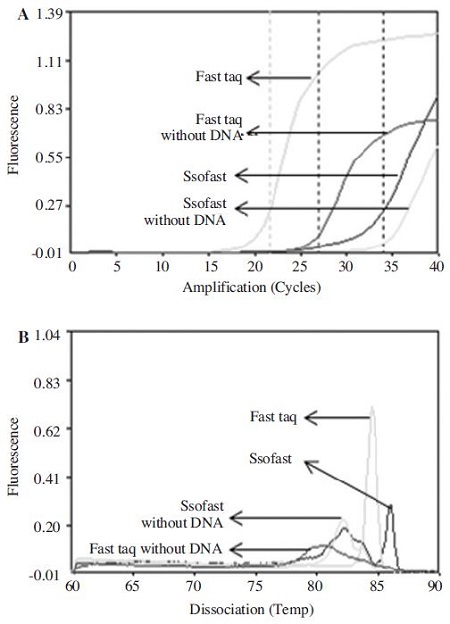
Comparison between DNA-polymerase (master mix) using Ultra-rapid real-time PCR. (A) URRT-PCR fluorescence graph using kSBV-vp2 primer set, (B) Melting analysis. 2x fast RT-PCR premix was performed under the 95°C pre denaturation 1min, 94°C denaturation 5 sec, 60°C, annealing 5 sec, 72°C Extension 5 sec conditions, using kSBV-vp2 primer set. 2x SsoFast¢‚ EvaGreen Supermix was performed under the 95°C pre denaturation 1 min, 94°C denaturation 5 sec, 60°C annealing/extension 5 sec conditions, using kSBVvp2 primer set. In URRT-PCR, 2x fast RT-PCR premix had 21.55 ct value, 2x SsoFastTM‚ EvaGreen Supermix had 33.99 Ct value. 2x fast RT-PCR premix had confirmed more fast amount of 12.44 cycles than 2x SsoFastTM‚ EvaGreenㅁ Supermix.
URRT-PCR 중 각 단계(step) 온도의 최적화
최적 조건의 kSBV-specific URRT-PCR은, kSBV-vp2 primer set를 사용할 경우, 전 변성은 94°C, 10 sec로 진행 하고, 이후 변성 온도는 94°C에서 가장 높은 형광값을 보이는 것으로 나타났으며, 중합 온도는 60°C에서 가장 높은 형광 값을 보이는 것으로 나타났다(Fig. 7).
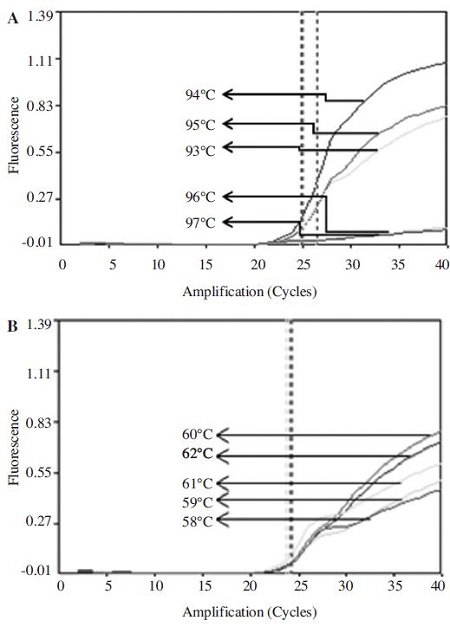
Optimization of denaturation and annealing conditions using kSBV-vp2 primer set in URRT-PCR. 2.49×107 molecules of target DNA, 10 pmole kSBV-vp2 primer sets and 2x fast RT-PCR premix were used for URRT-PCR in total volume 6 ul. (A) Denaturation temperature was performed to 93°C, 94°C, 95°C, 96°C, 97°C under the fixed 60°C, annealing temperature. (B) Annealing temperature was performed to 58°C, 59°C, 60°C, 61°C, 62°C under the fixed 94°C, denaturation temperature. URRT-PCR denaturation and annealing conditions were defined to 94°C, 60°C, because they had shown the highest fluorescence value.
kSBV-vp2 primer을 사용한 URRT-PCR의 진단 한계
kSBV-vp2 plasmid DNA는 107-1 분자로 serial dilution하여 검출한도를 측정하였다. kSBV-vp2 primer를 사용한 kSBV-specific URRT-PCR의 진단 한계는 2.49×102 molecules (target DNA) 까지 검출할 수 있었으며, 초기 주형량 2.49×107~2.49×105 범위에서 정량성을 입증할 수 있었다(Fig. 8).
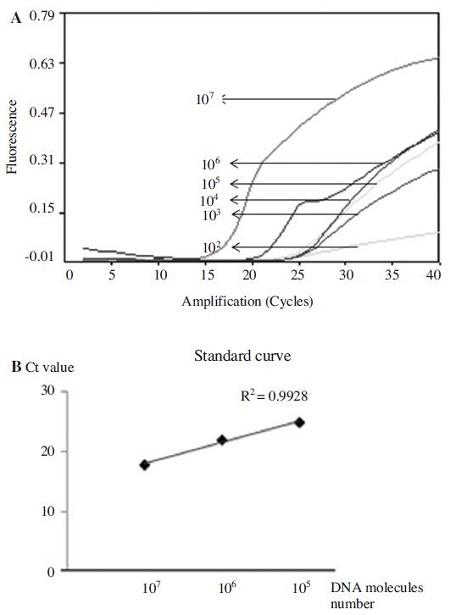
Detection limit using URRT PCR with kSBV-vp2 primers. Using serial dilution of pGEM-kSBV-vp2 plasmid DNA, each template of 2.49×107,6,5,4,3,2,1 molecules was amplified by URRT-PCR. kSBV-specific URRT-PCR was performed under optimum conditions. Detection limit of kSBV-specific URRT-PCR was 2.49×102 molecules, and a permitted limit of quantity was 2.49×107~2.49×105.
진단 가능한 URRT-PCR 최소화 시간
kSBV-vp2 plasmid DNA의 107 분자의 target DNA를 사용하여 URRT-PCR 조건의 변성, 중합, 신장 시간을 감소시킨 결과, 변성 1 sec, 중합 1 sec, 신장 3 sec의 시간에서도 kSBV-vp2 Primer를 이용한 kSBV-specific 초고속 PCR에 의한 target DNA 증폭이 가능함이 확인되었다(Fig. 9).
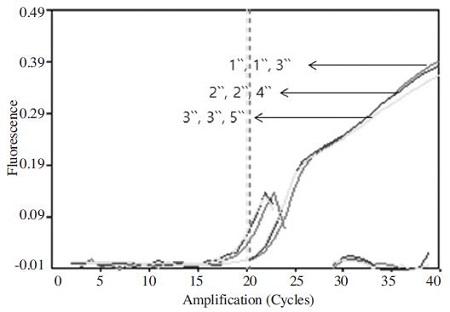
URRT PCR for minimizing kSBV detection time : minimized time of each cycle step. URRT-PCR was amplified under the optimum conditions, but time of each cycles step were performed differently as denaturation 95°C 3 sec, 2 sec, 1 sec, annealing 60°C 3 sec, 2 sec, 1 sec, extension 72°C 5 sec, 4 sec, 3 sec. In URRT-PCR, minimized time of each step shown that denaturation 1 sec, annealing 1 sec and extension 3 sec conditions could amplify target gene. Process of amplification has same appearance of Ct value and amplicon. Minimized times of each steps were rarely affect to total PCR reaction. In graph was indicated sec (second).
가장 빠른 진단 방법을 이용한 kSBV 진단
kSBV-vp2 primer를 사용하고, 2.49×105 분자의 target DNA를 대상으로 40회전의 URRT-PCR을 수행하였다. 반응조건은 95°C 전 변성 1 min 이후, 95°C 변성 1 sec, 60°C 중합 1 sec, 72°C 신장 3 sec 이었으며, 그 결과, 40회전 PCR에 총 14 min 28 sec의 시간이 소요되었고, 이 중 검출에 소요된 시간은 6 min 12 sec (24.86 CT)에서 kSBV 증폭 및 검출이 확인되었다(Fig. 10).
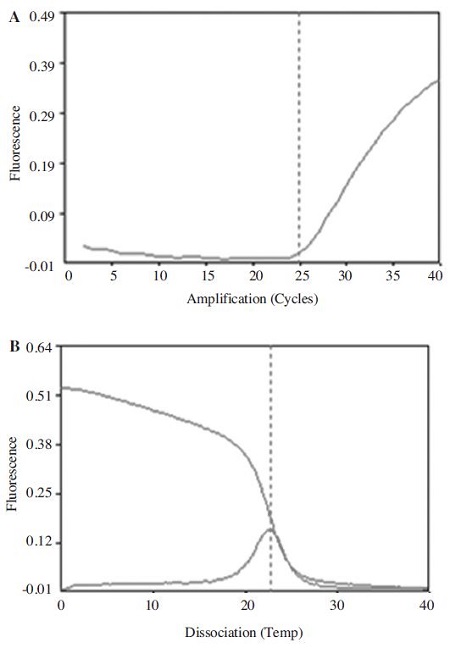
Ultra-rapid kSBV detection using ultra-rapid real-time PCR. (A) URRT-PCR Ct graph using kSBV-vp2 primer set, (B) Melting temperature analysis, kSBV-specific URRT-PCR was performed under the optimum conditions. URRT-PCR 40 cycle was taken for 14 min 28 sec, detection of kSBV was confirmed at 6 min 12 sec in 24.86 Ct value.
고 찰
꿀벌로부터 병원체를 검출해 내는 방법 중 가장 민감하고 특이성이 높은 방법은 실시간(real-time) PCR법으로 인정되고 있다. 그러나 실시간 PCR법은 병원체 검색의 다른 방법인 면역크로마토그라피법(Immunochromatography)과 비교하면 간편성(easy)과 검사 소요시간(rapidity)의 면에서 크게 불리하여 현장검사와는 거리가 먼 실험실적 방법으로 제한되어 왔다.
본 연구는 실시간 PCR법을 검사시간의 측면에서 크게 개선하여 초고속 PCR의 개념으로 확장시키고자 하였으며, 한국산낭충봉아부패병 바이러스와 같은 RNA-genome의 병원체를 검출하기 위하여 역전사-실시간 PCR을 최단시간에 완료시킬 수 있는 방법을 강구한 것이다.
먼저, 한국산낭충봉아부패병 바이러스와 같은 RNA genome을 DNA로 전환시키는 cDNA 합성법은, 역전사 효소(예 MMLV-reverse transcriptase)를 사용하여 왔고, 오랜 기간 그 반응시간은 30분 이상 1시간까지 별다른 숙고 없이 수행되어 왔다. 그러나, 역전사 반응의 DNA합성의 시간 당 생산성은, PCR의 그것과는 비교될 수 없을 정도로 낮으며, 초고속 역전사 실시간 PCR(URRTRT-PCR)을 추구하기 위하여 cDNA-generation의 시간 단축은 필수적이라 할 것이다. 이역전사 반응의 절대 반응시간에 대하여, Giang et al. (2015)이 BQCV (Black queen cell virus)의 검출 실험에서 적시한 바 있으며, 역전사 반응을 1분, 5분 진행 후 PCR결과도 특이 PCR산물을 충분 양 생산할 수 있음을 보여주었다.
본 연구의 결과는 한국산낭충봉아부패병 바이러스를 대상으로 한 것이며, cDNA 합성시간을 1분으로 단축하여도 target DNA를 충분히 PCR 증폭시킬 수 있음을 입증하였으며, 30분의 cDNA 합성시간을 사용한 시료에 비하여 단지CT 값이 0.71 밀리는 정량적 결과만을 보였다. 또한 역전사 반응액을 조성하고, 즉시, 즉 반응 온도 및 시간을 주지 않고 바로 PCR증폭과 연결시켰을 경우에도 target DNA의 증폭이 가능하였던 것은, 실험목적 상 이 과정의 축소가 고려되어야 할 것이다(Fig. 5; Table 3).
초고속 PCR에서는 1초 변성, 1초 중합, 3초 신장의 초고속 PCR 조건을 수행한다. primer쌍에 따라 이를 수행할 수 있는 능력에서 우열이 있음을 보여주었으며, 이는 우선 primer의 염기서열에 기인한 주형 염기서열과 혼성화 능력(annealing ability)이 primer쌍에 따라 다른 것으로 해석되었다. 초고속 PCR에서 이 혼성화 능력이 우수한 primer쌍을 선발하는 것은 기본적 중요 요소로 생각된다.
한편, 초고속 실시간 PCR의 결과에서 분석되는 초기기질의 양에 대한 정량성은, 기존의 실시간 PCR과 같은, 높은 정량성이 인정되며, kSBV-vp2 primer쌍을 사용한 초고속 실시간 PCR은 각 회전(cycle), 각 단계(step)의 반응시간을 각 3초~5초 수준으로 늘릴 경우, target DNA 101 분자 수준까지 매우 정확하게 초기 주형량의 정량이 가능함을 보여주었다. 그러나, 각단계의 반응시간을 1초~3초로 감소시켰을 경우 유효한 정량성은 target DNA 105 분자 이상으로 약화됨을 보여주었으며, 이는 초고속 PCR이 나타내는 정량 PCR(quantitative PCR)로써의 한계일 수도 있을 것이다.
별도로, 초고속 PCR에 가장 적합한 DNA-polymerase의 발굴가능성을 조사해 보았다. 2X fast RT-PCR premix와 2X SsoFast™ EvaGreenㅁ Supermix는 각기 다른 기 원 의 극 내 열 성 DNA 중 합 효 소 (extreme thermophile DNA-dependent DNA polymerase) 및 부가 요소를 사용하는 것으로 알려져 있으며, 이 두 종의 premix가 URRT-PCR의 조건에서 어떤 차이를 보이는 지를 보고자 한 것이다. 결과를 요약하자면 전자는 21.55의 Ct 값을 보였으며, 후자는 33.99의 Ct 값을 보임으로, 매우 큰 차이를 나타냈으며, 이는 URRT-PCR이라는 새로운 조건에서 최적의 DNA중합효소는 기존의 DNA중합효소와 다를 수 있음을 보여준 것이라 할 것이다.
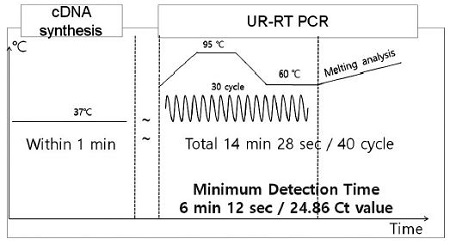
URRT-PCR model for the most fastest detection of kSBV. Using minimized times of cDNA synsthesis method and ultra-rapid real-time PCR method, UR-RT-RT PCR model for detection of Korean sacbrood virus rapidly. Using kSBV minimized detection model, detection time was totally 14 min 28 sec / 40 cycles containing cDNA synthesis, melting analysis. Minimum detection time of kSBV was 7 min 12 sec (in 24.86 Ct value) containing cDNA synthesis.
본 연구에서 개발된 한국산 낭충봉아부패병-특이 초고속 역전사 실시간 PCR (kSBV-specific Ultra-Rapid Reverse Transcription Real-Tme PCR)법은, cDNA 제작시간 1 분, 초고속 실시간 PCR 6분 12초(CT 값 24.86)으로, 한국산 낭충봉아부패병 검출을 위한 가장 빠른 유전자 검사법일 것이다. 본 검사법의 개발로써 꿀벌의 타 병원체 검출을 위한 초고속 유전자 진단법이 후속 개발되기를 기대하며, 나아가 지카바이러스 등 공공의 관심을 받고 있는 병원체의 검출에도 적용되기를 기대한다.
Acknowledgments
본 연구는, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(과제번호 115058-02, 과제번호 115102-03), 농생명산업기술개발사업(312027-03) 및 수출전략기술개발사업(과제번호 115067-02), 그리고 2016 학년도 경기대학교대학원 연구원장학생 장학금 지원에 의하여 수행되었음.
References
-
Bailey, L., (1975), Recent research on honey bee viruses, Bee World, 56, p55-64.
[https://doi.org/10.1080/0005772X.1975.11097544]

-
Cho, Y.K., J.T. Kim, Y.S. Lee, Y.A. Kim, K. Namkoong, H.K. Lim, K.W. Oh, Sh.H. Kim, J.I. Han, C.S. Pak, C.S. Ki, J.R. Choi, C. Myeong, H.K. Ko, (2006), Clinical evaluation of micro-scale chip-based PCR system for rapid detection of hepatitis B virus, Biosens. Bioelectron, 21, p2161-2169.
[https://doi.org/10.1016/j.bios.2005.10.005]

-
Ghosh, R.C., B.V. Ball, M.M. Willcocks, and M.J. Carter, (1999), The nucleotide sequence of sacbrood virus of the honey bee: an insect picornavirus, J. Gen. Virol, 80, p15541-1549.
[https://doi.org/10.1099/0022-1317-80-6-1541]

-
Giang, T.H.L., J.S. Lee, S.J. Yong, and B.S. Yoon, (2015), Development of ultra-rapid reverse transcription real-time PCR for detection against Black queen cell virus in honeybee, J. Apiculture, 30(3), p171-179.
[https://doi.org/10.17519/apiculture.2015.09.30.3.171]

- Han, S.H., Y.S. Choi, and M.L. Lee, (2011), Development of highly specific quantative real-time PCR method for the detection of Sacbrood virus in Korean honeybees, Apis cerana, J. Apiculture, 26(3), p233-240.
-
Kang, M.H., I.W. Kim, D.W. Lee, M.S. Yoo, S.H. Han, B.S. Yoon, (2011), Development of a rapid detection method to detect tdh gene in Vibrio parahaemolyticus using 2-step ultrarapid real-time polymerase chain reaction, Diagnostic Microbiology and Infectious Disease, 69, p21-29.
[https://doi.org/10.1016/j.diagmicrobio.2010.08.020]

-
Kim, I.W., M.H. Kang, S.H. Kwon, S.H. Cho, B.S. Yoo, S.H. Han, B.S. Yoon, (2010), Rapid detecton of virulence stx2 gene of Enterohemorrhagic Escherichia coli using twostep ultra-rapid real-time PCR, Biotechnology Letters, 32, p681-688.
[https://doi.org/10.1007/s10529-010-0205-0]

- Lee, D.W., E.H. Kim, I.U. Kim, M.S. Yoo, BS. Yoon, (2007), Ultra-rapid two-step real-time PCR for the detection of human immunodeficiency virus (HIV), Kor J Microbiol, 43, p264-272.
-
Ma, M., C. Ma, M. Li, S. Wang, S. Yang, S, Wang, (2011), Loop-mediated isothermal amplification for rapid detection of Chinese sacbrood virus, Journal of virological methods, 176(1), p115-119.
[https://doi.org/10.1016/j.jviromet.2011.05.028]