다중PCR증폭과 특이 DNA-chip을 활용한 꿀벌 주요 11종 병원체의 검출법 개발
초록
꿀벌의 주요 병원체 11종에 대한, 다중 PCR 및 특이 DNA-chip을 이용한 새로운 검출법을 개발하였다. 주요 병원체 11종은 Paenibacillus larvae와 Melissococcus plutonius 세균 2종, Aspergillus flavus, Ascosphaera apis, Nosema ceranae 진균 3종, Black queen cell virus, Chronic bee paralysis virus, Deformed wing virus, Israeli acute paralysis virus, Sacbrood virus, Korean sacbrood virus 6종이었다.
다중 PCR 및 특이 DNA-chip의 개발은, 유충 또는 성충 질병시료에서 분리된 DNA와 cDNA를 바로 사용할 수 있도록 최적화시켰으며, DNA-Chip 분석에 의하여, 11 병원체 중 어느 것이 시료 중 우점하였는지 쉽게 인식할 수 있도록 하였다. 본 시험법이 꿀벌질병 진단의 확정시험법으로 유용할 것을 기대한다.
Abstract
Against 11 major honeybee pathogens, new detection-method using multiplex PCR and specific DNA-chip were developed. 11 major pathogens were 2 species of bacteria, 3 of fungi and 6 of viruses, including Paenibacillus larvae, Melissococcus plutonius, Aspergillus flavus, Ascosphaera apis, Nosema ceranae, Black queen cell virus, Chronic bee paralysis virus, Deformed wing virus, Israeli acute paralysis virus, Sacbrood virus, and Korean sacbrood virus. Development of multiplex PCR and DNA-chip were well optimized just using DNA and cDNA isolated from a infected sample, such as larva or adult honeybee. Based on analysis of DNA chip, it could be easy recognized, which are dominant among 11 pathogens in test sample. It might be useful as confirmation-test for diagnosis of honeybee diseases.
Keywords:
Multiplex PCR, DNA chip, Honey bee diseases, Qualitative detection, Confirmation test서 론
꿀벌의 감염성 병원체는 바이러스, 세균, 진균 모두에서 총 35종이 보고되어 있으며(Chen and Siede, 2007; Runckel et al., 2011; Li et al., 2014), 이중 국내에서는 15종 병원체의 존재가 확인된 바 있다. 본 연구는 국내 양봉에 가장 많은 피해를 준 것으로 판단된 11종의 주요 병원체를 대상으로, 한 번의 검사로써 어느 병원체가 시료 중 우점적으로 존재하는가를 판정하고자 하였다.
11종 꿀벌 주요 병원체는, 오랜 기간 국내 양봉에 큰 피해를 입혀온 미국부저병(American Foulbrood disease; AFB)의 원인균, Paenibacillus larvae, 유럽부저병(European Foulbrood disease; EFB)의 원인균 Melissococcus plutonius의 2대 세균성 병원균을 포함하며, 백묵병(Chalkbrood; CB)의 병원체 Ascosphaera apis, 석고병(Stonebrood; SB)의 주요 병원체인 Aspergillus flavus 그리고 근래 맹위를 떨치고 있는 노제마병의 원인균 Nosema ceranae의 진균 3종도 대상으로 하였다. 또한 검출이 어려운 이유로 그 피해가 추정되어 왔던 바이러스성 질병에 대하여 BQCV(Black Queen Cell Virus), CBPV(Chronic Bee Paralysis Virus) DWV(Deformed Wing Virus; 날개불구병), IAPV(Israeli Acute Paralysis Virus), SBV(Sacbrood Virus), kSBV(Korean Sacbrood Virus; 한국형 낭충봉아부패병)의 6종을 검출대상에 포함하였다.
이중 kSBV는 국내 동양종 꿀벌에서 발견된 SBV의 일종으로 2010년 이래 국내 토종벌의 75% 이상을 폐사시킨 원인체로 주목되고 있으며(Choi et al., 2010). 또한 진균으로 분류되는 Nosema ceranae는 종래의 노제마병 원인체인 Nosema apis를 대체할 정도로 맹위를 떨치고 있는 기생성 병원체이기도 하다.
상기 11종의 꿀벌 병원체들에 대한 특이 유전자 검사법은, 주로 PCR을 기반으로 수많은 개별검사법이 개발되었으며, 다양한 검사 목적을 충족시키기 위하여 다양한 방식의 유전자 검사법이 개발되었다(Carletto et al., 2010; 임등, 2016).
우선, 개별병원체의 정량적 검출을 위하여, 다수의 정량 PCR(quantitative PCR)법이 보고되었으며(Han et al., 2011; Bu et al., 2005), 특정 병원체의 빠른 검출을 위하여 초고속 PCR(Ultra-rapid PCR)법이 개발되었고(Han et al., 2008; Yoo et al., 2011, 2012; Lim and Yoon, 2013), 현장신속검사를 위하여 Ultra-fast PCR (UF-PCR)법 및 LAMP (Loop-mediated amplification)법이 보고되었다(왕 등, 2016; Lee et al., 2016). 또한 보다 쉬운 병원체 검사를 위하여, 각 꿀벌 병원체에 대한 면역크로마토그래피법(Immunochromatography)이 개발되고 있으며, 이는 가까운 미래에 상업적 생산이 시작될 것으로 파악되고 있다(Unpublished communication).
앞에 거론된 병원체의 검사법들은, 모두 특정 병원체의 존재여부 및 그 양을 판정하는 방법이기에, 꿀벌 질병시료로부터 원인체가 무엇인지를 밝혀내기 위하여, 많은 종류의 특정 병원체에 대한 검출법들을 하나씩 병렬 수행하여야 하였다. 꿀벌질병의 진단분야에서 어느 병원체인가를 알아낼 수 있는 종합적 검사법(정성검사)에 가장 접근한 것은 multiplex PCR 검사법이며, Sguazza 등(2013)은 꿀벌 병원성 바이러스 6종 ABPV, BQCV, CBPV, DWV, IAPV, SBV의 개별 존재여 부를 판정할 수 있는 multiplex PCR검사법을 제시하였다. 그러나 이 multiplex PCR검사법은 아가로오스 전기영동 상에서 해당 PCR 산물의 크기로 판정하여야만 하고, 또한 꿀벌 바이러스 6종만을 대상으로 한다는 점에서 응용에 제한적인 면이 있었다.
한편, DNA chip은 합성 oligonucleotide(probe)들을 각각 공유 결합으로 slide glass 표면에 미세배열(microarray)로 in situ 탑재(spotting)시켜, 상보적 염기서열을 가진 특이 DNA가 각 probe에 DNA:DNA hybridization 되게 하는 실험법이다(Southern et al., 1992). 근래, 특이적으로 증폭시킨 PCR 산물들을, 별도의 특이 염기서열을 가진 probe들을 탑재한 DNA-chip에 hybridization시킴으로써, 염기서열 특이성이 PCR과 hybridization으로 배가 시킨 실험법들이 개발되었고, 높은 특이성과 이중 확인의 장점, probe의 종수에 제한이 거의 없다는 점 등에 고무되어, 여러 병원체의 종합적 검사(정성)에 다양하게 응용되게 되었다(Sarshar et al., 2015; Li, 2016). 그러나, 꿀벌 질병에 대하여 통합적, 정성적 병원체 검사를 수행할 수 있는 DNA chip에 관한 개발은 아직까지 보고된 바가 없었다.
따라서, 본 연구는 특정 병원체의 각개 검출에 기반을 둔 현재의 꿀벌 질병 진단법을, 통합적, 정성적 병원체 검사로 통합시키기 위하여, 11종 꿀벌 주요병원체의 개별 검출을 위한 multiplex PCR법을 개발하고, 각 병원체 특이 PCR산물을, 각 병원체 특이 probe를 탑재시킨 DNA-chip으로 판정하는 일련의 새로운 검사법을 새로이 개발하고자 하였다.
재료 및 방법
시료 수집과 RNA 및 DNA 확보
꿀벌 질병 시료는 2016년 5월 경기대 양봉장에서 수집된 kSBV 감염체를 사용하였다. 치사되어 일벌에 의하여 소문 밖으로 배출된 유충을 수집하였으며, 이를 MagNa lyser(Roche, Korea)로 분쇄하고 RNA iso plus(Takara, Japan)를 이용하여 total RNA를 순수분리하였다. 분리된 RNA는 spectrophotometer에 의한 정량 후, 그 중 1 μg을 200 unit M-MLV reverse transcriptase(Bioneer, Korea)를 이용하여 cDNA 합성을 수행하였고, 이 cDNA와 total RNA들은 -70°C에서 보관하며 실험에 사용하였다.
본 연구에서 PCR 주형으로 사용한 재조합 DNA체들은 각 꿀벌 병원체에서 유래된 특이 염기서열을 포함하고 있는 것이며, 경기대 유전학실험실에 보관된 것들이다. 본 연구를 위해 공여 받은 재조합 DNA들의 목록 및 각 유전자 정보, 문헌 정보들은 다음 표와 같다(Table 1).
꿀벌 병원체 11 종에 대한 multiplex PCR 설계 및 제작
꿀벌 병원체 11종에 대한 특이적 PCR primer 11쌍은, 22개 primer들에 의한 multiplex PCR을 가능하게 하기 위하여, 또한 생성된 특이 PCR산물이 DNA-chip에 탑 재 시 킨 probe들 (oligonucleotides)과 우수한 hybridization이 가능하게 하기 위하여, Table 1의 재조합체에 탑재된 각 병원체에 특이적 염기서열을 근거하여 설계하였다. 각 primer의 주문제작 후, multiplex PCR의 적합성을 Real-Time PCR을 통하여 평가하였으며, 부적합한 primer에 대하여 퇴출, 재설계, 재선발하였고, 최종적으로 DNA-chip에 탑재시킨 probe들과의 특이성을 분석하여 재설계 및 재선발을 반복하였다. DNA-chip의 probe oligonucleotide들과 hybridization 결과를 판정하기 위하여 PCR primer 11쌍은 각 primer의 5´말단에 형광원 Cy3가 결합된 형태로 주문 제작되었으며(Bioneer, Korea), Primer쌍들의 평가에 사용된 Real-Time PCR은 SyberGreen을 형광원으로 사용하였다. Real-Time PCR 기기는 Exicycler™96(Bioneer, Korea)을 사용하였다.
꿀벌 병원체 11종에 대한 22-multiplex PCR의 최적화
11종 각 병원체 특이 DNA 주형은 각 재조합체를 사용하였으며, 실험에 사용된 평균 DNA copies는 105개로 하였다. 먼저, 각 병원체 특이 DNA 주형을 대상으로 특이 PCR primer쌍을 사용하여 각기 혼성화(annealing) 온도구배 PCR(temperature-gradient PCR)을 수행하여, 최적 혼성화 온도 및 가능 온도 범위를 구하였으며, 이를 기준으로 22-multiplex PCR의 최적 혼성화 온도를 설정하였다. PCR 반응액은 일반적으로 20 μl를 기준으로 하였으며, 사용된 PCR premix는 Greenstar master mix (Bioneer, Korea)이었다.
11종 DNA 주형에 대한 22-multiplex PCR은 PCR cycle에 따른 형광강도의 증가 및 각 병원체 특이 증폭 산물의 양에 따라 평가하였으며, 이에 따라 22-multiplex PCR의 조성 및 조건을 최적화하였다.
최적화된 22-multiplex PCR의 조건은 22-Asy-primer mix를 사용하고, 초기 변성 단계 94°C 5분 후, 변성 단계 94°C 30초, 중합 단계 52°C 30초, 신장 단계 72°C 30초씩 진행하여 총 35 cycles 반복하는 것이었다.
22-Asy-primer mix는, 특이 PCR산물과 DNA-chip에 탑재된 probe oligonucleotide들과 hybridization을 증진시키기 위하여, 비대칭(asymetric) 양의 PCR primer 쌍들로 primer-mixture를 조성한 것이다. 즉, 각 병원체에 대한 특이 primer쌍들을 1:4, 4:1 또는 1:2(forward : reverse primer의 양)로 맞추어 조성한 것으로 hybridization 후 각 SBR(Spot/Background Ratio)값을 토대로 최적비율을 구하고, 이 결과를 토대로 최적 22-asy-primr mix를 조성하였다.
DNA chip의 oligonucleotide probe 설계 및 제작
DNA-chip에 탑재시킬 oligonucleotide probe들은 각 병원체 특이 PCR산물의 내부 염기서열을 사용하였으며, 병원체별로 특이적 hybridization이 가능하도록 설계하여 주문 제작되었다(Bioneer, Korea). 특이 oligonucleotide probe들은 DNA-chip, K-CAP™에 탑재되어 hybridization 및 평가과정을 거쳤으며, 이 역시 multiplex-PCR에 의한 특이 PCR 산물들과 특이적 hybridization, 그리고 비특이적 hybridization을 평가하여 재설계, 교체 탑재의 과정을 반복하였다. 설계된 DNA-chip의 제작은 K-MAC(Korea)에서 담당하였으며, hybridization의 평가에서도 K-MAC의 K-SCAN-CAP™ scanner(K-MAC, Korea)와 관련 software program(K-MAC, Korea)을 사용하였다.
11종 꿀벌 주요 병원체 검출을 위한 DNA chip 제작
K-CAP™은 K-Mac사에서 제작된 소형 DNA-chip이며, 외형상 200μl의 원심분리관을 닫을 수 있는 마개에 유리봉이 관통된 형상이다. PCR용액에 원심분리 관내 유리봉이 잠기게 되며, 그 유리봉 단면이 probe oligonucleotide들을미세배열시킬수있는면이다(Fig. 1).
꿀벌 질병 원인균 11종에 특이적으로 합성된 probe oligonucleotide들은, 고정액에 각 probe가 20 pmole/μl 또는 40 pmole/μl가 되도록 농도를 조정하였으며, 이 probe 용액은 sciFLEXARRAYER S11(Scienion, Germany)을 사용하여, aldehyde기로 표면처리를 한 K-CAP™의 유리봉 단면에 위치 특이적으로 각 200 pl씩 분주(spotting)하였다. 분주된 probe는 직경 200μm의 spot 형태가 되었으며, 중심간 거리(center to center; CTC)는 180μm가 되도록 조정되었다. Probe의 분주 후 25°C, 70%의 습도 하에서 3시간 동안 K-CAP™ 표면의 aldehyde기와 probe의 amine기와의 결합을 유도하였으며, 반응 종료 후 Prehybridization Buffer(2x SSPE, 0.2% SDS)를 사용하여 표면에 결합하지 못한 여분의 probe들을 제거하였다(K-MAC, Korea).
Hybridization 및 Washing 최적화
22-Asy primer mix를 사용하여 각 병원체의 특이 염기서열을 주형으로 하여 multiplex PCR을 마친 후, 각 병원체의 특이적인 PCR 산물이 생성된 PCR용액들은 각기 혼성화(hybridization)에 사용되었다. 혼성화는 95°C 5분 변성 후, 52°C, 300 rpm의 진동(Thermo-mixer comfort, Eppendorf, Germany)하에 4시간 수행하는 것이 최적의 결과를 보였으며, 혼성화 후 세척(washing) 과정은 4× SSC buffer, 0.2× SSC buffer, D.W.순으로 진동 300 rpm과 42°C 온도 조건에서 5분씩 세척하는 것이 최적의 것으로 나타났다.
이 최적 혼성화 온도를 정하기 위하여 47°C, 52°C, 60°C를 비교하였으며, 세척과정의 온도 및 세척액 조성(2xSSC, 4xSSC)을 비교하였다. 결과의 평가는 증류수로 세척 후 각 spot의 SBR(Spot/Background Ratio)값을 기준으로 비교 분석하였다.
Multiplex PCR과 DNA-chip을 이용한 꿀벌 주요 병원체 11종에 대한 표준 검사법의 확립
11종의 병원체에 대한 표준 주형은 각 특이유전자 105 분자(약 2 pg)를 사용하는 것이며, 표준 primer set는 22종의 PCR primer가 비대칭적으로 혼합된 22-Asy-primer mix(100 pmole/reaction)와 표시자 primer PC1을 0.2 pmole/reaction을 사용하는 것이다. PCR 반응액은 총 20 μl이었으며, Greenstar master mix(Bioneer, Korea)를 기반으로 multiplex PCR을 수행하였다.
Multiplex PCR 조건은, 초기 변성 단계 94°C 5분 후, 변성 단계 94°C 30초, 중합 단계 52°C 30초, 신장 단계 72°C 30초씩 진행하여 총 40 cycles 반복하는 것이며, hybridization은 52°C, 300 rpm의 진동 하에서 4시간 진행하고, washing은 42°C, 300 rpm의 진동 하에서 각기 0.5 ml 4×SSC buffer, 5분, 0.5 ml 0.2×SSC buffer, 5분, 0.5 ml D.W. 5분으로 순차적 세척을 하고, 최종적으로 LightCycler® 480 Multi-well-plate 96, white(Roche, Korea)를 사용하여 3,500 rpm, 2분 간 원심분리하여 DNA-chip 표면을 건조시키는 것이다. Hybridization의 영상은 K-SCAN-CAP™으로 스캔하고, 산출된 SBR 값(Spot/Background Ratio)을 토대로 평가하는 것을 표준실험법으로 하였다.
현장 적용을 위한 kSBV에 감염된 검체를 사용한 확정 시험
kSBV에 감염된 동양종 꿀벌의 유충으로부터 total RNA를 순수분리하였고, 그 중 1μg을 사용하여 총 20μl cDNA 변환을 수행한 후, 그 중 1μl을 주형으로 사용하여 22-Asy primer mix를 사용한 multiplex PCR 및 DNA-chip에 의한 hybridization을 수행하였다. 모든 실험은 표준검사법에 준하여 실시하였다.
한편, 검증을 위하여, DNA-chip실험에서 사용된 cDNA-target의 분자수를 kSBV-specific Real-Time PCR을 사용하여 target 분자수를 측정하였다. Real-Time PCR에서 사용된 primer 쌍은 22-Asy primer mix에 포함된 kSB-PCR-F1/R1이였으며, Real-Time PCR의 조성은 5mM kSB-PCR-F1/R1와 PCR premix인 Greenstar master mix(Bioneer, Korea)로 하였으며, multiplex PCR의 경우와 같이 주형은 1μl kSBV cDNA로 진행하였다. Real-Time PCR의 조건은 초기 변성 단계 94°C 5분 진행 후, 변성 단계 94°C 30초, 중합 단계 52°C 30초, 신장 단계 72°C 30초씩 총 40 cycles 반복하였다. 한편 정량을 위하여 pGEM-kSBV-VP1 재조합DNA를 target로 하였으며, 이를 초기주형양 105 분자, 104 분자, 103 분자 수 순으로 단계별 희석을 하여, 정량을 위한 회귀직선을 구하였다.
결 과
꿀벌 11종 병원체에 대한 특이 PCR 및 multiplex PCR 최적화
설계된 각 병원체에 대한 특이적 PCR 증폭을 확인하기 위하여, 그리고 multiplex PCR에서 최적 annealing 온도를 구하기 위하여, 각 특이 재조합 DNA 105분자를 주형으로, 각 특이 primer를 반응 당 5 pmole씩 사용하여, 각기 계산된 융점(temperature of mid-point; Tm) 값을 중심으로 온도구배PCR을 시행하였다(Fig. 2).
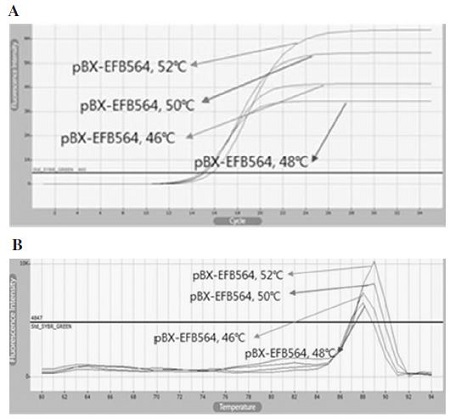
The amplification of M. plutonius (EFB)-specific DNA with specific primers using annealing temperature-gradient PCRs. (A) Fluorescence curves. M. plutonius-specific PCR products were differently amplified depend on annealing temperature, 46°C, 48°C, 50°C and 52. (B) Melting-temperature analysis. The Tms of M. plutonius-specific PCR products were estimated in range of 87.5~89.0°C
11종 병원체에 대한 특이 primer 쌍들은 각 PCR에서 각기 특이적인 증폭을 보여주었으며, 융점분석(melting temperature analysis)에서 각기 고유의 Tm값을 나타내었다. 11종 특이 온도구배 PCR의 결과를 바탕으로, 가장 다수의 병원체들에서 높은 증폭양을 보이는 PCR annealing온도가 52°C인 것으로 집계되었기에, 이를 multiplex PCR의 annealing 온도로 선정하였다(결과 미제시).
꿀벌 병원체 11종에 대한 specific multiplex-PCR은, 11쌍(22개)의 특이 primer들을 각 1 pmole씩 혼합하여 반응 당 총 22 pmole의 primer들 사용하고, 각 특이 DNA 105분자를 주형으로 최적화 하였다.
그러나, 각 primer들의 특이 결합친화도(binding affinity)가 서로 상이한 이유로, 11종 특이 PCR product들을 균일한 수준으로 생산할 수 없었기에, 특이 primer의 재선발 및 교체, primer간 혼합비의 변경 등을 통하여 가장 유사한 양의 특이 PCR산물을 각각 만들 수 있고, 동시에 DNA-chip상의 특이 probe들과 반응성이 좋은 조건을 만들 수 있는 primer-mix를 재구성하여야 하였다. 최종적으로 DNA-chip과의 반응성에서도 가장 우수한 multiplex PCR용 premix를 조성할 수 있었으며, 이를 22-Asy primer mix(100 pmole/ each PCR)라 명명하였다(Table 3).
각 특이 DNA 105분자를 주형으로, 22-Asy primer mix를 사용한 최적화된 multiplex PCR은, 각 특이 DNA를 정확히 증폭시켰으며, 9종의 target DNA(105분자)에 대하여 각기 Ct(Threshold cycles)값이 20-23 cycles로 나타났다(단, kSBV와 IAPV의 Ct값은 28 cycles). 그러나, 11종 모두의 최종 형광값(final fluorescence intensity)은 3.3~5.0 K의 범위에 들어, 이후의 실험인 hybridization에서 충분한 양의 분자가 생산된 것이라 판단하였다(Fig. 3).
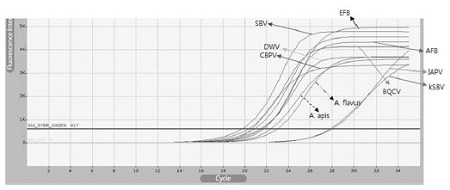
Multiplex PCR using 22-Asy primer mix with each pathogen-specific DNA. As template, each 105 molecules of pathogen-specific DNA were added. 22-Asy primer mix were used to each PCR. The Ct values of BQCV, CBPV, DWV, SBV, AFB, EFB, Ascosphaera apis, Aspergillus flavus and Nosema ceranae were estimated 20.0~23.0 cycles, except 28.0 cycles of IAPV and kSBV. All final fluorescence intensities were pointed on 3.3~5.0 K.
꿀벌 질병 진단용 DNA chip 제작
본 연구에서 사용된 DNA chip은 K-CAP™(K-MAC, Korea)으로, 유리봉에 실리콘 라드가 결합되어 있는 형태로, 0.2ml PCR tube에 그대로 장착하여 PCR과 DNA 혼성화 반응을 동시에 진행할 수 있도록 고안되었다. Tube내의 유리봉 끝은 DNA 미세배열에 사용되는 지름 2.0mm의 원형 유리면이며, 미세배열의 기본 설계는 원형 유리면에 가로 7개, 세로 7개, 총 49개의 oligonucleotide probe들을 결합시킨 spot들의 배열이며, 각 spot의 지름은 0.12mm로, spot 중심간 거리(Center to Center)는 0.18mm로 하였다. 각 probe oligonucleotede들을 별도 제작하여, 설계대로 probe들을 spotting하여 DNA-chip을 제작하였고, spot들의 위치를 영상으로 파악할 수 있도록 위치 표시(HC; hybridization control)의 위치를 미리 지정하였다.
사용된 spot의 수는 Set 1에서 19개, Set 2에서 19개, 위치 표시 4개로 총 42개이었으며, 11종 꿀벌 병원체에 특이한 probe 19종을, Set 1에서 20 pmole로 spotting하고, 같은 배열로 Set 2에서 40 pmole을 spotting하여, 한번의 DNA-chip실험에서 두 번의 반복 결과를 얻을 수 있도록 하였다(Fig. 4).

Positions of each spot containing oligonucleotide probes on DNA chip, K-CAPTM. 19 different oligonucleotide probes were mounted on spots in each set. Different quantities of oligonucleotide probes were used on spots in Set 1 (20 pmole) and on spots in Set 2 (40 pmole), 4 positions of Hybridization control (HC) were located.
11종 꿀벌 주요병원체에 대한 특이 oligonucleotide probe의 수는 총 19종으로 각 병원체의 유전자 변이성 등을 고려하여 복수의 probe들을 적용하였다. SBV와 kSBV의 경우 각 3종, 2종의 다른 특이 probe를 spotting 하였으며, DWV, IAPV, AFB, EFB의 경우 각 2종의 특이 probe를, BQCV, CBPV, Nosema cerana, Ascosphaera apis, Aspergillus fluvus들은 각 1종의 특이 probe를 탑재하였다(Table 4).
꿀벌병원체 11종의 특이 DNA에 대한 multiplex asymm etric PCR/ DNA-chip의 적용
Paenibacillus larvae(AFB), Melissococcus plutonius(EFB), Nosema ceranae, Ascosphaera apis, Aspergillus fluvus, SBV, kSBV, DWV, IAPV, BQCV, CBPV의 11종 꿀벌 병원체 특이 DNA를 주형으로 사용하여 표준조건에 의한 표준검사를 수행하였다. 표준 조건은 105 분자의 DNA를 PCR주형으로, 22-Asy primer mix를 사용하여 PCR을 진행하고, 이를 DNA-chip에 4시간 혼성화를 진행한 후 세척과정을 거쳐 영상분석하는 것이다.
22-Asy primer mix를 사용한, 각 병원체 특이 multiplex PCR 산물들은 바로 DNA-chip상의 특이 probe들과 hybridization시켰으며, 각 특이 PCR산물에 따라 특징적인 형상을 보여주었다(Fig. 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15).
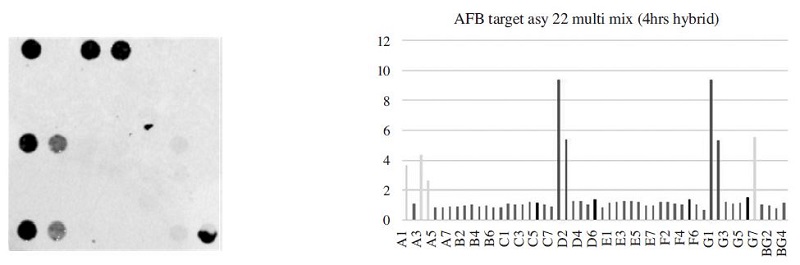
The fluorescence image and signal intensity of Paenibacillus larvae detection using 22-Asy primer mix and target DNA. The 4.94×105 molecules of AFB-specific DNA were used and 22-Asy primer mix and HC primer were added. The AFB specific D1 (probe set 1, 20 pmole) and G1 (Probe set 2, 40 pmole) were 9.36. D2 (probe set 1, 20 pmole) and G2 (probe set 2, 40 pmole) were 5.4 and 5.34. HC sites were A1, A3, A4, and G7.
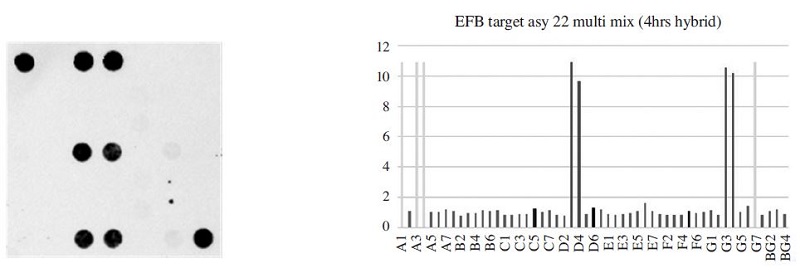
The fluorescence image and signal intensity of Melissococcus plutonius detection using 22-Asy primer mix and target DNA. The EFB specific D3 (probe set 1, 20 pmole), D4 (probe set 1, 20 pmole) were 10.95 and 9.68. The SBR value of G3 (probe set 2, 40 pmole), and G4 (probe set 2, 40 pmole) were 10.56, and 10.24.
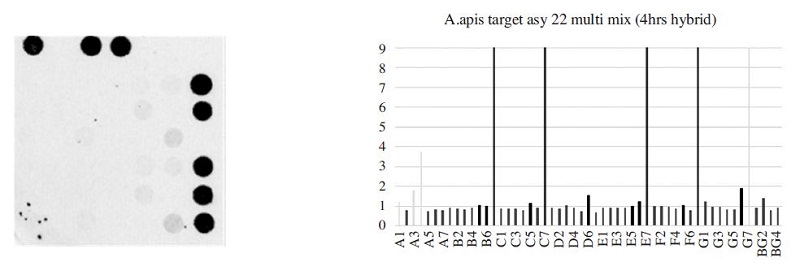
The fluorescence image and signal intensity of Ascosphaera apis detection using 22-Asy primer mix and target DNA. All B7, C7, E7 and F7 of Ascosphaera apis specific probes showed 10.99 on SBR value. B7 and C7 contain probe set 1 (20 pmole). E7 and F7 contain probe set 2 (40 pmole).
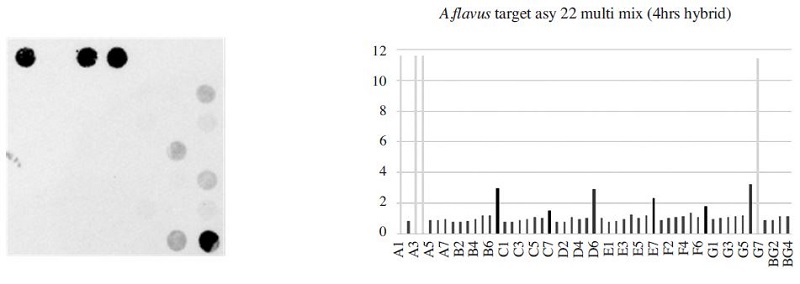
The fluorescence image and signal intensity of Aspergillus flavus detection using 22-Asy primer mix and target DNA. The Aspergillus flavus specific probe D6 (probe set 1, 20 pmole) and G6 (probe set 2, 40 pmole) were 2.91 and 3.23.
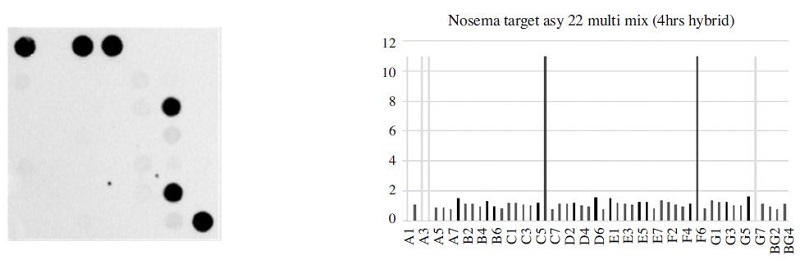
The fluorescence image and signal intensity of Nosema ceranae detection using 22-Asy primer mix and target DNA. Both specific probes C6 (probe set 1, 20 pmole) and F6 (probe set 2, 40 pmole) of Nosema ceranae were 10.98.
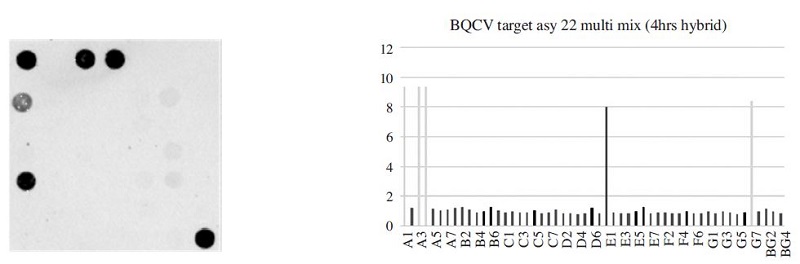
The fluorescence image and signal intensity of BQCV detection using 22-Asy primer mix and target DNA. The E1 (probe set 2, 40 pmole) of BQCV specific probe showed 7.97 on SBR value (B1 did not match up with grid in software program).
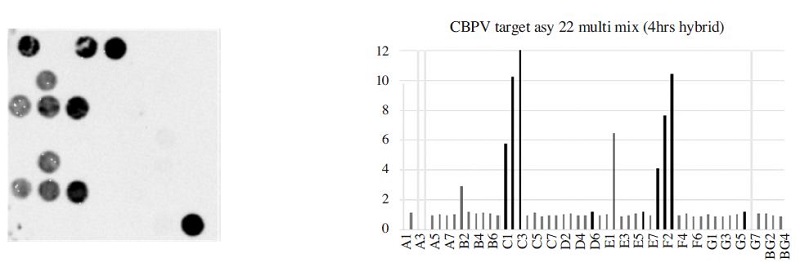
The fluorescence image and signal intensity of CBPV detection using 22-Asy primer mix and target DNA. The B2 (probe set 1, 20 pmole) and E2 (Set 2, 40 pmole) of CBPV specific probe showed 2.93 and 6.45.
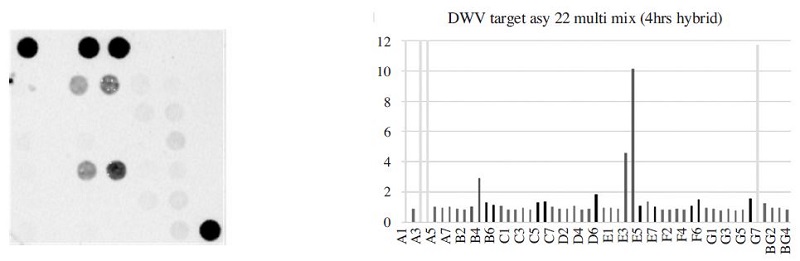
The fluorescence image and signal intensity of DWV detection using 22-Asy primer mix and target DNA. The E3 and E4 of DWV in probe set 2 (Set 2, 40 pmole) showed 4.56 and 10.13 (B3 and B4 did not match up with grid).
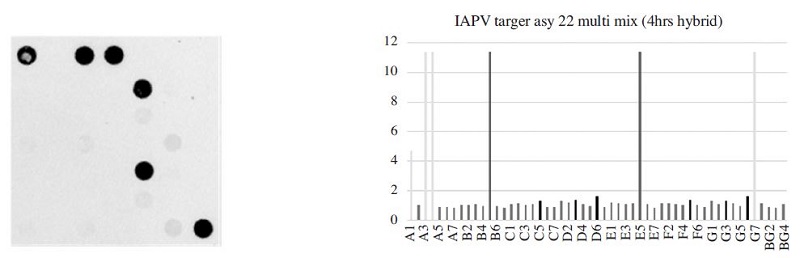
The fluorescence image and signal intensity of IAPV detection using 22-Asy primer mix and target DNA. Both B5 (probe set 1, 20 pmole) and E5 (probe set 2, 40 pmole) value of IAPV specific probes were 11.42.
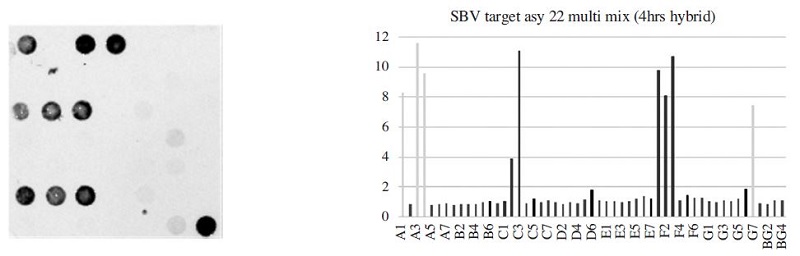
The fluorescence image and signal intensity of SBV detection using 22-Asy primer mix and target DNA. The SBV specific C2 and C3 in probe set 1 (20 pmole) were 3.88 and 11.1. F1, F2, and F3 in porbe set 2 were 9.79, 8.13, and 10.72.
DNA chip 진단의 현장 적용
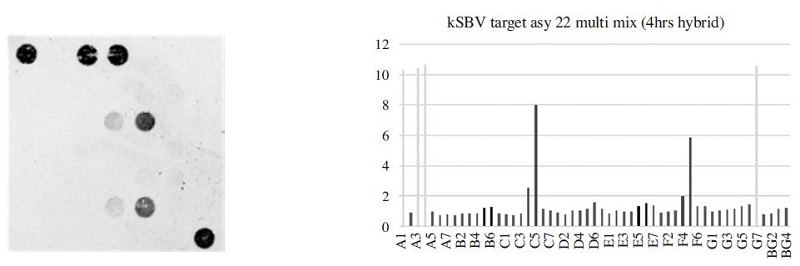
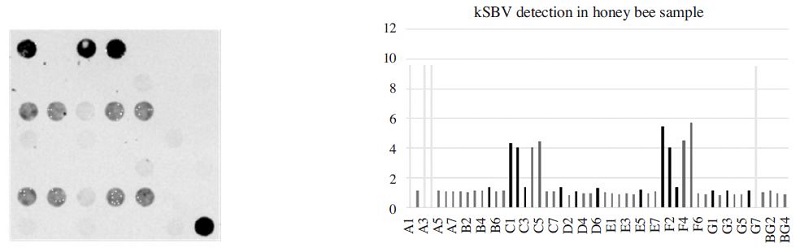
정립한 표준조건을 따라 kSBV 감염 시료의 표준 정성 검사를 수행하였다. 병성 시료는 토종벌(Apis cerana)의 유충으로, kSBV-특이 실시간PCR에 의하여 kSBV에 감염된 것이 확인된 것이다. 본 연구에서 개발된 다중 PCR에 사용된 주형의 양은, 별도 실험에서, 1.97×106 분자인 것으로 계산되었고, 22-Asy primer mix를 사용한 다중 PCR에서 kSBV 특이 증폭 산물이 관찰되었다. 이를 DNA-chip에 적용하여 4시간 혼성화를 진행한 후 1시간 4× SSC buffer 세척과정을거쳐DNA-chip의 영상을 분석하였다.
DNA-chip영상은 kSBV 특이 probe들의 위치들(C4, C5, F4, F5)에 이 다중 PCR 산물이 혼성화되었음 보여주었으며, 일부 SBV probe들(C1, C2, C3, F1, F2, F3)과의 비특이적 반응이 또한 관찰되었다. 그러나, 비특이적 반응 중 일부 SBV probe들(C3, F3)은 다른 SBV probe들(C1, C2, F1, F2)에 비하여 약한 반응을 보였고, 이는 SBV 병원체에 대한 DNA-chip 반응의 특성에 불일치한 것이었다. 따라서, 이 병성시료에 대한 DNA-chip에 의한 판정은, kSBV-특이 실시간 PCR에 의하여 확인된 바와 같이, kSBV 감염으로 판정할 수 있었다(Fig. 16).
고 찰
DNA-chip 검출 분석은 형광 염료가 표지된 multiplex PCR 산물이 DNA chip 표면의 oligonucleotide에 결합하며 특정 파장에서 나타나는 형광의 강도를 기준으로 한다. hybridization과 세척의 효율은 본 검출법의 중요한 요소이며 hybridization 온도, 세척액 농도, 세척 과정 중 진동 조건의 영향을 각각 확인하였다. 그 결과 hybridization 온도는 52°C, 세척액 조성은 4x SSC buffer, 진동 조건은 진동이 있는 것이 각기 유리한 결과를 보였다. 특히, asymmetric primer와 hybridization 시간은 hybridization의 효율 개선에 있어 상당한 영향을 나타내었다. 각 병원체의 asymmetric primer 결과를 종합하였을 때, 평균 3.82배 SBR값 증가율을 보였고 11종 병원체 검출에 적합한 22-Asy primer mix를 확정하였다. 또한 4시간 hybridization은 1시간 hybridization에 비해 최저 117.51%, 최대 391.25% 증가하였고, 이에 표준 hybridization 시간 조건을 4시간으로 제시하였으나, hybridization 2시간에서도 결과를 보여 실험자의 판단에 따라 2시간 또는 1시간의 hybridization에 의한 분석도 가능할 것으로 예상된다.
본 연구에서 DNA-chip은 oligonucleotide probe 선정, 퇴출, 재선정 및 형광 민감도 개선, 교차 반응 개선 과정 등을 거쳐 oligonucleotide들이 선정되었고 multiplex PCR 조건 및 실험표준조건이 확립되어 최종 개발되었다. 꿀벌 병원체 검출에 있어 대부분 특정 병원체의 정량적 검출법 연구가 넓게 진행되었고, 이제까지 여러 병원체를 통합적으로, 정성적으로 검사하는 방법은 몇 개의 multiplex PCR 혹은 특정 병원체 검출법을 종합하여 진행되었다. 그러나 본 검출법은 한 번의 검사로 꿀벌병원체 정성검사를 할 수 있는 최초의 multiplex PCR 및 DNA-chip검출법으로, 진단시료 주형에서 검출을 검증함으로 실제 진단법으로써 효용성을 보여주었다.
한편, CBPV 검출에서의 SBV probe의 비특이적인 반응(Fig. 11)과 Aspergillus flavus 검출에서 Asocsphaera apis probe와 비특이적인 반응(Fig. 8)이 관찰되었다. 현 시점에서 이들은 각기 단독 감염일 경우, CBPV의 양성 probe들(B2, E2)과 SBV의 양성 probe들(C1, C2, C3, F1, F2, F3)의 반응여부에 따라 각기 CBPV와 SBV의 검출을 구분할 수 있으며, Aspergillus flavus 검출의 경우, Ascosphaera apis의 probe들 중 B7, E7에서 강한 반응, C7과 F7에서 약한 반응의 특징을 이용하면 Aspergillus flavus와 Ascosphaera apis의 검출을 각기 구분할 수 있을 것이다. 그러나 양자 모두 교차반응을 나타내는 두 가지 병원체가 혼합 감염되었을 경우 이를 구분할 수 없을 것이다. 이의 근원적 개선을 위하여, 새로운 특이 probe의 도입이 향후 개선점으로 판단된다.
또한, reverse transcription을 multiplex PCR과 DNA-chip에 도입하여 One-step DNA chip 검출을 추구하는 것은 실험자의 편의를 증대하는 하나의 플랫폼이 될 것이며 reverse transcription, multiplex PCR, hybridization까지 한 번에 진행함으로써 오염의 위험을 줄일 수 있을 것이다.
본 연구와 같은 꿀벌 병원체 multiplex PCR 및 DNA-chip 검출법은 계속적인 개발을 통해 진화되어야 할 것이며, 이는 꿀벌의 병원체에 국한되지 않고 다양한 분야의 병원체 검출 등으로 연구가 확장되기를 아울러 기대한다.
Acknowledgments
본 연구는 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 수출전략기술개발사업(115067-02), 첨단생산기술개발사업(115058-02, 115102-03), 농생명산업기술개발사업(312027-03)과 2016학년도 경기대학교 대학원 연구원장학생 장학금 지원에 의하여 수행되었습니다.
References
- 왕지희, 민상현, 임수진, 윤병수, (2016), 꿀벌의 석고병, 백묵병 현장 진단을 위한 신속 Ascosphaera apis 및 Aspergillus flavus 검출법 개발, 한국양봉학회지, 31, p31-39.
- 이주성, 루옹티홍장, 윤병수, (2015), 꿀벌 병원성 바이러스 Deformed Wing Virus의 4종 특이 단백질들의 대량생산, 한국양봉학회지, 30, p359-372.
- 이혜민, 하정순, 조용호, 남성희, 윤병수, (2004), 꿀벌 진균성 질병의 신속 확인을 위한 Ascosphera apis, Aspergillus flavus 의 PCR 검출법, 19, p139-148.
- 임수진, Giang Thi Huong Luong, 민상현, 왕지희, 윤병수, (2016), 역전사 실시간 Recombinase Polymerase Amplification (RT/RT RPA)에 의한 꿀벌 Black Queen Cell Virus의 신속 검출, 한국양봉학회지, 31, p41-50.
- 최용수, 김혜경, 이명렬, 이만영, 이광길, (2008), Minus-strandspecific RT-PCR에 의한 Chronic Bee Paralysis Virus(CBPV) 진단, 한국양봉학회지, 23, p119-126.
- 하정순, 이혜민, 김동수, 임운규, 윤병수, (2005), 유럽부저병(European Foulbrood)의 신속 확인을 위한 Melissococcus Pluton의 PCR Detection Method, 한국양봉학회지, 20, p9-18.
-
Bu, R., R. K. Sathiapalan, M. M. Ibrahim, I. Al-Mohsen, E. Almodavar, M. I. Gutierrez, and K. Bhatia, (2005), Monochrome LightCycler PCR assay for detection and quantification of five common species of Candida and Aspergillus, J. Medical Microbiology, 54, p243-248.
[https://doi.org/10.1099/jmm.0.45856-0]

- Carletto, J., A. Gauthier, J. Regnault, P. Blanchard, F. Schurr, and M. Ribière-Chabert, (2010), Detection of main honey bee pathogens by multiplex PCR, EuroReference, No. 4, ER04-10R02.
-
Chen, Y.P., and R. Siede, (2007), Honey Bee Viruses, Adv. Virus Research, 70, p33-80.
[https://doi.org/10.1016/S0065-3527(07)70002-7]

- Choi, Y.S., M.Y. Lee, I.P. Hong, N.S. Kim, H.K. Kim, K.G. Lee, and M.L. Lee, (2010), Occurrence of Sacbrood Virus in Korean Apiaries from Apis cerana (Hymenoptera: Apidae), J. Apiculture, 25, p187-191.
- Giang, T. H. L., H.Y. Lim, and B.S. Yoon, (2014), Over-expression and Purification of RNA Dependent RNA Polymerase from Black queen cell virus in Honeybee, J. Apiculture, 29, p173-180.
-
Han, S.H., D.B. Lee, D.W. Lee, E.H. Kim, and B.S. Yoon, (2008), Ultra-rapid real-time PCR for the detection of Paenibacillus larvae, the causative agent of American Foulbrood (AFB), J. Invertebrate Pathology, 99, p8-13.
[https://doi.org/10.1016/j.jip.2008.04.010]

- Han, S.H., Y.S. Choi, and M.L. Lee, (2011), Development of Highly Specific Quantative Real-Time PCR Method for the Detection of Sacbrood Virus in Korean Honeybees, Apis cerana, J. Apiculture, 26, p233-240.
- Kim, C.N.T., M.S. Yoo, I.W. Kim, M.H. Kang, S.H. Han, and B.S. Yoon, (2008), Development of PCR Detection Method for Sacbrood Virus in Honeybee (Apis mellifera L.), J. Apiculture, 23, p177-184.
-
Lee, J.S., S.J. Yong, H.Y. Lim, and B.S. Yoon, (2015), A Simple and Sensitive Gene-Based Diagnosis of Aspergillus flavus by Loop-Mediated Isothermal Amplification in Honeybee, J. Apiculture, 30, p53-59.
[https://doi.org/10.17519/apiculture.2015.04.30.1.53]

-
Lee, J.S., T.H.L. Giang, and B.S. Yoon, (2016), Development of infield-diagnosis of Aspergillus flavus by Loop-Mediated Isothermal Amplification in Honeybee, J. Apiculture, 31, p25-30.
[https://doi.org/10.17519/apiculture.2016.04.31.1.25]

-
Li, J. L., R. S. Cornman, J. D. Evans, J. S. Pettis, Y. Zhao, C. Murphy, W. J. Peng, J. Wu, M. Hamilton, H. F. Boncristiani Jr, L. Zhou, J. Hammond, and Y. P. Chen, (2014), Systemic spread and propagation of a plantpathogenic virus in European honeybees, Apis mellifera, MBio, 5, pe00898-13.
[https://doi.org/10.1128/mBio.00898-13]

-
Li, Y. J., (2016), Establishment and Application of a Visual DNA Microarray for the Detection of Food-borne Pathogens, J. Analytical Sciences, 32.2, p215-218.
[https://doi.org/10.2116/analsci.32.215]

- Lim, H.Y., and B.S. Yoon, (2013), Rapid and Sensitive detection of Deformed Wing Virus (DWV) in Honeybee using Ultrar-apid Real-time PCR, J. Apiculture, 28, p121-129.
-
Runckel, C., M. L. Flenniken, J. C. Engel, J. G. Ruby, D. Ganem, R. Andino, and J. L. DeRisi, (2011), Temporal analysis of the honey bee microbiome reveals four novel viruses and seasonal prevalence of known viruses, Nosema, and Crithidia, PLOS one, 6, pe20656.
[https://doi.org/10.1371/journal.pone.0020656]

-
Sarshar, M., N. Shahrokhi, R. Ranjbar, and C. Mammina, (2015), Simultaneous Detection of Escherichia coli, Salmonella enterica, Listeria monocytogenes and Bacillus cereus by Oligonucleotide Microarray, Int. J. Enteric Pathogens, 3, pe30187.
[https://doi.org/10.17795/ijep30187]

-
Sguazza, G. H., F. J. Reynaldi, C. M. Galosi, M. R. Pecoraro, (2013), Simultaneous detection of bee viruses by multiplex PCR, J. Virological Methods, 194.1, p102-106.
[https://doi.org/10.1016/j.jviromet.2013.08.003]

-
Southern, E. M., U. Maskos, and J. K. Elder, (1992), Analyzing and comparing nucleic acid sequences by hybridization to arrays of oligonucleotides: evaluation using experimental models, Genomics, 13, p1008-1017.
[https://doi.org/10.1016/0888-7543(92)90014-J]

- Yoo, M.S., S.H. Han, and B.S. Yoon, (2011), Development of Ultra-Rapid Real-Time PCR Method for Detection of Black Queen Cell Virus, J. Apiculture, 26, p203-208.
-
Yoo, M.S., C.N.T. Kim, P.V. Nguyen, S.H. Han, S.H. Kwon, and B.S. Yoon, (2012), Rapid detection of sacbrood virus in honeybee using ultra-rapid real-time polymerase chain reaction, J. Virological Methods, 179, p195-200.
[https://doi.org/10.1016/j.jviromet.2011.10.014]