꿀벌 병원성 바이러스의 정량적 검출을 위한 정량 실시간 재조합효소-중합효소 증폭법의 개발
초록
재조합효소-중합효소 증폭법(RPA)은 등온에서 매우 빠른 반응속도를 보이는 유전자 증폭법으로 근래 여러 병원체의 검출법에 폭넓게 적용되고 있다. 본연구는 kSBV의 빠른 검색을 위하여 kSBV 특이 실시간 재조합효소-중합효소 증폭법을 새로이 개발하였으며, 특이 cDNA로부터 2분 46초만에 kSBV특이 유전자의 존재를 확인할 수 있었다. 또한, 이를 바탕으로 목적 유전자의 정량이 가능한, 보편적 응용이 가능한 정량 실시간 재조합효소-중합효소 증폭법(quantitative real-time RPA)을 제안하였으며, 이 정량법이 현장시료에서 유용함을 입증하였다. 본 실험법은 꿀벌 질병체의 정량 검출 뿐 아니라, 일반 병원체의 정량 검출에서도 응용되기를 기대한다.
Abstract
Recombinase polymerase amplification (RPA) is a specific DNA amplification method which shows high speed in isothermal condition and it has been widely applied to the detection of various pathogens. In this study, kSBV specific real-time recombinase polymerase amplification method was newly developed for rapid detection of kSBV. The existence of kSBV specific gene could be detected using total cDNA within 2 min 46 sec using this method. Based on this method, we proposed the quantitative real-time recombinase polymerase amplification which was able to quantify the target gene and to be applied universally. Moreover, this assay was proved to be useful to detect the pathogen from real samples. It is expected that this assay will be applied as quantitative detection method for general pathogens as well as honeybee pathogens.
Keywords:
Real-time recombinase polymerase amplification, Quantitative real-time RPA, Korean sacbrood virus, Quantitative detection method서 론
2010년 이래 한국형 낭충봉아부패병 바이러스(korean Sacbrood virus; kSBV)는 국내의 토봉 (Apis ceranae)을 95%이상 폐사시킨 것으로 알려져 있으나, 같은 기간 국내 서양종 꿀벌(Apis mellifera)은 이에 대한 큰 피해를 받지 않아, 다행히 국내 양봉의 치명적 몰락을 피할 수 있었다. kSBV의 발생은 꿀벌의 병원성 바이러스에 대한 제어방법을 진지하게 고민하게 하였으며, 우선, 특정 바이러스의 검출법은 해당 바이러스에 대한 정보 축적의 가장 기초적인 도구이기에, 간편하고 정확한 그리고 정량적 검출이 가능한 검출법에 대한 개발 보고는 고무적인 것이라 할 것이다(유 등, 2009, 2010; 이등, 2011; Han et al., 2011).
현재 꿀벌바이러스 검출법은 polymerase chain reaction(PCR)법을 기반으로 한 다양한 유전자 검사법이 사용되고 있으며, 실시간 정량 PCR(real-time quantitative PCR; qRT-PCR)법이 민감성과 정확성의 면에서 거의 표준적 실험법으로 인식 되고 있다. 그러나 이 실험법은 고가의 장비가 필요하여 현장성에서 약점을 보이고 있으며, 검사 시간이 오래 걸리는 단점을 가지고 있다.
한편, Recombinase polymerase amplification(RPA)는 PCR법과 다르게 등온 조건에서 특정 유전자의 염기서열을 증폭시키고, 보다 빠르게 유전자 증폭시킬 수 있는 실험법으로 주목을 받고 있다. 즉, RPA법은 recombinase인 uvsX와 co-factor, oligo-nucleotide primer와 결합하는 uvsY, DNA 신장하는 Sau polymerase를 사용하여 37°C 등온에서 반응을 진행하며, 총 반응시간은 40분 내외가 추천되고 있다(Piepenburg et al., 2006). 현재 RPA 방법에 의한 다양한 병원체의 검출법들이 계속 보고되고 있으며, 우수한 유전자 진단법으로 점차 적용범위를 넓혀 나아가고 있다.
병원체 검출법으로써 RPA법의 강점은 신속성과 37°C 등온반응이라는 현장적용성이라 할 것이다. Xia 등(2014)은 감염성 세포괴사 바이러스(IHHNV; infectious hypodermal and hematopoietic necrosis virus)를 7분 이내에 검출한 것을 보여주었으며, Kim and Lee(2016)은 Salmonella enterica serovar enteritids를 10분 이내에 검출한 것을 보여준 바 있다. 최근, 꿀벌 바이러스인 black queen cell virus(BQCV)의 검출에 real-time RPA법을 적용한 결과는 3분 26초만에 빠른 target gene 검출을 확인하였고, 시료로부터 추출한 total RNA로부터 8분 36초에 target gene의 검출이 확인됨을 보고하였다(임등, 2016).
한편, RPA법을 사용하여 병원체의 양을 정량하는 것은, RPA법의 빠른 반응속도 등의 이유로 용이하지 아니한 면이 있다. Xia 등(2014)은 IHHNV의 검출에서, fluorophore로 6-carboxyfluorescein FAM을, quencher로 BHQ-1(black hole quencher 1)을, spacer로 THF(tetrahydrofuran spacer)를 사용한 real-time RPA법을 제시하였으나, 정량 실험 결과에서 회귀상수 R2값이 0.943이었음을 보여주었다. 또한, shrimp white spot syndrome virus의 검출에 사용된 real-time RPA는, 정량 실험결과, R2값이 0.90이었음을 보여주어 정확한 정량이 어려움을 나타내었다(Yang et al., 2015).
따라서 본 연구에서는 kSBV 특이 유전자를 빠르게 검출할 수 있는 real-time RPA 방법을 새로이 개발하고자 하였으며, 아울러 모든 특이 유전자의 양을 정량적으로 검출할 수 있는 새로운 정량 실시간 RPA법을 확립하여 보편화 시키고자 하였다.
재료 및 방법
질병시료 및 꿀벌 바이러스의 특이 재조합 DNA들
본 연구에 사용된 질병시료는 kSBV(korean sacbrood virus)에 감염이 확인된 토종벌의 유충이었다. 이 유충들은 경기대학교 양봉장 소재 토봉(Apis ceranae)의 봉군에서 일벌에 의해 소문 밖으로 배출된 유충을 채집한 것이며, kSBV-특이 실시간 PCR법에 의하여 kSBV가 우점하고 있음이 확인된 것이다.
kSBV의 특이 염기서열을 포함하는 pGEM-kSBV-VP1 재조합 DNA는 GenBank 유전자 등록번호 HQ322114의 capsid protein 영역 (823-1,351 bp)인 528 bp를 탑재하고 있다. 또한 pGEM-BQCV-VP3는 GenBank 유전자 등록번호 KR074231의 BQCV capsid protein VP3유전자를 탑재하고 있다(Giang et al., 2015). 이들을 본 연구에서 주형으로 사용하였다.
Total RNA isolation과 cDNA 합성(Reverse transcription)
토종벌의 유충 시료 1마리를 1.5ml tube에 넣고 homogenizer로 분쇄한 후, R&A-Blue™ (iNtRON, Korea) 1ml, chloroform 200μl을 넣고 vortexing으로 혼합하였으며, 이를 13,000 rpm, 4°C 10분간 원심분리 하였다. 원심분리 후, 상등액에 존재하는 total RNA를 spectrophotometer로 정량하였고, -70°C에 보관하며 사용하였다.
cDNA 합성은 전체 RNA 1μg을 주형으로, oligo dT를 사용하였다. 2.5mM dNTP, 100mM DTT, 1x RT-buffer, MML-V reverse transcriptase (Bioneer Inc, Korea)를 사용하였으며, 37°C에 30분 정치시켜 cDNA를 합성하였다. 이후 94°C, 5분으로효소활성을정지시켰다.
RPA 및 real-time PCR용 Primer 설계 및 제작
kSBV(Genbank Accession Number. HQ322114)의 genome structure 내부 open reading frame(ORF) 중 capsid protein을 암호화하는 위치로 특이 primer 쌍을 제작하였다(Fig. 1, Table 1).

Location of primers in Korean sacbrood virus genome (Genbank accession Number HQ322114). PCR product using kSBV-RPA primer set was located at 914-1034 bp in GenBank Accession No. HQ322114.
BQCV의 경우, Genbank Accession Number. KR-074231의 정보를 근거로 구조 단백질 VP3 유전자를 증폭할 수 있는 BQCV-VP3-RPA-F1/R1을 제작하였다(Table 1).
Real-time recombinase polymerase amplification (RT-RPA)
RPA는 Twist Amp® Basic kit (TwistDx, UK)를 사용하여 수행하였다. RPA는 먼저 reaction mix를 제조하였으며, freeze-dried reaction과 280mM Magnesium acetate를 제외한 나머지 즉, 각 2.4 pmole primer (kSBV-RPA-F/R), 1x rehydration buffer, 1x SYBR Green (Bioneer, Korea)을 혼합한 후, freeze-dried reaction의 tube로 옮겨, pipetting 혼합 제조하였다. 이 reaction mix는 RPA반응의 시작 직전에 real-time PCR용 tube로 옮겼으며, 필요량의 Magnesium acetate (MgOAc)를 가하여 반응을 시작하였다.
RPA 전체 반응액의 양은 10μl로 축소하였으며, 그 외의 것은 제작사의 추천방법과 동일하게 진행하였다. RPA 반응을 실시간으로 확인하기 위해 1x SYBR Green 을 첨가하였으며, 반응의 전 과정은 Exicycler quantitative thermal blockTM (Bioneer, Korea)에서 수행하여, 형광값으로 나타나는 DNA 산물의 증감을 실시간으로 측정하였다.
반응시간을 정확히 측정하기 위하여, Exicycler의온도/시간 조건을 37°C의 등온에서 20초를 1 cycle로 설정하였다(반응시간 20분=60 cycles).
Specific-kSBV real-time RPA의 신속 검출법 개발
kSBV의 감염의심 되는 유충시료로부터, total RNA를 추출하였고, 그 중 1μg을 사용하여 cDNA로 제작하였다. 제작된 cDNA를 주형으로 specific-kSBV real-time RPA 방법을 사용하여 target gene을 검출하였다. Real-time RPA 반응은 ExicyclerTM quantitatve thermal block (Bioneer, Korea)를 사용하여 형광 값을 측정하였고, 1 cycle을 20 초로 하여 시간 변화에 따라 형성되는 그래프를 확인하여 주형 DNA의 검출을 실시간 측정하였다.
Specific-kSBV real-time RPA의 민감도 측정
주형DNA의 연속희석을 통하여 7.55×106에서 7.55×102 분자를 각기 RPA에 사용하였다. Quantitative real-time RPA는 ExicyclerTM quantitative thermal block(Bioneer, Korea)를 사용하여 형광 값을 시간에 따라 측정하였다. 주형 DNA의 양에 따른 정량 곡선은 threshold time line을 통하여 회귀 상수 값으로 변환하여 정량을 측정하였다.
다른 주형을 사용한 정량성의 검사에서, 주형 DNA인 pGEM-BQCV-vp3를 2.32×108에서 2.32×104분자까지 연속 희석하여 동일한 방법으로 real-time RPA를 진행하여 정량을 측정하였다.
Primer 농도 조정에 따른 RT-RPA 형광그래프의 변화
Real-time RPA 반응에서 specific primer 농도를 조절하여 RPA의 변화를 형광그래프로 측정하였다. Primer 농도는 최종농도 0.96, 0.48, 0.36, 0.24, 0.12μM로 각기 다르게 하였으며, 주형인 pGEM-kSBV-VP1은 7.55×106에서 7.55×103 분자를 정량, 희석하여 각기 RPA에 사용하였다. Real-time RPA 반응은 ExicyclerTM quantitative thermal block (Bioneer, Korea)를 사용하여 형광 값을 측정하였다.
Magnesium acetate 농도 조정에 따른 RT-RPA 형광그래프의 변화
Real-time RPA 반응에서 magnesium acetate의 농도를 조절하여 RT-RPA의 변화를 형광그래프로 측정하였다. Magnesium acetate의 농도는 14mM, 10mM, 7mM, 3.5mM로 점차 줄여 RT-RPA 반응을 진행하였으며, 주형으로 pGEM-kSBV-VP1를 7.55×106에서 7.55×103 DNA 분자, pGEM-BQCV-VP3를 2.32×108에서 2.32×104 분자까지 희석하여 사용하였다.
kSBV 질병시료를 사용한 qRPA와 qPCR의 정량성 비교
건강한 토봉 애벌레, 감염 의심 애벌레를 각각 육안으로 판별하여, 시료를 S1, S2, S3로 구분하였다. 각 시료로부터 total RNA를 추출하여 약 150μg을 각기 확보하였으며, 그중1μg을 사용하여 cDNA 20μl로제작하였다. cDNA 1μl를 각기 주형으로 사용하여 Ksbv-specific, 정량 real-time RPA 방법과 정량 real-time PCR방법을 각기 사용하여 kSBV 특이 DNA의 정량성을 비교하였다.
결과 및 고찰
kSBV 신속 검출을 위한 kSBV 특이 실시간 재조합 효소-중합효소 증폭법(specific-kSBV real-time RPA)의 개발
kSBV의 감염이 확인된 실제 시료로부터, total RNA를 추출하였고, 그 중 1μg을 사용하여 cDNA로 제작하였다. 제작된 cDNA를 주형으로 kSBV-specific real-time RPA 방법을 사용하여 target gene을 검출하였다.
반응 시간은 총 20분이 소요되었고, cDNA로부터 검출에 소요된 시간은 2분 46초(Threshold time값 8.62)에서 kSBV 유전자의 증폭 및 검출이 확인되었다. Positive control로 사용된 kSBV-specific DNA에서는 53초(Tt값 4.66)만에 kSBV 특이 DNA 증폭이 측정되었다(Fig. 2).
Detection of kSBV-specific DNA by using kSBV-specific real-time RPA with cDNA generated from kSBV infected larvae samples. kSBV-specific real-time RPA was performed in 20 minutes. Amplification of kSBV-specific DNA could be recognized on 2 minutes 46 seconds after beginning. 7.55 ×105 molecules of pGEM-kSBV-VP1 were used for positive control and distilled water was used for negative control.
kSBV 특이 실시간 재조합효소-중합효소 증폭법의 민감도 측정
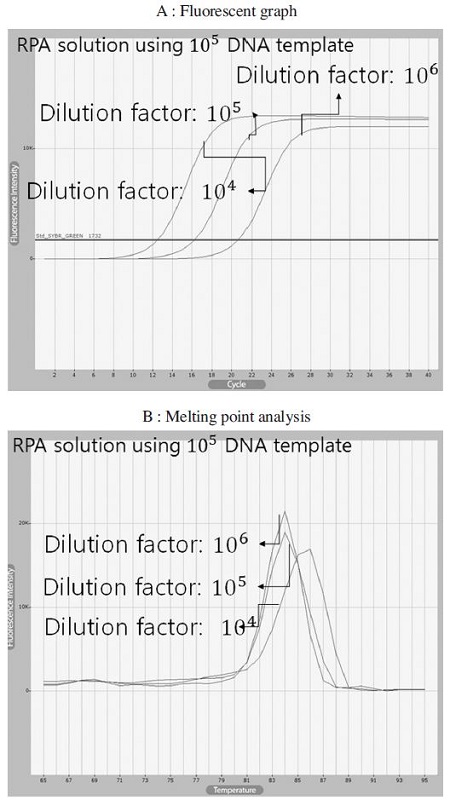
주형 DNA의 연속희석을 통하여 kSBV-specific real-time RPA의 민감도를 측정하였다. Real-time RPA의 초기주형이 7.55×103 분자 이상일 때 형광값의 증가에 의하여 증폭되는 것으로 측정되었으나, 증류수를 사용한 negative control에서도 증폭이 확인되고, 오히려 7.55×103 분자의 결과보다 더 빠른 증가양상을 보여주었다(Fig. 3A).
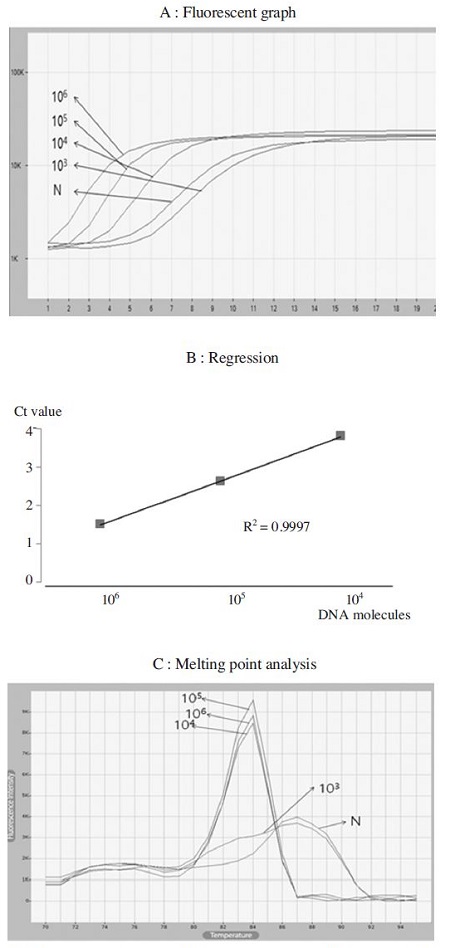
Detection limit of kSBV-specific real-time RPA. 7.55×106 to 7.55×103 molecules of pGEM-kSBV-VP1 were amplified by Using kSBV-specific real-time RPA. Regression coefficiency (R2) was calculated as 0.9997 and Tm value of target gene was estimated 84.5°C from all RPA products except for negative control.
그러나, 융점분석(Melting temperature analysis)에서 증폭된 DNA 산물의 분석결과, 초기주형 7.55×104 분자 이상을 사용한 증폭산물들은 동일한 84.5°C의 Tm(Temperature of midpoint)를 나타낸 것에 반하여, 초기주형 7.55×103 분자를 사용한 증폭산물은 Tm값이 2.0°C 이상의 차이를 보이며, 또한 그래프의 형태도 전형적인 비특이적 증폭산물임을보여주었다(Fig. 3C).
따라서, kSBV real-time RPA는 초기주형 7.55×106~7.55×104 분자범위에서 정량이 가능함을 보여주었고, 이 범위에서 회귀상수(Regression coefficient, R2)값은 0.9997로 높은 정량성을 보여주었다(Fig. 3B).
다른 주형을 사용한 정량성의 검사에서, 주형 DNA인 pGEM-BQCV-VP3 DNA를 2.32×108 분자에서 2.32×104 분자까지 연속 희석하여 real-time RPA를 진행한 결과, 정량이 가능함을 확인 할 수 있었다. 증폭산물을 융점분석으로 측정한 Tm값은 모두 87.5°C로 일치하였으며, 2.32×108~2.32×104 분자범위에서 회귀상수 R2값은 0.9861로 역시 높은 정량성을 보여주었다(Fig. 4).
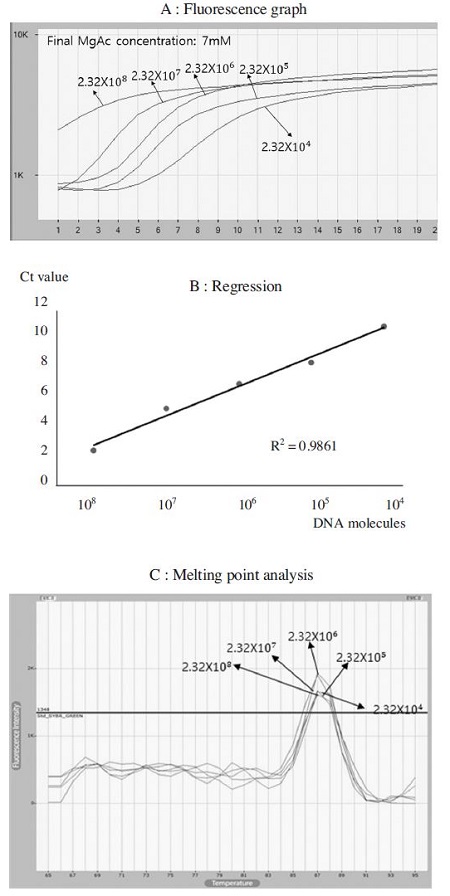
Detection limit of BQCV-specific real-time RPA. 2.32×108 to 2.32×104 molecules of pGEM-BQCV-VP3 were amplified by Using BQCV-specific real-time RPA. Regression coefficiency (R2) was calculated as 0.9861 and Tm value of target gene was estimated 87.5°C from all RPA products.
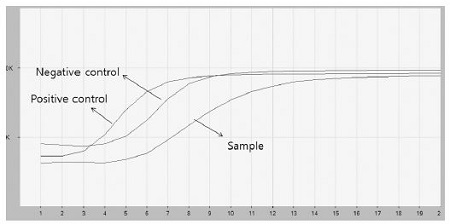
한편, BQCV-RPA 반응 후 용액에 있는 RPA 증폭 산물이 특이 DNA인지를 확인하기 위하여, pGEM-BQCV-VP3를 사용한 실시간PCR을 시행하였다.
먼저, 초기기질의 Ct(Threshold cycles)값 대비 정량 측정을 위하여, 2.32×107~2.32×100의 pGEM-BQCV-VP3 분자를 주형으로 실시간 PCR을 수행하였으며, 얻어진 Ct값들을 이미 알고 있는 초기 기질양에 대비하여, 회귀식 y=-0.3019+11.2239를 산출하였으며, 이의 회귀상수 값은 0.997로 계산되었다.
또한, BQCV-specific real-time RPA solution(최종 농도 7mM magnesium acetate 사용)을 1/104에서 1/106으로 희석하여, 이들을 각기 주형으로 real-time PCR을 진행하였으며, 각 RPA 희석액을 주형으로 한 RT-PCR의 Ct값들은 앞에 제시한 회귀식에 대입하여 계산한 결과, 각 RPA 최종 반응 산물 내에는 1.36×1012, 1.94×1012, 3.52×1012, 1.99×1012, 1.92×1012 분자의 BQCVspecific DNA가 존재하는 것으로 측정되었다. 이는 BQCV-specific real-time RPA가 주어진 특이 DNA를 생산하였음을 보여주고, RPA에서 초기 주형량과는 무관하게 1012수준의 specific DNA로 증폭시킬 수 있음을 보여주었다. 또한 RPA의 최종 형광값이 특이 DNA의 양을 어느 수준 반영할 수 있음도 보여주었다(Fig. 5).
Primer 농도 감소 에 따른 RPA법에서 정량성의 변화
RPA의 빠른 반응 속도는 오히려 초기주형의 양을 정확히 정량하는데 어려움을 주게 된다. 따라서, 반응속도를 적절히 제어하고자 RPA에서 사용하는 primer 농도를 감소시켜 real-time RPA 반응을 진행하였다.
Primer 쌍의 농도는 최종농도 0.96μM, 0.48μM, 0.36μM, 0.24μM, 0.12μM으로 각각 다르게 사용하였으며, 이로써 반응 속도가 점차 느려짐을 발견하였고, 이 조건에서 초기주형량을 각기 104, 105, 106, 107분자로 조정하여 Real-time RPA를 수행하고, 각 형광 값의 변화들을 측정하였다. 얻어진 각 형광그래프에서 초기 주형 량 대비 정량성을 알아보기 위하여 각기 회귀상수R2값을 측정하여 비교 분석하였다.
결과로써, primer의 최종농도 0.48-0.12μM의 범위에서는 회귀상수 R2 값이 0.96 이상임을 보여주었으며, 그 중 최종농도 0.36μM의 primer는 RPA에서 회귀상수 R2 값이 1.000 으로 나타나 가장 높은 정량성을 보였다. 따라서 정량 real-time RPA법에서는 반응 속도 면에서 불리하더라도, 높은 정량성을 추구하기 위해서는 primer의 농도를 과량이 아닌 0.36μM로 조정하는 것이 최적의 조건으로 판단된다(Fig. 6).
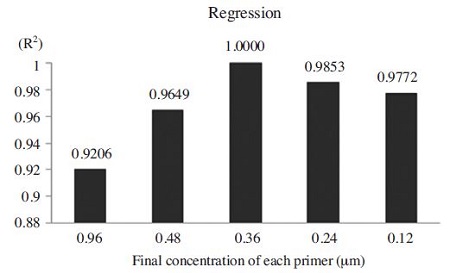
Regression coefficients of kSBV-specific RT-RPA according to different primer concentrations. Using different primer concentrations, RT-RPAs were performed with different quantities of initial templates. Based on Ct values from each set, the regression coefficients were calculated, respectively. The highest value of regression coefficient was showed from RPA using 0.36μM primer concentration.
Magnesium acetate 농도 변화에 따른 RPA법에서 정량성의 변화
Magnesium acetate(MgOAc)는 real-time RPA 반응을 시작하게 하는 필수적 요인으로, 반응속도가 매우 빠른 RPA법을 보다 우수한 정량 방법으로 응용하기 위하여 사용되는 MgOAc의 농도를 낮추어 RPA 반응 속도를 제어하고자 하였다. 각 MgOAC 농도에 따라, 초기주형량을 각기 103, 104, 105, 106분자를 사용하여 Real-time RPA를 수행하였으며, RPA 정량의 결과는 회귀상수R2값으로 측정하여 비교하였다.
MgOAc의 양을 줄여서 RPA정량을 수행하는 것은 10.0mM-7.0mM의 범위에서 유효하며, 7.0mM의 농도에서 회귀상수 R2값은 0.9984로 가장 높은 정량성을 보여주었다(Fig. 7).
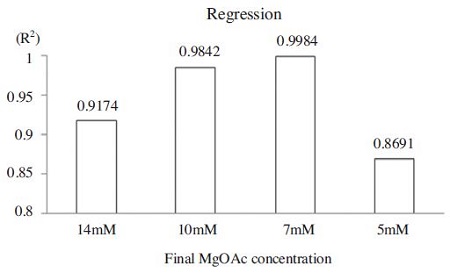
Regression coefficients of kSBV-specific RT-RPA according to different magnesium acetate concentrations. Using different concentrations of magnesium acetate, RT-RPAs were performed with different quantities of initial templates. Based on Ct values from each set, the regression coefficients were calculated, respectively. The highest value of regression coefficient was 0.9984 from RPA using 7mM magnesium acetate.
정량 RPA법의 정립을 위한 Threshold time line(Tt line)의 설정
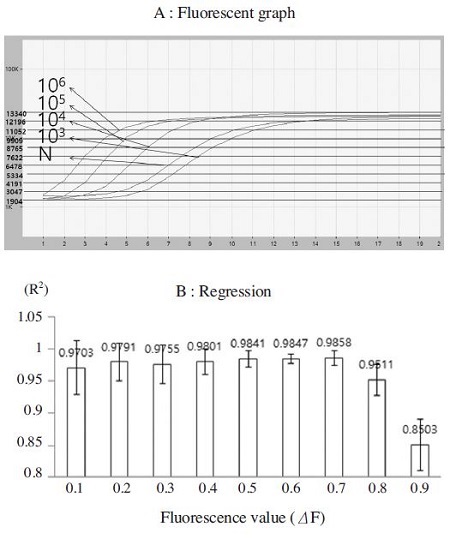
Real-time RPA의 정량을 보다 정확하게 하기 위하여 threshold line의 위치를 정립하고자 하였다. Threshold time line은 real-time RPA에서 형광값의 증가를 나타내는 각 sigmoid형 그래프의 threshold time을 구하고자 하는 것으로, 특정 형광 값을 기준으로 가로선을 그은 것을 말한다. 이 특정 형광값을 보편화하기 위하여, 먼저, RPA 반응에서 처음 측정되는 initial template (I)의 형광 값을 측정하고, 더 이상 형광값이 증가하지 않는 end product (E)의 형광값을 측정한 후, 측정된 양자의 형광값 차이(ΔF)을 구하였다. I와 E의 중간값인 0.5x ΔF값은 절대값으로 I +0.5x ΔF의 형광값이 될 것이나, 편의상 0.1x ΔF, 0.2x ΔF, 0.3x ΔF, 0.4x ΔF, 0.5x ΔF 등의 형광값을 기준으로 threshold time line을 설정하고, 초기주형량 103, 104, 105, 106 분자를 사용한 RPA형광그래프에 적용하여 어느 threshold time line에서 회귀상수R2 값이 가장 우수한지를 비교하였다.
결과로써, 0.1x ΔF부터 0.7x ΔF의 형광값을 사용한 회귀상수 R2은 0.9700 이상의 값을 보였으며, 중간 ΔF값인 0.5x ΔF를 선택하는 것이 무난할 것으로 판단되었다(Fig. 8).
Real-time RPA의 표준 정량법
Real-time RPA에서 주형 DNA를 정확히 정량하기 위하여 RPA의 반응 속도를 적당한 수준으로 제어하는 것은 필요하다. 이에 본 연구는, 정확한 정량을 위하여 real-time RPA에 중요 요소인 primer의 농도 또는 MgOAC의 농도를 감소시켜 RPA 반응 속도를 제어하였다. 즉, 정확한 정량을 위한 real-time RPA를 위하여, 최종농도 0.12~0.36μM primer사용이 유리하며, 과량인 최종농도 14mM MgOAC 보다 최종농도 7.0mM MgOAC 하에서 반응을 시작하여 정량치를 측정하는 것이 보다 유리하다는 것이다.
또한, 형광그래프로부터 threshold time line을 사용하여 정량치를 측정하되, 그 기준이 되는 형광값의 위치는, 최종형광값(end product; E)과 초기형광값(initial template; I)의 차이인 형광값의 변화량(ΔF)의 1/2의 형광값, 즉 1/2x ΔF이며, 이 기준이 가장 실시간 RPA 정량에 유리한 것으로 나타났다.
꿀벌 애벌레 감염 증상에 따른 kSBV 검출확인
토봉(Apis ceranae)의 봉군에서, 외형상 건강한 유충, kSBV 감염 의심 유충, kSBV 감염 확실 유충을 각각 채집하여 이를 각각 S1, S2, S3로 구별하였다.
각 시료로부터 total RNA를 추출하여 각기 약 150μg을 확보하였고, 그 중 1μg을 사용하여 cDNA 20μl로 제작하였다. 제작된 cDNA 1μl를 각기 주형으로 사용하여, kSBV-specific real-time PCR법과 kSBV-specific real-time RPA법을 각각 적용하였고, 그 정량성을 비교하였다.
Real-time PCR방법으로 kSBV-specific DNA 수를 측정한 결과, S1은 2.54×105, S2는 2.56×105, S3 는 7.71×107 분자수가 측정되었다(Fig. 9).
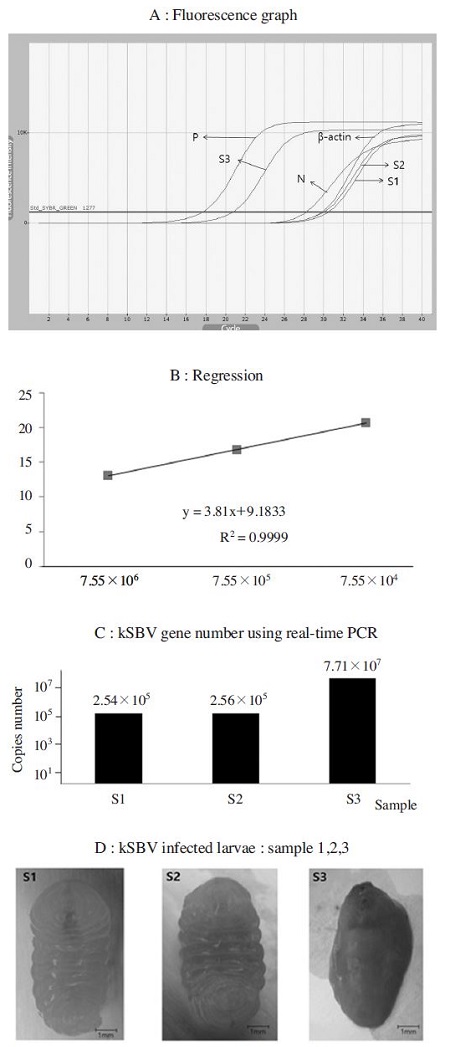
Quantitative detection of kSBV target DNA by using real-time PCR. Using quantitative real-time PCR, kSBV was detected from all larvae samples (S1-S3). The Regression coefficiency of standard curve graph was measured as 0.9999. The target DNA copies of kSBV were estimated as 2.54×105, 2.56×105, 7.71×107 respectively.
이는 우선 육안 판정에 의한 kSBV 감염시료가 건강해 보이는 유충시료보다 약 300배 이상 많은 kSBV를 가지고 있다는 것이며, 또한 동일 봉군내 건강 유충도 kSBV를 다량 보유하고 있다는 사실을 알 수 있다.
한편, Real-time RPA법으로 kSBV-specific DNA수를 측정한 결과, 토봉 유충에 따라서 S1 은 1.86×106, S2는 1.88×106, S3는 9.06×108 수가 측정되었다(Fig. 10).
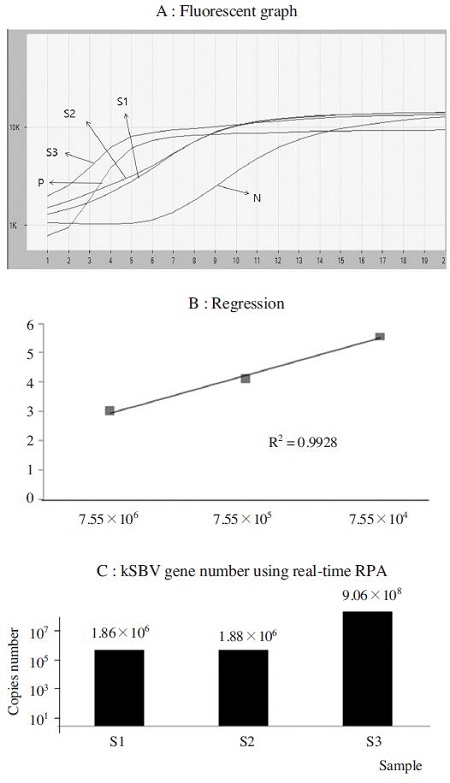
kSBV-specific Quantitative real-time RPA with infected larvae of Apis cerana. From the infected larvae samples which were used for RT-PCR, kSBV target DNA was also estimated by kSBV specific real-time RPA. The Regression coefficiency of standard curve graph was measured as 0.9928 and the target DNA copies of kSBV were estimated as 1.86×106, 1.88×106, 9.06×108 respectively.
실시간 PCR 및 실시간 RPA에서 정량의 기준으로 사용된 초기 주형량은 각기 7.55×104, 7.55×105, 7.55×106으로 동일한 분자수를 사용하였으며, 전자의 회귀상수 R2의 값은 0.9999, 후자의 회귀상수 R2의 값은 0.9928으로 거의 대등함을 보여주었다.
그러나, 양자간 산출된 kSBV 특이 분자들의 수는 건강해 보이는 유충시료 S1에서, RPA결과는 PCR결과에 비하여 7.3배 높은 측정치를 보여주었으며, kSBV 감염 확실 유충시료 S3에서도, RPA결과는 PCR결과에 비하여 11.8배의 높은 측정치를 보여주었다. 이런 양자간의 차이는 형광값 측정의 차이에서 기인하는 것으로 해석하며, 보다 비특이적 증폭이 많이 발생하는 RPA에서 보다 높은 형광값이 측정된 것이 아닌가 추측한다(Fig. 9, Fig. 10).
같은 시료로부터 Real-time PCR법과 real-time RPA법을 각기 사용하여, 동일 target DNA의 정량결과는 real-time RPA을 사용하는 것이 보다 빠른 시간에 정량결과를 볼 수 있으나, 민감도와 정량적 검출에서 약간의 손실이 있음을 보여주며, 절대 정량치에서 RPA에 의한 분자수 측정은 비특이적인 산물의 증산으로 인하여 형광값의 비정량적 증가가 있지 않았을까 추측한다.
Acknowledgments
본 연구는, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(과제번호 115058-02, 과제번호 115102-03), 농생명산업기술개발사업(312027-03) 및 수출전략기술개발사업(과제번호 115067-02), 그리고 2016학년도 경기대학교 대학원연구원장학생장학금지원에의하여수행되었음.
References
- 유미선, 김동수, 김일욱, 권순환, 윤병수, (2009), Israel Acute Paralysis Virus의 진단을 위한 Real-Time PCR 진단법의 개발, J. Apiculture, 24(1), p31-36.
- 유미선, 최용수, 박용하, 윤병수, (2010), Chrnic Bee Paralysis Virus의 진단을 위한 Real-Time PCR 진단법의 개발, J. Apiculture, 25(1), p31-37.
- 이보람, 유미선, Nguyen Van Phu, 노지나, 윤병수, (2011), Loop-mediated Isothermal Amplification (LAMP)법을 이용한 백묵병 원인균 Ascosphera apis의 검출법개발, J. Apiculture, 26(2), p103-111.
- 임수진, Giang Thi Huong Luong, 민상현, 왕지희, 윤병수, (2016), 역전사 실시간 Recombinase Polymerase Amplification(RT/RT RPA)에 의한 꿀벌 Black Queen Cell Virus의 신속 검출, J. Apiculture, 31(1), p41-50.
-
Giang Thi, Huong Luong, J-S. Lee, S-J. Yong, B-S. Yoon, (2015), Development of Ultra-Rapid Reverse Transcription Real-Time PCR for Detection against Black Queen Cell Virus in Honeybee, J. Apiculture, 30(3), p171-179.
[https://doi.org/10.17519/apiculture.2015.09.30.3.171]

- Han, S.H., Y.S. Choi, and M.L. Lee, (2011), Development of Highly Specific Quantative Real-Time PCR Method for the Detection of Sacbrood Virus in Korean Honeybees, Apis cerana, J. Apiculture, 26(3), p233-240.
-
Kim, J.Y., J.L. Lee, (2016), Rapid detection of salmonella enterica serovar enteritidis from eggs and chicken meat by real-time recombinase polymerse amplification in comparison with the two-step real-time PCR, Journal of Food Safety.
[https://doi.org/10.1111/jfs.12261]

-
Piepenburg, O., Colin H. Williams, Derek L. Stemple, Niall A. Armes, (2006), DNA Detection Using Recombination Proteins, PLoS Biol, 4(7), p1115-1121.
[https://doi.org/10.1371/journal.pbio.0040204]

-
Xia, X., Y. Yu, M. Weidmann, Y. Pan, S. Yan, & Y. Wang, (2014), Rapid detection of shrimp white spot syndrome virus by real time, isothermal recombinase polymerase amplification assay, PloS one, 9(8), pe104667.
[https://doi.org/10.1371/journal.pone.0104667]

-
Yang, Y., X. Qin, G. Wang, Y. Zhang, Y. Shang, & Z. Zhang, (2015), Development of a fluorescent probe-based recombinase polymerase amplification assay for rapid detection of Orf virus, Virology journal, 12(1), p206.
[https://doi.org/10.1186/s12985-015-0440-z]