
봉변에서 특이 유전자 검출법에 의한 봉군 내 꿀벌가시응애류 (Tropilaelaps)의 정량적 검출
Abstract
Rapid detection of Tropilaelaps, an external parasite of honeybees that lead to malformation of honeybee or colony collapse disorder, is becoming important. But it is very difficult to find with the naked eye of Tropilaelaps. In this study, we have developed a method to detect the specific gene of Tropilaelaps from the hive debris and to know the number of Tropilaelaps in the hive through Tropilaelaps-specific quantitative detection. Tropilaelaps-specific gene amplified in DNA extracted from hive debris by consecutive PCR (1st detection, 2nd nested PCR). It could detect 101 molecules level of Tropilaelaps-specific gene and confirm the amplification of the Tropilaelaps-specific gene. It was possible to accurately quantify the number of Tropilaelaps from the hive debris sample, which is difficult to discriminate the presence of Tropilaelaps visually, through Tropilaelaps-specific detection. Under the microscope, Tropilaelaps was collected and quantitative detection of Tropilaelaps-specific genes was performed. It was possible to quantify the number of Tropilaelaps present in the hive through the molecules of the quantified Tropilaelaps-specific genes. We suggest that hive debris can represent as a micro-environment to hive and show that it can be a simpler and more accurate sample than using a parasitic host honeybee. We expect that hive debris should facilitate the monitoring of Tropilaelaps in hive.
Keywords:
Detection, Honeybee, Parasite, Quantification, Tropilaelaps, Ultra-rapid PCR서 론
Tropilaelaps는 꿀벌의 외부 기생충으로서 서양종 꿀벌에서 1961년 처음으로 발견되었다 (Delfinado and Baker, 1961). 이들의 주요 기생 숙주는 아시아 꿀벌인 Apis dorsata로 알려져 있다 (Laigo and Morse, 1968). 하지만 최근 Apis mellifera, Apis cerana 등의 여러 종의 꿀벌에서도 발견됨이 보고되었다 (Bailey and Ball, 1991; Schmid-Hempel, 1998). Tropilaelaps의 성체는 소비 내에서 겉으로 보기에 매우 작고 (길이 1 mm 미만), 길며 밝은 갈색을 띠며, 빠른 움직임을 가지고 있는 것이 일반적인 특징이다 (Delfinado and Baker, 1961; Delfinado-Baker and Baker, 1982). Tropilaelaps는 Varroa destructor와 유사하게 기생하며 haemolymoph를 빨아들여 꿀벌의 기형 또는 죽음에 이르게 하며, colony의 감소 및 폐사를 이끌 수 있다 (Luo et al., 2011).
지금까지 Tropilaelaps는 4가지 종이 있는 것으로 보고되어 있으며, Tropilaelaps clareae, Tropilaelaps mercedesae, Tropilaelaps koenigerum, Tropilaelaps thaii가 존재한다. T. clareae는 A. dorsata 및 A. mellifera에 기생하며, T. koenigerum과 T. mercedesae는 Apis dorsata dorsata에도 기생한다고 보고된 바 있으며, T. mercedesae는 A. mellifera에 기생하는 것으로 보고되어 왔다. T. thaii는 히말라야 지역에서 발견되는 A. laboriosa에 기생하는 것으로 보고되었다 (Anderson and Morgan, 2007). 서로 다른 4개의 종들 중에 T. mercedesae와 T. clareae는 A. mellifera에 가장 치명적인 것으로 보고되어 왔다 (de Guzman et al., 2017).
A. mellifera에도 Tropilaelaps의 존재가 확인된 이래로 숙주의 범위를 넓혀가며 이란, 아프가니스탄, 케냐, 한국 및 뉴기니 서부 태평양 섬에 이르기까지 확산되었다 (Burgett et al., 1983; Woyke, 1984; Delfinado-Baker and Aggarwal, 1987; Kumar et al., 1993; Anderson, 1994; Matheson, 1996; Sammataro et al., 2000; Otis and Kralj, 2001). 이렇듯 Tropilaelaps는 여러 지역에서 A. mellifera의 해충으로 발전하였으며, 전 세계적으로 새로운 위협으로 관심받고 있다 (OIE 2004). 국내에서는 벌통 내 서식하는 응애류에 대한 연구로서 T. clareae가 최초로 보고된 바 있으나 (Lee and Woo, 1995), 국내에서는 Tropilaelaps의 특이 유전자를 사용한 유전자 검사법은 보고된 바 없었다.
Tropilaelaps에 대한 기존의 연구는 주로 형태학적 연구들이 보고되어 왔으며 (Anderson and Morgan, 2007), 형태적 차이의 정확한 구분은 매우 힘들며, 특이 유전자를 사용한 분류학적 기술들이 필요하였다. 최근에는 핵의 ribosomal DNA (rDNA) 또는 미토콘드리아 DNA (mtDNA)를 사용한 계통 발생 연구들이 널리 사용되어 왔으며, rDNA의 ITS1-5.8S-ITS2의 영역 및 mtDNA의 cytochrome oxidase subunit I (COI) 유전자를 사용한 연구들이 많이 보고되어 왔다 (Anderson and Morgan, 2007). 또한 이러한 유전자들이 Tropilaelaps의 4종을 구별하는 데 있어 좋은 지표가 될 수 있었으며, 이들의 유전적 변이뿐만 아니라 그에 따른 지리적 분포까지 보고되었다 (Luo et al., 2011). 2013년에는 여러 가지 빠른 조사 기술을 통해 Tropilaelaps를 검출할 수 있는 방법들을 제시하였으며 (Pettis et al., 2013), Tropilaelaps는 또한 Varroa destructor와 마찬가지로 꿀벌의 바이러스를 전달하는 매개체 역할을 하는 것으로 보고되어 있으며, 그에 따른 검출의 중요성은 매우 크게 제시되어 왔다 (Dainat et al., 2009). 이처럼 Tropilaelaps를 검출하는 방법 및 분류할 수 있는 방법들은 다수 제시되어 왔으며, Tropilaelaps의 검출의 중요성 또한 크게 제시되어 왔다.
따라서, 본 연구는 Tropilaelaps의 특이 유전자를 빠르고 간단하게 검출하는 유전자 검사법이다. 매우 작은 크기의 Tropilaelaps를 봉변 또는 봉군에서 개체로서 수집하여 확인하는 것은 매우 숙련된 기술이 요구되는 어려운 방법이다. 벌통의 환경을 대변할 수 있는 일종의 미소환경인 벌통의 봉변을 사용하며, 봉변 내 존재하는 Tropilaelaps의 특이 유전자에 대한 정량분석을 통해 벌통 내 Tropilaelaps의 수를 추론할 수 있음을 보여주는 연구이다. 우리는 Tropilaelaps의 감염여부를 확인하기 위해 봉변이 벌통을 대변할 수 있는 수단으로 사용할 수 있음을 제시하고자 한다. 또한 Tropilaelaps의 중요성이 다수 보고된 만큼 본 방법을 통해 Tropilaelaps의 빠르고 간단한 검출이 가능함을 제시하고자 하였다.
재료 및 방법
Tropilaelaps의 시료 수집 및 DNA 추출
Tropilaelaps 시료의 수집은 경기도 용인 및 수원의 양봉장의 폐사된 벌통의 봉변을 수집하였으며, 봉변 내 Tropilaelaps의 존재를 육안 및 현미경을 통해 확인하였다. 수집된 시료는 50 mL conical tube에 담아 -20℃에 보관하며 본 연구에 사용되었다. 육안 및 현미경을 통해 확인된 Tropilaelaps 시료는 따로 모아서 DNeasy® blood & tissue kit (Qiagen, Germany)를 사용하여 200 μL의 Elution buffer에 DNA를 추출하여, Tropilaelaps 1마리에 포함된 특이 유전자 분자수 정량에 사용되었다.
벌통에서 채집된 봉변 0.5 g을 glass bead (pore size : 1 mm, Germany)가 들어있는 2.0 mL tube에 담고, 1 mL의 D.W를 넣어 MagNa Lyser (Roche, Switzerland)로 분쇄하였다. 분쇄된 시료 중 200 μL는 DNeasy® blood & tissue kit (Qiagen, Germany)를 사용하여 100 μL의 Elution buffer (Qiagen, Germany)에 DNA를 추출하였으며, 이는 -20℃에 보관하며 본 연구의 PCR 주형으로 사용되었다.
Tropilaelaps의 특이 유전자의 primer 설계 및 재조합 DNA 확보
Tropilaelaps의 특이 유전자의 증폭을 위하여 Tropilaelaps 특이 primer를 새로이 설계하였다 (Table. 1). Tropilaelaps를 특이적으로 증폭하기 위한 유전자는 Cytochrome Oxidase subunit I (COI)과 ITS1-5.8S-ITS2 (18S rRNA)를 사용하였으며, 각 유전자에 특이적인 염기서열은 NCBI에 등록된 정보를 통해서 Tropilaelaps 특이 primer를 설계하였다. 설계된 primer들은 보고된 Tropilaelaps 속의 4가지 종을 검출할 수 있으며, Apis mellifera와 다른 기생충 (Varroa destructor, Acarapis woodi, Aethina tumida)들을 검출 하지 않는 염기서열을 기본으로 하여 설계되었다 (Supplement Fig. 1).
본 연구의 표준 DNA로 사용할 Tropilaelaps 특이 유전자의 재조합 DNA를 확보하기 위해 봉변에서 Tropilaelaps의 특이 유전자가 증폭된 PCR 산물을 pGEM-T easy vector (Promega, USA)에 T-A cloning하여 대장균 DH5αFʹ에 형질 전환하여 확보하였다. 확보된 재조합 DNA는 각각 pTro-COI 256, pTro-18S241이라 명명하였으며, 이들은 sequence 분석 (Bionics, Korea)하여 기존 Genbank의 정보와 비교하여 sequence의 homology를 분석하였다. pTro-COI256에 삽입된 Tropilaelaps COI 유전자 (256 bp)는 Tropilaelaps mercedesae (Accession number. KY865195)와 98% (250/254 bp)로 가장 높은 상동성을 확인하였다. 또한, Tropilaelaps thaii (Accession number. EF025452)와 86% (222/257 bp), Tropilaelaps clareae (Accession number. EF025464)와 86% (221/257 bp), Tropilaelaps koenigerum (Accession number. EF025449)와 83% (213/256 bp)로 4종의 상동성을 확인하였다. pTro-18S241에 삽입된 Tropilaelaps 18S 유전자 (241 bp)는 Tropilaelaps mercedesae (Accession number. KP774529)와 99% (241/242 bp)로 가장 높은 상동성을 확인하였다. 또한, Tropilaelaps clareae (Accession number. AF544013)와 99% (240/242 bp), Tropilaelaps thaii (Accession number. EF025477)와 98% (236/241 bp), Tropilaelaps koenigerum (Accession number. EF025475)와 95% (230/241 bp)로 4종의 상동성을 확인하였다. 염기서열의 분석 결과로 Tropilaelaps 특이 유전자의 재조합 DNA는 Tropilaelaps mercedesae로 추정할 수 있었다. 재조합 DNA의 염기서열은 각각 accession number MH719215 (COI), MH725269 (18S)로 Genbank에 새로이 등록되었다. 확보된 재조합 DNA는 -20℃에 보관하며 차후 연구의 표준 DNA로서 사용되었다.
Tropilaelaps 특이 유전자의 검출한계 확인
Tropilaelaps 특이 유전자의 정량에 앞서 본 연구에 사용된 Tropilaelaps 특이 primer 쌍들의 검출한계 및 정량 회귀선을 확인하였다. 표준 DNA로서 사용된 각 Tropilaelaps 특이 유전자의 재조합 DNA는 1.0×108 분자부터 1.0×100 분자까지 연속 희석하여 Tropilaelaps 특이 PCR의 주형으로 사용하였다. 사용된 PCR 기기는 GENECHECKERTM (Genesystem Co. Ltd., Korea)를 사용하였으며, 2×Rapi : Detect Master mix (Genesystem Co. Ltd., Korea)를 사용하여 총 10 μL로 조성하였다. PCR 조건은 Pre-denaturation 95℃, 30초 이후에 Denaturation 95℃, 1초 - Annealing 58℃, 3초 - Polymerization 72℃, 1초를 1 cycle로 하여, 총 50 cycle을 수행하였다. Tropilaelaps 특이 PCR에서 측정된 연속 희석된 표준 DNA의 CT값을 통해 정량 회귀선을 작성하였다. 정량 회귀선은 차후 봉변으로부터 1차 Tropilaelaps 특이 PCR 검출을 통해 측정된 CT값을 대입하여 Tropilaelaps 특이 유전자의 분자수를 정량하는 데 사용하였다.
Tropilaelaps 특이 유전자의 확인을 위한 nested PCR의 적용
1차 Tropilaelaps 특이 PCR에서 특이적인 검출이 확인되지 않은 PCR 산물에 대해서는 2차 nested PCR을 적용하여 Tropilaelaps 특이 유전자의 존재를 확인하고자 하였다. 또한, 1차 Tropilaelaps 특이 PCR에서 검출이 확인된 PCR 산물에도 2차 nested PCR을 적용하여 Tropilaelaps 특이 유전자를 재확인하고자 하였다. Nested PCR은 1차 Tropilaelaps 특이 PCR 산물을 D.W에 100배 희석한 뒤, 주형으로 사용하였다. Nested PCR 조건은 Pre-denaturation 95℃, 30초 이후에 Denaturation 95℃, 1초-Annealing 58℃, 3초-Polymerization 72℃, 1초를 1 cycle로 하여, 총 50 cycle을 수행하였다.
봉변 시료로부터 Tropilaelaps의 특이 유전자의 정량 검출
꿀벌의 벌통에서 수집된 봉변들에서 각각 DNA를 추출하였으며, 이를 주형으로 사용하여 Tropilaelaps 특이 PCR을 수행하였다. 봉변의 Total DNA 내 존재하는 Tropilaelaps 특이 유전자의 분자수 정량을 통해 봉변 내 존재하는 Tropilaelaps의 수를 counting 확인하고자 하였다.
1차적으로 봉변 시료 내 Tropilaelaps의 존재를 해부현미경 하에 눈으로 직접 관찰하였다. 관찰된 Tropilaelaps는 모아서 따로 10마리의 Tropilaelaps에서 DNA를 추출하였으며, Tropilaelaps 특이 정량 PCR을 통해 1마리에 존재하는 특이 유전자의 분자수를 정량하였다. 1마리에 포함된 Tropilaelaps 특이 유전자의 분자수를 통해 봉변 내 존재하는 Tropilaelaps의 수를 counting할 수 있었다.
각 봉변 시료에서 추출된 DNA를 사용한 Tropilaelaps 특이 PCR에서 측정된 CT값을 사용하여 봉변 DNA에 포함된 Tropilaelaps 특이 유전자의 분자수를 계산하였으며, 계산된 분자수와 Tropilaelaps 1마리에 해당하는 특이 유전자 분자수를 비교하여 봉변 1 g에 포함된 Tropilaelaps의 마리수를 정량할 수 있었다.
봉변으로부터 추출된 DNA 용액 1 μL에서 Tropilaelaps 특이 정량 PCR을 통해 측정된 Tropilaelaps 특이 유전자의 분자수는 총 DNA 용액 100 μL에 포함된 총 분자수를 계산하였다. 이는 사용한 봉변 용액 1 mL 중 200 μL에 포함된 분자수이며, 다시 총 봉변 용액 1 mL에 포함된 분자수를 계산하였다. 최종적으로 Tropilaelaps 1마리에 해당하는 분자수와 비교하여 봉변 1 g 당 Tropilaelaps의 개체수 (unit/g)를 계산할 수 있었다.
결과 및 고찰
Tropilaelaps 특이 유전자의 검출한계 확인
본 연구에서 사용된 Tropilaelaps 특이 primer 쌍들의 검출한계를 확인하고자 하였다. Tropilaelaps 특이 COI 및 18S 유전자의 detection primer와 nested primer, 총 4쌍의 검출한계를 연속 희석된 Tropilaelaps 특이 표준 DNA를 통해 확인하였으며, COI 및 18S 유전자의 detection primer로 증폭시킨 Tropilaelaps 특이 표준 DNA의 CT값을 통해 정량 회귀선 및 회귀상수를 확인하였다.
Tropilaelaps 특이 primer 쌍들은 102 분자 수준의 낮은 분자까지 검출 가능하였으며, COI 및 18S 유전자 특이 표준 DNA로부터 측정된 각 CT값을 통해 정량회귀선 및 회귀상수를 얻어내었으며, 회귀상수는 각각 0.9958과 0.9951로 높은 정량성을 확인할 수 있었다 (Fig. 1).
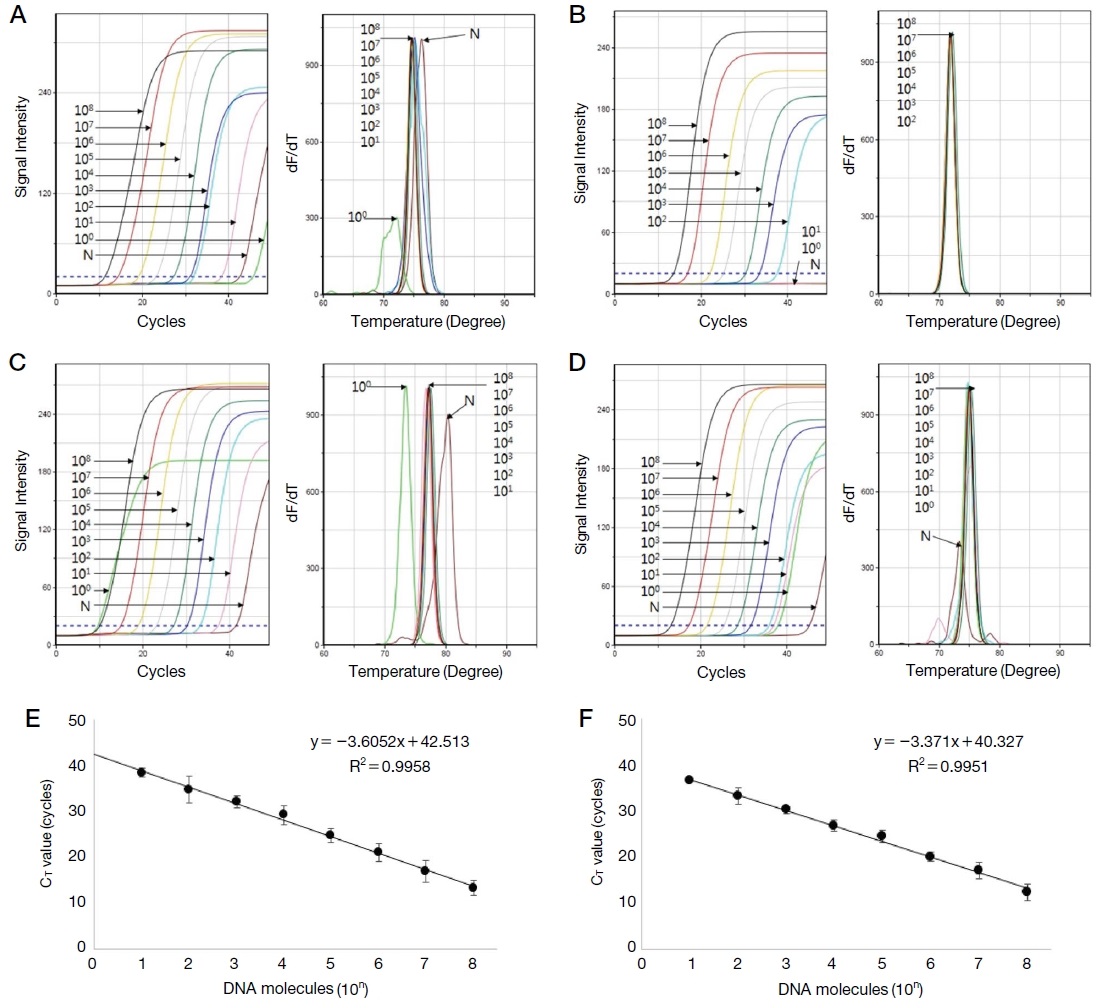
Detection limit and quantitative regression equation of Tropilaelaps-specific gene. This shows the detection limit and quantitative regression equation of Tropilaelaps-specific ultra-rapid PCR. Recombinant DNA of each gene was serially diluted from 108 molecules to 100 molecules and used as a template. A and B represent detection limit of Tropilaelaps COI-specific detection and nested PCR. C and D represent Tropilaelaps 18S-specific PCRs. E and F represent quantitative regression equation of Tropilaelaps-specific PCRs, COI and 18S gene.
Tropilaelaps 특이 유전자의 정량 회귀선은 실제 봉변 시료로부터 추출된 DNA로부터 Tropilaelaps 특이 PCR을 통해 측정된 CT값을 대입하여 Tropilaelaps 특이 유전자의 분자수를 계산하는 데 사용되었다.
봉변 시료에서 Tropilaelaps의 관찰 및 특이 유전자의 정량
봉변 시료에서 Tropilaelaps의 존재를 확인하고자 채집된 봉변 시료로부터 해부 현미경 (Olympus; SZ61, Japan)을 사용하여 존재를 확인하였다 (Fig. 2). Tropilaelaps는 육안상 다른 봉변들과 구분하기 매우 힘들었으며, 현미경 상에서도 그 존재를 찾기 매우 어려웠다. 또한 꿀벌의 기생충인 꿀벌 응애보다도 더 작은 크기로 인해 Tropilaelaps를 찾는 것이 어려웠다. Tropilaelaps의 크기는 꿀벌 응애와 비교하여, 0.51 mm×0.90 mm로 측정되었다. 현미경 하에서 봉변 시료로부터 존재를 확인한 Tropilaelaps 시료는 모아서 1마리에 해당하는 Tropilaelaps 특이 유전자의 분자수 정량을 수행하였다.

Observation of Tropilaelaps under a microscope. Tropilaelaps and Varroa destructor collected from same hive debris were compared in size under a microscope (Olympus; SZ61, Japan). A: Hive debris, B: abdomen, C: dorsum. The left is Tropilaelaps and the right is Varroa destructor. The magnification of the microscope were ×13.4 (picture A) and ×30 (picture B, C). The size of Tropilaelaps was 0.51 mm of width and 0.90 mm of length.
Tropilaelaps 10마리에서 추출한 DNA는 200 μL의 Elution buffer (Qiagen, Germany)에 녹였으며, 이 중 1 μL를 사용하여 Tropilaelaps 특이 PCR을 수행하였다. Tropilaelaps 특이 PCR에서 측정된 각 CT값 (26.09/18.23 cycles; COI/18S gene)을 Tropilaelaps 특이 유전자의 각 정량 회귀선에 대입하여 특이 유전자의 정량을 수행하였다. 1 μL 당 유전자는 3.0×104 (COI), 9.9×106 (18S) 분자수이며, 200 μL에는 총 6.0×106 (COI), 2.0×109 (18S) 분자가 존재하며, 1마리에 존재하는 유전자 수는 6.0×105 (COI), 2.0×108 (18S) 분자수로 계산되었다.
봉변 시료로부터 Tropilaelaps 특이 유전자의 정량 검출
각 봉변으로부터 추출된 DNA는 Tropilaelaps 특이 PCR을 수행하여 특이 유전자의 분자수를 정량할 수 있었다. 각 봉변 시료로부터 측정된 CT값은 표준 DNA에 의해 제작된 각 Tropilaelaps 특이 유전자의 정량 회귀선에 대입하여 Tropilaelaps 특이 유전자의 분자수를 정량하였다. 또한 봉변 시료뿐만 아니라 채집된 꿀벌 시료에도 본 방법을 적용하여 Tropilaelaps 특이 유전자 정량을 수행하였다.
Tropilaelaps 특이 PCR을 통해 용인과 수원에서 채집한 봉변 시료 모두에서 특이 유전자가 검출됨을 확인하였으며, 각 Tropilaelaps 특이 유전자 PCR에서 측정된 CT 값을 정량 회귀선에 대입하여 특이 유전자의 분자수 정량이 가능하였다. 계산된 유전자 분자수는 Tropilaelaps 1마리에 해당하는 유전자 수로 계산하여 봉변 1 g에 존재하는 Tropilaelaps의 수를 counting할 수 있었다 (Fig. 3, Table 2).
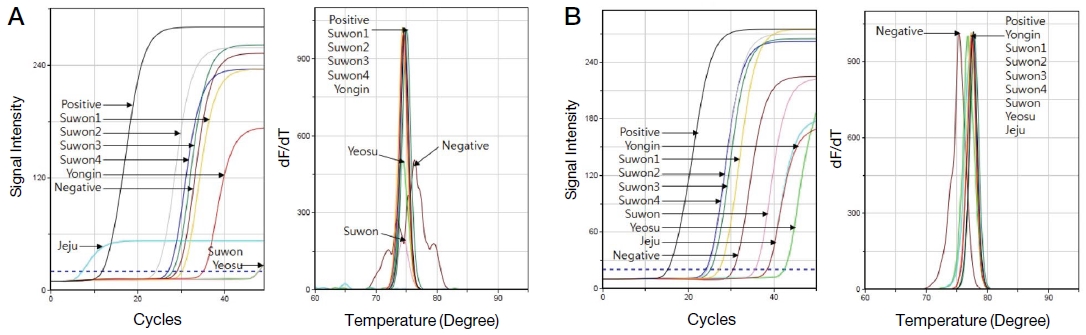
Detection for Tropilaelaps-specific gene from hive debris and adults. This shows the results of the Tropilaelaps-specific ultra-rapid PCR of DNAs extracted from hive debris and honeybee adults. The CT values measured by Tropilaelaps-specific PCRs were assigned to Tropilaelaps-specific quantitative regression equation and the molecules of Tropilaelaps-specific genes could be quantified. A shows the fluorescence graph and the melting analysis of Tropilaelaps COI-specific PCR. B shows the Tropilaelaps 18S-specific PCR. Positive Tm value of COI-specific PCR is 75.41℃ and 18S-specific PCR is 77.68℃.
봉변 시료뿐만 아니라 일부 꿀벌 성체에서도 Tropilaelaps 특이 유전자가 확인되었으나, 매우 적은 양의 특이 유전자가 정량 검출되어 오염 수준으로 판단된다. 봉변 및 꿀벌 성체의 DNA로부터 증폭된 PCR 산물은 2차 nested PCR에 적용하여 그 증폭여부를 재확인하였으며, 성체의 결과와 같이 미량 또는 증폭되지 않은 산물에도 적용하여 Tropilaelaps의 특이 유전자의 여부를 확인하였다.
본 결과에서 보여주는 것은 같은 벌통에서 채집된 봉변과 성체에서 다른 결과를 확인하였으며, 봉변 시료는 꿀벌 성충보다 Tropilaelaps의 존재를 대변해 주기에 더 적합한 시료이며, 벌통의 환경을 분석하기에 더 좋은 지표라 판단된다.
봉변 시료에서의 Tropilaelaps 특이 유전자의 확인을 위한 nested PCR
봉변 시료에서 Tropilaelaps 특이 유전자의 존재를 확인하고자, 1차 PCR 산물에 2차 nested PCR을 적용하여 Tropilaelaps 특이 유전자를 확인하고자 하였다. Nested PCR의 주형은 각 1차 특이 PCR 산물을 D.W에 100배 희석하여 사용하였다.
1차 PCR에서 증폭이 확인된 봉변 시료의 DNA 내 존재하는 Tropilaelaps 특이 COI 및 18S 유전자 모두 2차 nested PCR을 통해 그 증폭여부를 다시 확인할 수 있었으며, 증폭이 되지 않았거나 미량의 Tropilaelaps 특이 유전자가 검출되었던 꿀벌 성체의 DNA에서도 2차 nested PCR을 통해 Tropilaelaps 특이 유전자의 존재를 확인할 수 있었다 (Fig. 4, Table 3).
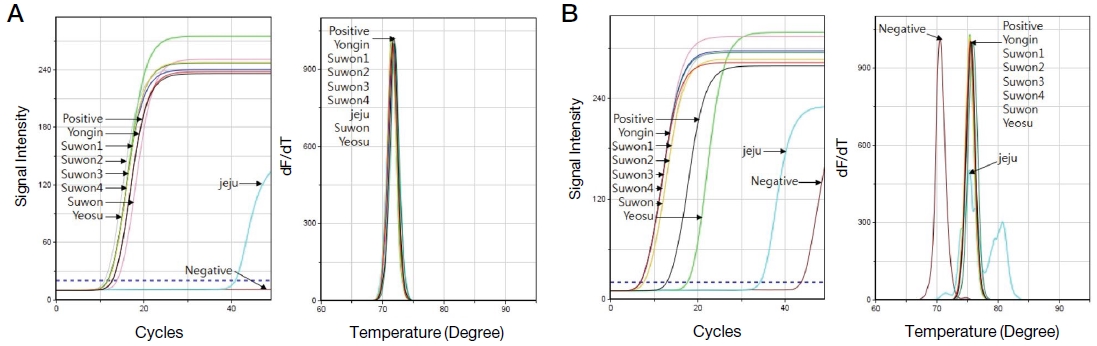
Confirmation of Tropilaelaps-specific gene amplification by nested PCR. This shows the results of the Tropilaelaps-specific nested PCR of primary Tropilaelaps-specific PCR products. Nested PCR could detect to a tiny amount of Tropilaelaps-specific gene and confirm the amplification of the Tropilaelaps-specific gene. A shows the fluorescence graph and the melting analysis of the Tropilaelaps COI-specific nested PCR. B shows the Tropilaelaps 18S-specific nested PCR. Positive Tm value of COI-specific nested PCR is 71.90℃ and 18S-specific nested PCR is 74.98℃.
Nested PCR은 1차 PCR 증폭 산물에 존재하는 Tropilaelaps의 특이 유전자를 확인하는 수단으로 사용되었으며, 1차 PCR에서 증폭이 확인된 산물에서는 재확인의 수단으로 사용되었으며, 증폭이 확인되지 않은 산물에서는 2차 검출을 통해 검출 유무를 확인할 수 있었다.
결 론
본 연구는 꿀벌 및 벌통에 기생하는 Tropilaelaps의 정량적 검출을 위한 특이 유전자 검사법을 개발하였다. 일반적으로 잘 알려져 있는 꿀벌의 기생충인 꿀벌 응애 (Varroa destructor) 보다 크기가 더 작은 Tropilaelaps는 육안 또는 현미경 하에서도 발견 및 판별이 어려웠다. 그로 인해 봉군 내 존재하는 Tropilaelaps의 수는 더욱더 파악하기 힘들었으며, 그러한 어려움은 유전자 검사법을 통해 해결 가능하였음을 보여주었다.
또한, 2번의 연속된 유전자 검사법 (1차 detection 후, 2차 nested PCR)은 유전자 검사법의 정확성을 향상시켜 줄 뿐만 아니라, 극미량의 유전자까지도 검출을 가능하게 하였다. 본 연구에서도 1차 PCR을 통해 봉변 시료에서 Tropilaelaps의 존재를 정량 검출할 수 있었으며, 2차 nested PCR을 통해 그 존재 여부를 재확인하였으며, 1차 PCR에서 검출하지 못했던 시료에서는 2차 nested PCR을 통해 검출 가능하였다.
Tropilaelaps의 정량적 검출을 위해 현미경 하에서 Tropilaelaps의 존재를 확인하고 이를 수집하였으며, 수집된 Tropilaelaps에서 DNA를 추출하여 1마리에 해당하는 특이 유전자의 정량을 수행하였다. 그 결과 측정된 Tropilaelaps 1마리의 특이 유전자 수를 통해 봉변 1 g에 존재하는 Tropilaelaps의 수를 추정할 수 있었다.
최근 봉변은 벌통의 환경을 대변할 수 있는 미소환경으로서 병원체의 감염확인의 수단으로 꿀벌 시료보다 더 분명하고 신뢰 있는 결과를 보여주기도 하였다 (Kim et al., 2018). 따라서, Tropilaelaps 검출 시료로서 봉변의 사용은 Tropilaelaps 특이 유전자의 정량을 통해 그 벌통에 Tropilaelaps 수를 추정할 수 있기에 봉군의 모니터링이 용이할 뿐만 아니라, 그 존재의 신뢰성을 증가시켜 주었다.
봉변 시료를 사용한 Tropilaelaps 특이 유전자의 정량 검출법은 육안상 발견이 힘든 점을 극복하고자 간편하고 더 빠른 유전자 검사법을 적용한 방법이며, 꿀벌의 외부 기생충 Tropilaelaps의 존재를 꿀벌이 아닌 봉군의 봉변 시료에서 확인함으로써, 더 정확하고 간편함을 보여주었다. 꿀벌 기생충 확인의 수단으로서 봉변의 사용이 실제 양봉 농가에 만연해 있는 Tropilaelaps에 의한 피해 이전에 빠른 검출을 봉변에서 확인하여 그에 따른 빠른 제어가 가능할 것으로 기대한다. 또한 본 연구에서는 Tropilaelaps의 한 종에 국한된 것이 아닌 Tropilaelaps 속 그 자체의 특이 검출을 다루었으며, 추후 4종을 각각 특이적으로 검출할 수 있는 primer 설계를 통해 종 분류가 가능한 유전자 검사법 개발이 가능할 것으로 사료된다.
Acknowledgments
본 연구는 2018학년도 농촌진흥청 연구사업 (세부 과제번호: PJ01408002) 지원에 의하여 수행되었으며, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 농생명기술개발 사업 (과제번호 318093-03), 수출전략기술개발사업 (과제번호 115067-02) 및 첨단생산기술개발사업 (과제번호 115058-02)에 의한 연구 결과물임을 밝힙니다.
References
-
Anderson, D. L., (1994), Non-reproduction of Varroa jacobsoni in Apis mellifera colonies in Papua New Guinea and Indonesia, Apidologie, 25, p412-421.
[https://doi.org/10.1051/apido:19940408]

-
Anderson, D. L., and M. J. Morgan, (2007), Genetic and morphological variation of bee-parasitic Tropilaelaps mites (Acari: Laelapidae): new and re-defined species, Exp. Appl. Acarol., 43, p1-24.
[https://doi.org/10.1007/s10493-007-9103-0]

- Bailey, L., and B. V. Ball, (1991), Honey Bee Pathology, Academic Press, London, p193.
-
Burgett, M., P. Akratanakul, and R. A. Morse, (1983), Tropilaelaps clareae: a parasite of honeybees in south-east Asia, Bee World, 64, p25-28.
[https://doi.org/10.1080/0005772x.1983.11097904]

-
Dainat, B., T. Ken, H. Berthoud, and P. Neumann, (2009), The ectoparasitic mite Tropilaelaps mercedesae (Acari, Laelapidae) as a vector of honeybee viruses, Insectes Soc., 56, p40-43.
[https://doi.org/10.1007/s00040-008-1030-5]

- Delfinado, M., and E. W. Baker, (1961), Tropilaelaps, a new genus of mites from the Philippines (Laelapidae s. lat.), Acarina, Fieldana: Zool., 44, p53-56.
- Delfinado-Baker, M., and K. Aggarwal, (1987), Infestation of Tropilaelaps clareae and Varroa jacobsoni in Apis mellifera ligustica colonies in Papua New Guinea, Am. Bee J., 127, p443.
- Delfinado-Baker, M., and E. W. Baker, (1982), A new species of Tropilaelaps parasitic on honey bees, Am. Bee J., 122, p416-417.
-
de Guzman, L. I., G. R. Williams, K. Khongphinitbunjong, and P. Chantawannakul, (2017), Ecology, Life History, and Management of Tropilaelaps Mites, J. Econ. Entomol., 110, p319-332.
[https://doi.org/10.1093/jee/tow304]

-
Kim, J. M., A. T. Truong, S. M. Kim, B. H. Kim, M. J. Kim, and B. S. Yoon, (2018), Multi-Ultra-Rapid PCR against Viral Pathogens of Honeybee using Hive Debris, J. Apic., 33, p135-147.
[https://doi.org/10.17519/apiculture.2018.09.33.3.135]

- Kumar, N. R., R. Kumar, I. Mbaya, and R. W. Mwangi, (1993), Tropilaelaps clareae found on Apis mellifera in Africa, Bee World, 74, p101-102.
-
Laigo, F. M., and R. A. Morse, (1968), The mite Tropilaelaps clareae in Apis dorsata colonies in the Philippines, Bee World, 49, p116-118.
[https://doi.org/10.1080/0005772x.1968.11097211]

- Lee, J. H., and K. S. Woo, (1995), The Study on the Mites Inhabiting the Bee-hives in Korea II, Korean J. Apiculture, 10, p29-34.
-
Luo, Q., T. Zhou, Q. Wang, P. Dai, Y. Wu, and H. Song, (2011), Identification of Tropilaelaps mites (Acari, Laelapidae) infesting Apis mellifera in China, Apidologie, 42, p485-498.
[https://doi.org/10.1007/s13592-011-0028-6]

-
Matheson, A., (1996), World bee health update 1996, Bee World, 77, p45-51.
[https://doi.org/10.1080/0005772x.1996.11099281]

- Otis, G. W., and J. Kralj, (2001), Parasitic brood mites not present in North America, p251-272, in Mites of the honey bee T. C. Webster eds. by, and K. S. Delaplane, Dadant and Sons, Illinois.
- Pettis, J. S., R. Rose, E. M. Lichtenberg, P. Chantawannakul, N. Buawangpong, W. Somana, P. Sukumalanand, and D. Vanengelsdorp, (2013), A rapid survey technique for Tropilaelaps mite (Mesostigmata: Laelapidae) detection, J. Econ. Entomol., 106, p1535-1544.
-
Sammataro, D., U. Gerson, and G. Needham, (2000), Parasitic mites of honey bee: life history, implications and impact, Annu. Rev. Entomol., 45, p519-548.
[https://doi.org/10.1146/annurev.ento.45.1.519]

- Schmid-Hempel, P., (1998), Parasites in Social Insects, Princeton, New Jersey, p392, (Princeton University Press edition).
-
Woyke, J., (1984), Survival and prophylactic control of Tropilaelaps clareae infesting Apis mellifera colonies in Afghanistan, Apidologie, 15, p421-434.
[https://doi.org/10.1051/apido:19840405]


