
벌꿀의 잔류 DNA에서 사탕수수 또는 사탕무 고유 유전자 검출을 위한 표준 유전자 검사법
Abstract
To distinguish between natural honey and adulterated honey, standard protocols for SugarCane-specific detection PCR and nested PCR or SugarBeet-specific detection PCR and nested PCR against residual DNA isolated from honey were established. SugarCane-specific detection PCR and/or SugarBeet-specific detection PCR were performed with residual DNA isolated from honey samples, as initial template. Then, SugarCane-specific nested PCR and/or SugarBeet-specific nested PCR were also performed with 1/10,000 part of dilution from 1st round PCR (detection PCR) products of same honey, as initial template. Every, each materials or reagents, and experimental conditions for this kinds of PCRs were described in detail with case-studies for 8 honey samples. The proposed protocols would be expected to be widely used as standard method for the analysis of residual DNA from honey.
Keywords:
Honey, Residual DNA, sugar-cane; sugar-beet, Ultra-rapid PCR, Nested PCR서 론
벌꿀에는 다양한 출처의 유전자 (DNA)가 혼입되어 보존되고 있으며, 밀원의 화분 유전자를 비롯하여 꿀벌 고유의 유전자 및 꿀벌 장내 미생물 유전자, 그리고 환경에서 유래된 미생물 유전자 등 꿀벌이 벌꿀을 생산하는 과정에서 접촉되는 모든 생물체의 흔적이 남아 있게 된다 (Ruppert et al., 2019). 이들 유전자 중 가장 많은 것은 화분유전자 (pollen DNA)이며, 이들은 대부분 화분의 형태로 벌꿀에 포함되어 있으나, 그 외의 것은 분해되어 DNA 분자의 형태 (naked DNA fragments)로 잔류되게 되며, 이 유전자들을 벌꿀의 잔류 유전자 (Residual DNA)라 한다. 잔류 유전자는 벌꿀뿐 아니라 다른 연구에도 사용되고 있다 (Wang et al., 2012; Zheng et al., 2019). 꿀벌이 벌꿀을 생산하는 과정에서 밀원이 되는 식물체들의 화분들은 자연스럽게 벌꿀 내 축적이 되며, 꽃이 피는 시기에 따라 벚꽃꿀, 아카시아꿀, 밤꿀의 순서로 해당 화분들도 벌꿀 내 일정 수준의 개별 함량을 보이고 있다. 최근 화분 DNA의 특정 유전자를 이용하여 화분의 정량을 시도하고 있다 (Bell et al., 2016; de Vere et al., 2017; Baksay et al., 2020). 사탕수수 설탕 또는 사탕무 설탕에 의해 만들어진 사양벌꿀은, 천연 벌꿀 (Natural honey)의 가치를 크게 위협하고 있으며, 특히 국내 주요 벌꿀인 아카시아벌꿀에 대한 신뢰를 크게 해치고 있다. 설탕은 Sucrose 함량이 99.99%로 표시될 정도로 높은 순도를 자랑하고 있으나, 설탕의 제조가 식물체 사탕수수 줄기 또는 사탕무의 뿌리를 수집, 세척, 분쇄하고, 이하 정제과정을 거치게 되는 것으로, 이 과정에서 사탕수수, 사탕무의 식물체 고유유전자는 대부분 제거되나, 극미량의 고유 유전자는 최종 제품인 설탕 (분말)에 잔류하게 되는 것으로 추론된다. 사양꿀의 제조에서, 꿀벌에게 설탕물을 사양하게 되면, 극미량의 사탕수수 또는 사탕무 고유유전자들도 벌꿀로 혼입되며, 이들 유전자들은 벌꿀의 보존성에 의해 생산된 벌꿀 제품에 그대로 잔류되게 된다.
근래 PCR 기술의 발전은 35회전의 PCR에 2시간이 소요되던 2000년대 초반에 비하여, 50회전의 PCR을 10분대에 완료하는 수준에 이르렀다 (Kim et al., 2019; Kim et al., 2020; Troung et al., 2021a, b). 이런 신속성은 연속된 2차례의 PCR을 수행하는, 즉 1차 PCR산물에 대한 PCR인 nested PCR (2차 PCR)까지 30분 이내에 왼료할 수 있게 되어, nested PCR의 활용은 극미량의 특이유전자에 대한 민감성 (sensitivity) 및 정확성 (accuracy)을 비약적으로 높일 수 있게 되었다 (Kim et al., 2018a; Kim et al., 2018b; Trong et al., 2021c).
본 연구는 사탕수수 및 사탕무 특이 유전자의 증폭법을 처음 발표한 이래 (Kim et al., 2018a; Kim et al., 2018b), 다양한 벌꿀 시료에 유전자 검사법을 적용한 결과들을 바탕으로, 사탕수수 유전자 및 사탕무 유전자의 검출 및 정량을 위한 1차 (detection PCR), 2차 (nested PCR) PCR법을 실용적으로 정리한 것이다. 이 실험법을 사양꿀과 천연꿀의 판별을 위한 벌꿀의 유전자 검사에 표준으로, 또는 실용적 protocol로 제시하고자 한다.
재료 및 방법
1. 국내 벌꿀 8종의 수집
국내에서 생산된 8종의 벌꿀을 수집하였다. 8종 중 6종은 구매한 것으로 서양꿀벌에 의해 생산된 아카시아벌꿀, 밤꿀, 잡화꿀 등이었으며, 2종은 토종벌에 의해 생산된 토종꿀이었다. 또한, 100% 사양꿀이라 표기된 사양꿀을 별도 구매하였으며, 사탕수수 특이 PCR 검사에서 검출 여부를 확인하였다. 그 외 본 연구진에 검사가 의뢰된 벌꿀들도 시료로 사용되었다 (Supplement Data).
2. 사탕수수 고유 유전자를 탑재한 표준물질 (standard DNA)
사탕수수 특이 PCR법의 표준물질 (Standard material)로서 사탕수수 고유유전자를 탑재한 plasmid, p-SugarCane-cp (pSC)를 사용하였다. 이 재조합 DNA는 사탕수수 (Sugar Cane; Saccharum officinarum) 엽록체에 위치한 maturase K (matK, GenBank accession No. LN 849913) 유전자를 pGEM-Vector에 탑재한 것으로, 사탕수수 씨앗의 DNA를 주형으로, cloning용 프라이머, Cane-cp-F (5′-GGATTAAATGGTTCCGAACCTG), Cane-cp-R (5′-TTCCGAAAAAACCCCGGGTA)의 증폭된 698 bp의 DNA를 클론화한 것이다 (Kim et al., 2018a).
3. 사탕무 고유 유전자를 탑재한 표준물질 (standard DNA)
사탕무 특이 PCR법의 표준물질 (Standard material)로서 사탕무 고유유전자를 탑재한 plasmid, p-SugarBeet-mt (pSB)를 사용하였다. 이 재조합 DNA는 사탕무 미토콘드리아 (Mitochondria)에 위치한 250 bp의 유전자 (GenBank accession No. BA000024.1)를 pGEM-Vector에 탑재한 것이다 (Kim et al., 2018b). 또한, pTOP-Beet-RdDp315는 사탕무의 genomic DNA에 위치한 RdDP (RNA dependent DNA Polymerase; GenBank Accession no. EF101866) 유전자를 탑재한 재조합 DNA로, 사탕무 고유 유전자 검출에 또 다른 표준물질로 사용되었다.
사탕무의 genomic DNA는 설탕 제조과정 중 naked DNA로 분쇄되어 설탕에 잔류된다. 이는 사양꿀의 잔류 유전자를 분리하는 과정에서 mitochondrial DNA 또는 chloroplast DNA보다 높은 수율로 분리되기에 표준물질로 채택되었다.
한편, 사탕무는 설탕의 제조에서 식물체의 뿌리부위를 사용하며, 사탕수수는 식물체의 줄기부위를 사용한다. 식물세포의 3종 DNA, 즉 Genomic, Mitochodria, Chloroplast DNA는 잎, 줄기, 뿌리에 따라 그 수가 다르며, 일례로 사탕무의 뿌리세포의 Chloroplast DNA는 상대적으로 매우 소수이며, 이는 벌꿀의 잔류 DNA 중 상대적으로 가장 낮은 분자수를 기록하였다.
4. 벌꿀시료에서 잔류 유전자 (residual DNA)의 분리
벌꿀에 잔류되어 있는 잔류 유전자 (residual DNA)는 극미량으로 1 mL의 꿀에 1 ng 이하의 양이 분리되며, spectrophotometer의 260 nm 파장에서 간섭파장 때문인지 신뢰성 있게 정량되지 않는다. 본 연구는 김 등 (2021)의 방법을 기본으로 2 mL의 벌꿀에서 잔류 DNA를 분리하여 그 중 1 μL를 각 detection PCR에 initial DNA로 사용하였다. 약술하면, 벌꿀시료를 5배양의 DNA binding buffer (5 M guanidine hydrochloride, 30% isopropyl alcohol)에 완전히 용해시킨 후, affnity column을 통과시키고, 이를 700 μL washing buffer (10 mM Tris-HCl pH 7.6, 80% ethanol)로 세척한 후, 50 μL 증류수로 용출시키는 것이다.
5. 사탕수수 고유 유전자 증폭을 위한 프라이머들
실험된 많은 프라이머쌍들 중 사탕수수 고유 유전자를 PCR 증폭하기 위한 검출용 (1차 PCR) 프라이머로 Cane-cp-dF/dR이 선발되었다. 또한, 1차 PCR 산물을 재증폭하기 위한 2차 PCR (Nested PCR) 프라이머로 Cane-cp-nF/nR이 선발되었다 (Table 1).
6. 사탕무 고유 유전자 증폭을 위한 프라이머들
실험된 많은 프라이머쌍들 중 사탕무 고유 유전자를 PCR 증폭하기 위한 검출용 (1차 PCR) 프라이머로 Beet-mt-dF/dR이 선발되었다. 이 경우 표준물질은 p-SugarBeet-mt (pSB)를 사용되며, 1차 PCR 산물을 재증폭하기 위한 2차 PCR (Nested PCR) 프라이머는 Beet-mt-nF/nR가 선발되었다 (Table 2).
또한, pTOP-Beet-RdDp315를 사탕무 고유 유전자의 표준물질로 사용하는 경우, 사탕무 고유 유전자를 PCR증폭하기 위한 검출용 (1차 PCR) 프라이머로 Beet-RdDP-dF/dR이 선발되었으며, 1차 PCR 산물을 재증폭하기 위한 2차 PCR (Nested PCR) 프라이머로 Beet-RdDP-nF/nR이 선발되었다 (Table 3).
7. 사탕수수 또는 사탕무 고유 유전자 증폭을 위한 초고속 PCR의 조성
사탕수수 및 사탕무 고유 유전자 검출을 위한 초고속 PCR은 1) Cane-specific detection PCR, 2) Beet-specific detection PCR, 3) Cane-specific Nested PCR, 4) Beet-specific Nested PCR의 4종 PCR을 순차적으로 실시하는 것이다. Detection PCR에서 주형 DNA 용액은, 표준 DNA 또는 벌꿀시료에서 분리한 DNA 용액을 사용하였으며, 한편 Nested PCR에서 주형 DNA용액은 해당 PCR 산물을 1/1000 또는 1/10000으로 희석하여 그 희석액 1 μL를 사용하였다. 초고속 PCR에서 각 개별 PCR의 총량은 10 μL이었으며, 증류수 2 μL에 주형 DNA를 1 μL 넣어주고, 이에 PCR 종류에 따른 Primer 들, 즉 forward primer와 reverse primer를 각 1 μL (10 pmole/μL) 씩을 넣고, 이에 5 μL 2×Rapi:Detect Master mix (Genesystem Co., Korea)을 넣어 총 10 μL로 조성하였다.
8. 사탕수수 또는 사탕무 고유 유전자 증폭을 위한 4종 초고속 PCR의 조건들
초고속 PCR은 Genechecker II (Genesystem, Korea)를 사용하였으며, 이 기기에 사용되는 PCR-Chip은, 각 10 μL씩인 10개 PCR이 동시에, 같은 온도 및 시간조건으로 운용할 수 있게 되었다. 사탕수수 및 사탕무 고유 유전자 검출을 위한 초고속 PCR의 온도 및 시간에 대한 일반조건은 초기변성 95℃ 30 sec 후, 95℃ 변성 3 sec, 55℃ 혼성 3 sec, 72℃ 중합 3 sec를 1회전으로 하여 총 50회전을 수행하는 것이다. 이는 10개의 개별 PCR을 4종 프라이머쌍들을 한번의 초고속 PCR로 동시에 수행 (같은 온도 및 시간)하였을 때 사용되었다.
한편 초고속 PCR에서 각 PCR의 종류에 따라 최적 온도 및 시간 조건은 조금씩 상이하였으며, 특히 혼성 온도 및 시간은 53℃ 혼성 4초 (beet-mt-detection PCR), 또는 55℃ 혼성 4초 (cane-detection PCR; cane-nested PCR), 57℃ 혼성 4초 (beet-RdDP-nested PCR), 59℃ 혼성 3초 (beet-RdDP-detection PCR)에서 보다 높은 증폭성능을 보여 주었다. 그러나, 전술한 일반 공통조건으로 4종 초고속 PCR을 모두 운용하더라도 PCR 증폭에 큰 차이는 보이지 않았으며, 같은 조성에서 약간의 Ct 값 감소를 보여주었다. 또한, 초고속 PCR에서 각 온도단계 (step)의 반응시간을 3초에서 4초로 연장하는 것은 유리한 것으로 보였으나, 5초 이상의 반응시간은 불필요한 것으로 판정되었다.
9. 일반 PCR 기기를 사용한 사탕수수 또는 사탕무 고유 유전자 증폭
일반 PCR 기기를 사용하여 본 검출법이 적용될 수 있는지 여부를 판단하고자, 일반 PCR 기기인 PTC-200 Peltier thermal cycler (MJ Research, USA), 그리고 ExicyclerTM 96 (Bioneer, Korea)를 사용하여 사탕무/사탕수수 고유 DNA 증폭을 시행하였다. PCR 조건은 초기변성 95℃에서 5분 진행한 후, 95℃ 변성 30초, 55℃ 혼성 30초, 72℃ 중합 30초를 1 cycle로 하였으며, 총 cycle 수는 40 cycle로 설정하였다. 각 PCR 반응액은 DNA 용액 중 1 μL를 주형으로 사용하였으며, Cane-specific Detection or nested PCR의 Cane-cp-dF or nF/dR or nR , Beet-specific Detection or nested PCR의 Beet-mt-dF or nF/dR or nR을 각 1 μM (최종농도)로 조정하여 주었고, AccuPower® 2X GreenStarTM qPCR Master Mix (Bioneer, Korea)을 사용하였다.
결과 및 고찰
1. Beet-specific Detection PCR의 특이성 및 정량성
표준물질 p-SugarBeet-mt (pSB)를 계단 희석하여 초고속 PCR을 수행하였다. 희석된 표준물질은 initial template로 2.1개 특이 분자를 주어졌어도 특이 증폭되었으며 (Ct, 31.71 cycles; Tm, 78.82℃), initial template 103~108 분자 구간에서 우수한 정량성이 인정되었다 (Fig. 1).
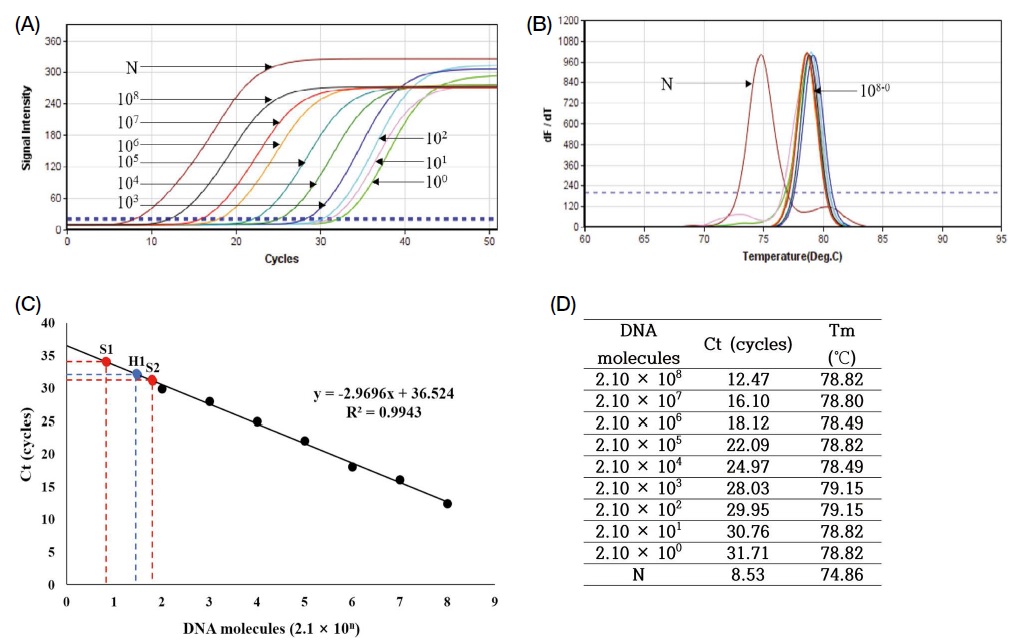
Beet-specific, quantitative detection PCR with 10-fold diluted standard DNA, pSugarBeet-mt (pSB). Beet-specific quantitative PCR were performed with 10-fold diluted standard DNA, pSugarBeet-mt (pSB). Panel A shows amplification curves, N is negative control using no template. Panel B shows analysis of melting-point. Apex of each peak (dF/dT) indicates each mid-point of melting temperature (Tm) of each PCR product. Panel C shows regression analysis. Regression coefficiency was calculated, as R2=0.9943. Panel D comprised the number of molecules of initial template, Threshold cycles (CT) in each PCR. In the range of 103~108 molecules of initial template show acceptable quantities for quantitative analysis.
2. 8개 벌꿀시료의 잔류 DNA에 대한 Cane-specific Detection PCR
벌꿀 내 사탕수수 고유 유전자가 존재하는지 여부를 가리기 위하여, 국내 8종의 벌꿀을 수집하여 벌꿀시료로 사용하였다. 벌꿀에서 잔류 DNA를 분리하였고 (Kim et al., 2021), 이들 DNA에 대한 Cane-specific Detection PCR을 수행하였다 (Fig. 2).
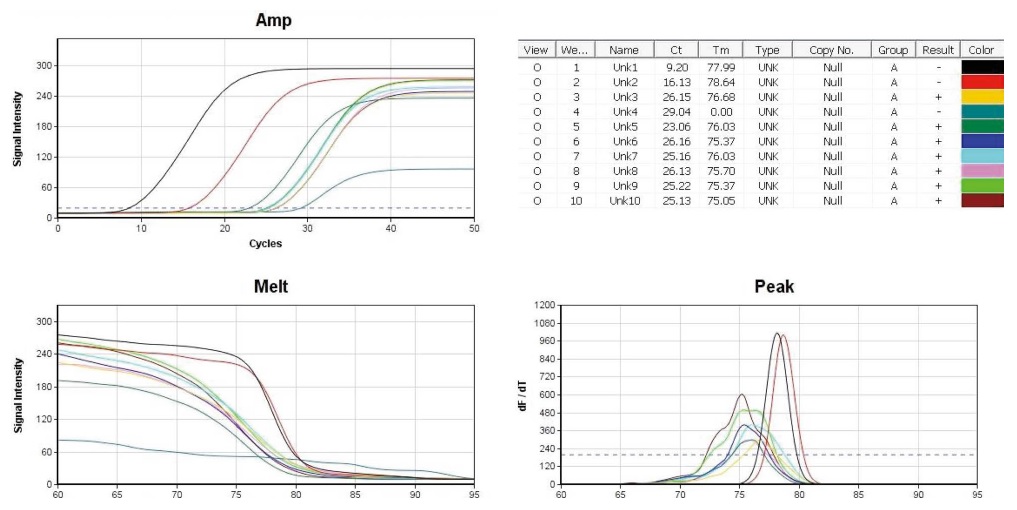
Cane-specific detection PCRs with 8 residual DNA from 8 different honey. The conditions of temperature/incubation-time in cane-detection PCR were 95℃ 4 sec, 55℃ 4 sec, 72℃ 4 sec (50 cycles). 10 pmole Cane-cp-dF/dR primers were used in each PCR. Lane 1 (positive control), 1 ng standard DNA p-Cane-cp (pSC) was used as initial template; lane 2 (positive control), 0.01 ng pSC. In lane 3~10, residual DNA from each honey sample was used as initial template. Left-high, amplification curves of 10 PCRs; Right-high, CT and Tm; Left-low, melting-point analysis; Right, peak-curve (dF/dT).
Cane-detection PCR은 95℃ 4 sec, 55℃ 4 sec, 72℃ 4 sec (50회전)의 조건에서, 융점분석 완료까지 총 24분 22초가 소요되었으며, positive control인 lane 1과 2는 Ct (Threshold cycles)가 각기 9.20 cycles, 16.13 cycles로 나타나 좋은 정량성을 보여주었다. Tm (Temperature of Mid-point)은 각기 77.99℃, 78.64℃로 측정되어, 같은 특이 PCR product임을 보여주었다 (차는 0.65℃). 한편, 벌꿀시료의 DNA를 기질로 사용한 lane 3~10의 일부는, 증폭곡선에서 Ct 값이 23~26 cycles 수준으로 증폭된 듯 보이나, 융점곡선에서 온도의 증가에 따른 형광강도의 기울기가 positive control인 lane 1과 2의 그것과 분명히 구분되며, 피크곡선에서도 lane 1, 2 (Tm평균 78.32℃)와 lane 3~10 (Tm: 75~76℃)은 분명히 다른 Tm값으로 계산되었다.
Cane-detection PCR에서, 이 측정값들은 lane 1, 2를 제외한. lane 3~10의 PCR 산물들이 비특이 증폭임을 나타내고 있으며, 이 PCR산물들을 Agarose gel 전기영동으로 확인하였다 (Fig. 3).
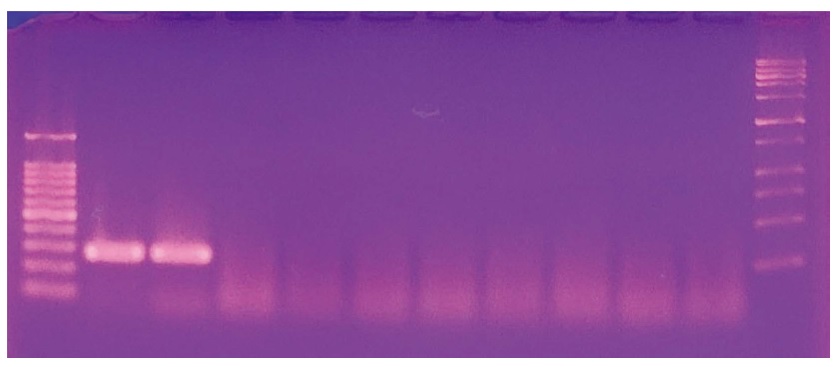
Agarose gel electrophoresis of 8 different cane-specific detection PCRs. 10 cane-specific detection PCR products were separatedly migrated in 2% Agarose gel, 1×TAE buffer, 50 volt, 45 min long. Left DNA size marker shows 100, 200, 300, 400 bp, etc, right DNA size marker indicates 250, 500, 750 bp etc from lowest band. As positive control, 271 bp long-cane specific PCR products were shown in lane 1 and 2. In lane 3~10 non-specific PCR products were shown.
3. 8개 벌꿀시료의 잔류 DNA에 대한 Beet-specific Detection PCR
벌꿀 내 사탕무 고유 유전자가 존재하는지 여부를 가리기 위하여, 국내 8종의 벌꿀을 수집하여 벌꿀시료로 사용하였다. 벌꿀에서 잔류 DNA를 분리하였고 (Kim et al., 2021), 이 DNA들 (Cane Detection PCR과 같은 시료)에 대한 Beet-specific Detection PCR을 수행하였다 (Fig. 4).
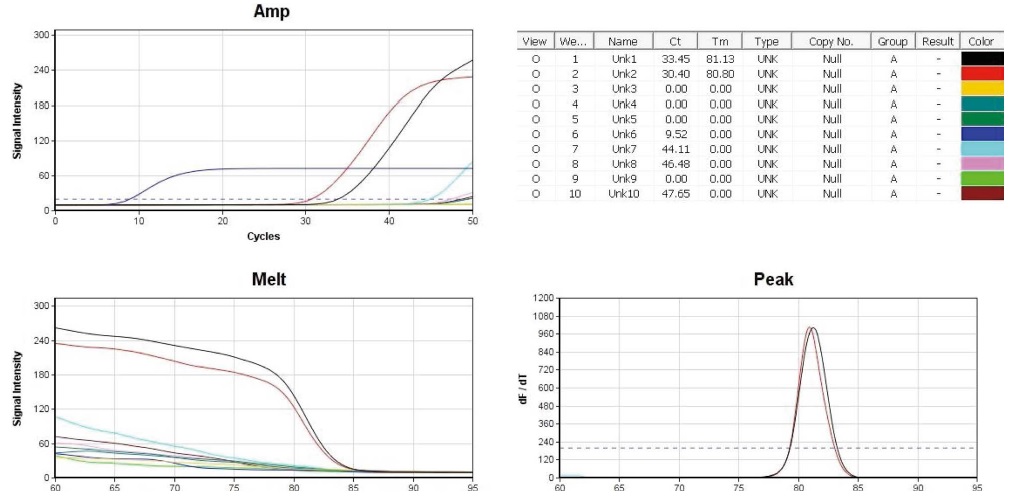
Beet-specific detection PCRs with 8 different residual DNA from 8 honey. The conditions of temperature/incubation-time in beet-detection PCR were 95℃ 4 sec, 59℃ 4 sec, 72℃ 4 sec (50 cycles). 10 pmole Beet-RdDP-dF/dR primers were used in each PCR. Lane 1 (positive control), 1 ng standard DNA pTOP-Beet-RdDp315 was used as initial template; lane 2 (positive control), 0.01 ng pTOP-Beet-RdDp315; In lane 3~10, residual DNA from each honey sample was used as initial template. Left-high, amplification curves of 10 PCRs; Right-high, CT and Tms; Left-low, melting-point analysis; Right-low, peak-curve (dF/dT).
Beet-detection PCR은 95℃ 4 sec, 59℃ 4 sec, 72℃ 4 sec (50회전)의 조건에서, 융점분석 완료까지 총 26분 11초가 소요되었으며, positive control인 lane 1과 2는 Ct (Threshold cycles)가 각기 33.45 cycles, 30.40 cycles로 나타나 표준물질의 농도가 정확한 것이었는지 의심이 들었으나, Tm (Temperature of Mid-point)은 각기 81.13℃, 80.80℃로 측정되어, 같은 특이 PCR 산물임을 보여주었다 (차는 0.33℃). 한편, 8종 벌꿀시료의 DNA를 기질로 사용한 lane 3~10의 일부는, 증폭곡선에서 Ct 값이 45 cycles 이후의 수준으로 증폭된 듯 보이나, 융점곡선에서 온도의 증가에 따른 형광강도의 기울기가 positive control인 lane 1과 2의 그것과 분명히 구분되며, 피크곡선에서 lane 1, 2 (Tm평균 80.97℃)와 lane 3~10 (Tm 모두 측정되지 않음; 0.00℃)은 분명히 다른 Tm 값으로 계산되었다.
Beet-detection PCR에서, 이 측정값들은 표준물질을 사용한 lane 1, 2를 제외한, lane 3~10의 PCR 산물들이 비특이 증폭임을 나타내고 있으며, 이 PCR산물들을 Agarose gel 전기영동으로 확인하였다 (Fig. 5).

Agarose gel electrophoresis of 8 different Beet-specific detection PCRs. 10 beet-specific detection PCR products were separatedly migrated in 2% Agarose gel, 1×TAE buffer, 50 volt, 45 min long. Left DNA size marker shows 100, 200, 300, 400 bp, etc, right DNA size marker indicates 250, 500, 750 bp etc from lowest band. As positive control, 315 bp long-beet specific PCR products wete shown in lane 1 and 2. Non-specific PCR products were shown in lane 3~10.
4. 8개 벌꿀시료의 Cane detection PCR product에 대한 Cane-specific Nested PCR
Cane-specific Nested PCR은, 국내 8종 벌꿀 시료에서 잔류 DNA를 분리하고, 각기 Cane Detection PCR (Fig. 2)을 수행하고, 그 PCR 산물들을 각기 1/10000로 희석하여, 희석액 1 μL를 주형으로 사용한 것이다 (Fig. 6).
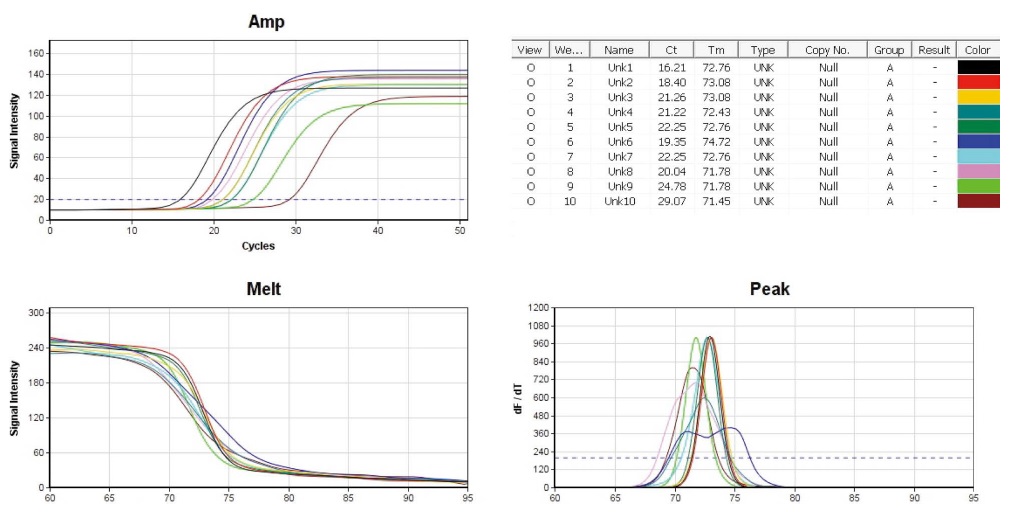
Cane-specific Nested PCRs with 8 different Cane-specific Detection PCRs. The conditions of temperature/incubation-time in cane-nested PCR were 95℃ 4 sec, 55℃ 4 sec, 72℃ 4 sec (50 cycles). 10 pmole Cane-cp-nF/nR primers were used in each PCR. Lane 1 (positive control), 1 ng standard DNA p-Cane-cp (pSC) was used as initial template; lane 2 (positive control), 0.01 ng pSC. In lane 3~10, each 1 μL of 1/10000 diluted Cane Detection PCR product (Fig. 2). Left-high, amplification curves of 10 PCRs; Right-high, CT and Tm; Left-low, melting-point analysis; Right-low, peak-curve (dF/dT).
Cane-Nested PCR은 95℃ 4 sec, 55℃ 4 sec, 72℃ 4 sec (50회전)의 조건에서, 융점분석 완료까지 총 24분 22초가 소요되었으며, positive control인 lane 1과 2는 Ct (Threshold cycles)가 각기 16.21 cycles, 18.40 cycles로 나타나 정량성을 보여주었고, Tm (Temperature of Mid-point)은 각기 72.76℃, 73.08℃로 측정되어, 같은 특이 PCR 산물임을 보여주었다 (차는 0.32℃). Cane-Nested PCR의 initial template는, 벌꿀시료의 DNA를 기질로 사용한 Cane Detection PCR (Fig. 2)의 lane 3~10 산물을 각기 1/10000 희석한 희석액 1 μL이었으며, 같은 순서로 Cane-Nested PCR의 lane 3~10을 수행하게 하였다. Lane 3~10은 증폭곡선에서 19.35 cycles에서 29.07 cycles까지 다양한 Ct 값으로 증폭된 듯 보였으며, 증폭선의 형태가 lane 1, 2와 유사하였고, 융점곡선에서도 온도의 증가에 따른 형광강도의 기울기가 positive control인 lane 1과 2의 그것과 매우 유사하게 나타났다. 피크곡선에서 lane 1, 2의 Tm 값 평균인 72.92℃와 유사하지 않은 것은 lane 6 (Ct, 19.35 cycles; Tm, 74.72℃)뿐이었다.
Cane Nested PCR에서 나타난 측정값들은 lane 3에서 lane 10까지 모두 사탕수수 특이 유전자의 증폭을 나타내고 있었으나, lane 6 은 Tm (74.72℃)이 표준 DNA의 평균 (72.92℃)과 1.80℃의 차를 보이기에, 개별검증을 수행하였다 (Fig. 7).
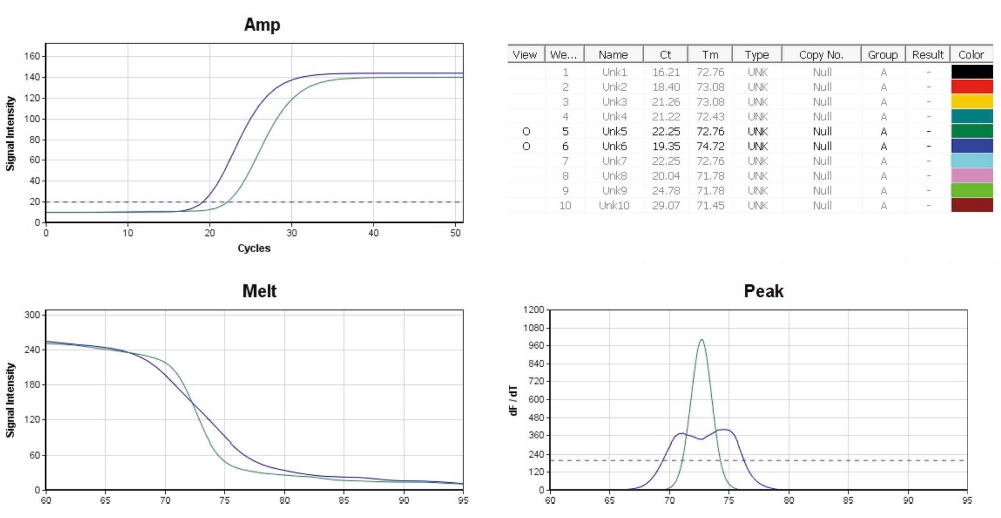
Cane-specific Nested PCRs with 2 different Cane-specific Detection PCRs. The conditions of temperature/incubation-time in cane-nested PCR were 95℃ 4 sec, 55℃ 4 sec, 72℃ 4 sec (50 cycles). 10 pmole Cane-cp-nF/nR primers were used in each PCR. Lane 5 (green line), lane 6 (blue line) were performed with each 1 μL of 1/10,000 diluted lane 5, lane 6 PCR products, respectively, as initial template in Cane Detection PCR (Fig. 2), Left-high, amplification curves of 2 PCRs; Right-high, CT and Tm; Left-low, melting-point analysis; Right-low, peak-curve (dF/dT). In peak-curve of lane 6, double peaks were observed.
또한, Cane Nested PCR의 각 산물들은 전기영동으로 과연 137 bp 크기의 사탕수수 특이 유전자의 여부를 확인할 수 있다. 따라서 이 Cane Nested PCR의 PCR 산물들은 Agarose gel 전기영동으로 PCR에 측정치에 의한 판정을 재확인하였다 (Fig. 8).

Agarose gel electrophoresis of 8 different Cane-specific Nested PCRs. 10 cane-specific nested PCR products were separatedly migrated in 2% Agarose gel, 1×TAE buffer, 50 volt, 60 min long. Left and right DNA size marker shows 100, 200, 300, 400 bp, etc from lowest band. As positive control, 137 bp long cane specific nested PCR products were shown in lane 1 and 2. Also, 137 bp long PCR products were observed in lane 3, 4, 5, 7, 8, 9 and 10, except lane 6.
Cane Nested PCR의 측정값들에 의한 판정은 lane 6을 제외한 lane 3에서 lane 10까지 모두 양성이었으나, PCR산물의 전기영동에 의한 판정은 lane 6의 음성 (검출되지 아니함)을 다시 확인시켜주는 것이었다. 따라서, Cane Nested PCR의 각 lane에 대한 정밀한 개별분석과 더불어 아가로우스 젤 상에서 분명한 137 bp 크기의 사탕수수 특이 유전자 산물을 확인하는 것이 정확한 판정을 위하여 요구된다 할 것이다.
Cane Nested PCR의 결과에 따라, 벌꿀시료 중 사탕수수 고유 유전자의 존재가 확인되는 것은 lane 3, 4, 5, 7, 8, 9, 10이라 최종 판정될 수 있을 것이다.
5. 8개 벌꿀시료의 Beet detection PCR product에 대한 Beet-specific Nested PCR
Beet-specific Nested PCR은, 국내 8종 벌꿀 시료에서 잔류 DNA를 분리하고, 각기 Beet Detection PCR (Fig. 4)을 수행하고, 그 PCR산물들을 각기 1/10,000로 희석하여, 희석액 1 μL를 주형으로 사용한 것이다 (Fig. 9).
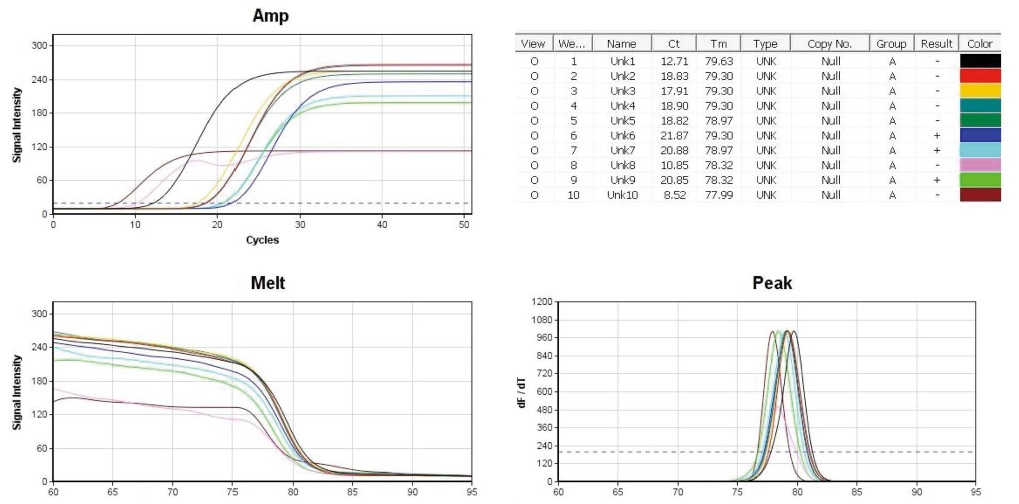
Beet-specific Nested PCRs with 8 different Beet-specific Detection PCR. The conditions of temperature/incubation-time in Beet-nested PCR were 95℃ 4 sec, 57℃ 4 sec, 72℃ 4 sec (50 cycles). 10 pmole Beet-RdDP-nF/nR primers were used in each PCR. Lane 1 (positive control), 1 ng standard DNA pTOP-Beet-RdDp315 was used as initial template; lane 2 (positive control), 0.01 ng In pTOP-Beet-RdDp315. lane 3~10, each 1 μL of 1/1000 diluted Beet Detection PCR product (Fig. 4). Left-high, amplification curves of 10 PCRs; Right-high, CT and Tm; Left-low, melting-point analysis; Right-low, peak-curve (dF/dT).
Beet-Nested PCR은 95℃ 4 sec, 57℃ 4 sec, 72℃ 4 sec (50회전)의 조건에서, 융점분석 완료까지 총 25분 45초가 소요되었으며, positive control인 lane 1과 2는 Ct (Threshold cycles)가 각기 12.71 cycles, 18.33 cycles로 나타나 정량성을 보여주었으며, Tm (Temperature of Mid-point)은 각기 79.63℃, 79.30℃로 측정되어, 같은 특이 PCR 산물이 만들었음을 보여주었다 (차는 0.33℃).
한편, 벌꿀시료의 DNA를 기질로 사용한 Beet Detection PCR (Fig. 4)의 lane 3~10 산물을 1/10000 희석하여 initial template로 사용한 본 Beet-Nested PCR의 lane ~10은 증폭곡선에서 다양한 Ct 값으로 증폭된 듯 보였으며, 융점곡선에서도 온도의 증가에 따른 형광강도의 기울기가 positive control인 lane 1과 2의 그것과 분명히 구분되지 아니하였고, 피크곡선에서도 lane 1, 2 (Tm평균 79.46℃)와 lane 3~10 (Tm : 77.99~79.30℃)은 매우 유사하여 같은 PCR 산물이 생성된 것으로 분석되었다.
Beet Nested PCR에서, 이 측정값들은 lane 3~10의 PCR 산물들이 모두 lane 1, 2 (positive control)과 같은 사탕무 특이 증폭임을 나타내고 있는 것으로 판단되었으며, 이는 PCR 산물들을 Agarose gel 전기영동으로 확인하였다 (Fig. 10).

Agarose gel electrophoresis of 8 different Beet-specific Nested PCRs. 10 Beet-specific nested PCR products were separatedly migrated in 2% Agarose gel, 1×TAE buffer, 50 volt, 45 min long. Left DNA size marker shows 100, 200, 300, 400 bp, etc, right DNA size marker indicates 250, 500, 750 bp etc from lowest band. As positive control, 160 bp long Beet-specific nested PCR products were shown in lane 1 and 2. Also, 160 bp long PCR products were observed in each lane of lane 3~10.
Beet Nested PCR 및 그 산물의 전기영동 결과는 검사된 8종 벌꿀시료 모두에서 160 bp 크기의 사탕무 특이 PCR 산물들이 존재하는 것으로, 이는 8종 벌꿀이 사탕무 설탕물로 사양된 사실이 있음을 입증하는 것이라 할 것이다.
6. 8개 벌꿀시료의 잔류 DNA에 대한 사탕수수 및 사탕무 유전자 검사의 판정
8종 벌꿀시료에 대한 Cane Detection PCR의 결과는 positive control인 lane 1, 2만 양성이었고, 나머지 lane 3~10은 사탕수수 특이 유전자를 검출할 수 없었다 (판정 보류). 그러나, 같은 시료에 대한 Cane Nested PCR의 결과는 명백하게 lane 3, 4, 5, 7, 8, 9 및 10이 양성임을, 즉 사탕수수 고유 유전자가 벌꿀시료 중에 존재한다는 것을 나타내며, 이는 해당 벌꿀의 생산과정에서 사탕수수 설탕물로 사양된 사실이 있음을 입증하는 것이었다.
또한, 같은 8종 벌꿀시료에 대한 Beet Detection PCR의 결과는 positive control인 lane 1, 2만 양성이었고, 나머지 lane 3~10은 사탕무 특이 유전자를 검출할 수 없었다 (판정 보류). 그러나, 같은 시료에 대한 Beet Nested PCR의 결과는 명백하게 lane 3~10 모두가 양성임을, 즉 사탕무 고유 유전자가 벌꿀시료 중에 존재한다는 것을 나타내며, 이는 해당 벌꿀의 생산과정에서 사탕무 설탕물로 사양된 사실이 있음을 입증하는 것이었다.
7. 일반 PCR 기기를 사용한 사탕수수 또는 사탕무 고유 유전자 증폭
일반 PCR 기기인 PTC-200 Peltier thermal cycler (MJ Research, USA), 그리고 ExicyclerTM 96 (Bioneer, Korea)를 사용하여 사탕수수 및 사탕무 고유 유전자 증폭을 시행한 결과는, 우선 충분히 활용할 수 있는 민감도 (sensitivity), 정확성 (accuracy)을 보인 것으로 나타났다. 그러나 예상대로 신속성 (rapidity)의 면에서 크게 뒤지는 것으로 판단되었다 (결과 미제시).
적 요
천연벌꿀을 사양벌꿀과 판별하기 위하여, 벌꿀에서 분리한 잔류 유전자에 대한 사탕수수 특이 detection PCR 및 nested PCR을, 그리고 사탕무 특이 detection PCR 및 nested PCR 을 표준 검사법으로 정형화하고자 하였다.
이 표준 검사법은 먼저 벌꿀시료에서 잔류 DNA를 분리해 내고, 이 DNA시료를 initial template로 사용하여, 사탕수수 특이 detection PCR 그리고 사탕무 특이 detection PCR을 수행하는 것이다. 그리고 해당 PCR 산물을 1/10,000 희석하여, 이 희석액을 initial template로 사탕수수 특이 nested PCR과 사탕무 특이 nested PCR을 각기 수행하는 것이다.
수천 분자 이하의 특이 분자를 기질로 사용하였던 detection PCR (1차 PCR)은 해당 특이 분자를, 형광검출기가 인지할 수 없는 수준이나마, 증폭시키기는 하였기에, 그 1차 PCR 산물을 희석하여 초기기질로 사용한 Nested PCR (2차 PCR)은 해당 특이분자를 검출이 가능한 분자수로 쉽게 증폭시킬 수 있었다.
벌꿀에서 분리한 사탕수수 또는 사탕무 고유 유전자의 PCR 검출로 해당 당원을 사양한 사양벌꿀은 보다 명백히 판정될 수 있을 것이며, 이 유전자 검사에 필요한 모든 물질과 시약, 그리고 세부 실험방법들을 8개 벌꿀에 대한 실험 예와 함께 적시하였다. 제안하는 실험법이 벌꿀의 잔류 유전자 분석의 표준 실험법으로, 국내 사양꿀 판별의 기본 실험법으로 널리 활용되기를 기대한다.
Acknowledgments
본 연구는 농촌진흥청 농업정책지원기술개발사업 (곤충기술경쟁력강화, 과제번호: PJ015778032021)과 Korea institute of Planning and Evaluation for Technology in Food, Agriculture and Forestry (IPET; 318093-03)의 지원에 의하여 수행되었으며, 이에 감사의 뜻을 표합니다.
References
-
Baksay, S., A. Pornon, M. Burrus, J. Mariette, C. Andalo and N. Escaravage. 2020. Experimental quantification of pollen with DNA metabarcoding using ITS1 and trnL. Sci. Rep. 10: 4202.
[https://doi.org/10.1038/s41598-020-61198-6]

-
Bell, K. L., N. de Vere, A. Keller, R. T. Richardson, A. Gous, K. S. Burgess and B. J. Brosi. 2016. Pollen DNA barcoding: current applications and future prospects. Genome 59: 629-640.
[https://doi.org/10.1139/gen-2015-0200]

-
de Vere, N., L. E. Jone, T. Gilmore, J. Moscrop, A. Lowe, D. Smith, M. J. Hegarty, S. Creer and C. R. Ford. 2017. Using DAN metabarcoding to investigate honey bee foraging reveals limited flower use despite high floral availability. Sci. Rep. 7: 42838.
[https://doi.org/10.1038/srep42838]

-
Kim, B. H., S. Kim, M. Kim, J. Kim, A. T. Truong and B. S. Yoon. 2018a. Detection of Sugar Cane (Saccharum officinarum)-specific Gene from Sugar and Sugar-honey. J. Apic. 33(3): 221-226.
[https://doi.org/10.17519/apiculture.2018.09.33.3.221]

-
Kim, B. H., S. Kim, M. Kim, J. Kim, A. T. Truong, K. J. Cho and B. S. Yoon. 2019. Detection of chronic bee paralysis virus using ultra-rapid PCR and nested ultra-rapid PCR. J. Apic. Res. 58(1): 133-140.
[https://doi.org/10.1080/00218839.2018.1517999]

-
Kim, J. M., S. J. Lim, S. Kim, M. Kim, B. H. Kim, A. T. Truong, S. Kim and B. S. Yoon. 2020. Rapid detection of deformed wing virus in honeybee using ultra-rapid qPCR and a DNA-chip. J. Vet. Sci. 20(1): e4.
[https://doi.org/10.4142/jvs.2020.21.e4]

-
Kim, S. M., B. H. Kim, M. Kim, J. Kim, A. T. Truong and B. S. Yoon. 2018b. Detection of Sugar Beet (Beta vulgaris)-Specific Gene from Honey Made by Sugar of Sugar Beet. J. Apic. 33(3): 213-219.
[https://doi.org/10.17519/apiculture.2018.09.33.3.213]

-
Kim, S. M., S. G. Kim, S. M. Han and B. S. Yoon. 2021. Optimization of practical protocol for residual DNA from honey (unpublished paper).
[https://doi.org/10.17519/apiculture.2021.09.36.3.195]

-
Ruppert, K. M., R. J. Kine and M. S. Rahman. 2019. Past, present, and future perspectives of enviromental DNA (eDNA) metabarcoding: a systematic review in methods, monitoring, and application of global eDNA. Glob. Ecol. Conserv. 17.
[https://doi.org/10.1016/j.gecco.2019.e00547]

- Sobrino-Gregorio, L., S. Vilanova, J. Prohens and I. Escriche. 2018. Detection of honey adulteration by conventional and real-time PCR. Food Control 95: 57-62.
-
Truong, A.-T., B. H. Kim, J. Kim, S. Kim and B. S. Yoon. 2019. Rapid detection of Israeli acute paralysis virus using multi-point ultra-rapid real-time PCR (UR-qPCR). J. Apic. Res. 58(5): 746-753.
[https://doi.org/10.1080/00218839.2019.1653424]

-
Truong, A.-T., S. Kim and B.S. Yoon. 2021c. Determination of honey adulterated with corn syrup by quantitative amplification of maize residual DNA using ultra-rapid real-time PCR. J. Sci. Food Agric. 2021.
[https://doi.org/10.1002/jsfa.11411]

-
Truong, A.-T., S. M. Kim, M. S. Yoo, Y. S. Cho and B. S. Yoon. 2021b. Susceptibility of Apis mellifera Larvae of Different Ages to Infection from Melissococcus plutonius, an European Foulbrood Disease-causing Pathogen. J. Apic. 36(2): 47-54.
[https://doi.org/10.17519/apiculture.2021.06.36.2.47]

-
Truong, A.-T., S. Sevin, S. M. Kim, M. S. Yoo, Y. S. Cho and B. S. Yoon. 2021a. Rapidly quantitative detection of Nosema ceranae in honeybees using ultra-rapid real time quantitative PCR. J. Vet. Sci. 22(3): e40.
[https://doi.org/10.4142/jvs.2021.22.e40]

-
Wang, X., D. M. Morgan, G. Wang and N. M. Mozier. 2012. Residual DNA analysis in biologics development: review of measurement and quantitation technologies and future directions. Biotechnol. Bioeng. 109(2): 307-317.
[https://doi.org/10.1002/bit.23343]

-
Zheng, W., L. Jiang, Q. Lei, J. Yang, X. Gao, W. Wang, Y. Zhang, T. Kong, Q. Chen and G. Li. 2019. Development and Validation of Quantitative Real-Time PCR for the Detection of Residual CHO Host Cell DNA and Optimization of Sample Pretreatment Method in Biopharmaceutical Products. Methodology 21: 17.
[https://doi.org/10.1186/s12575-019-0105-1]

Appendix
Appendix

Cane detection PCR products with residual DNAs from 8 different 100% adulterated honey. 10 Cane-specific detection PCR products were separatedly migrated in 2% Agarose gel, 1×TAE buffer, 50 volt, 60 min long. Left and right DNA size marker shows 100, 200, 300, 400 bp, etc from lowest band. As positive control, 271 bp long Cane-specific Detection PCR products were shown in lane 1 and 2. Also, same sized bands might be observed in lane 3, 5, 7 and 8. Howerer, it is very hard to clear decision based on this kind of Cane detection PCR (1st round PCR). Using Cane-specific nested PCR (2nd round PCR), all 100% adulterated honey samples were determined as clear positive (detection of 137 bp-long cane specific gene).
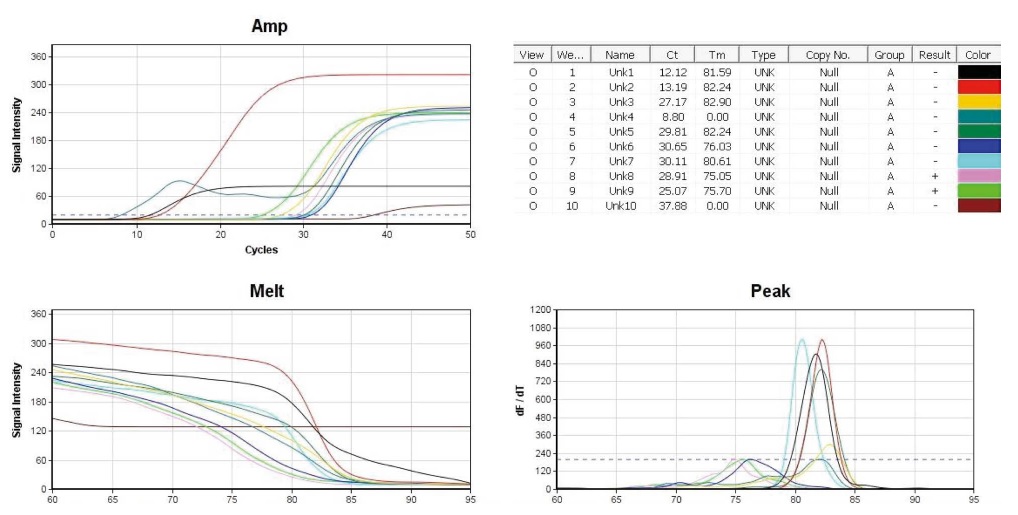
Beet-specific Detection PCRs with residual DNA from 8 commercial honey products. The conditions of temperature/incubation-time in Beet-nested PCR were 95℃ 4 sec, 59℃ 4 sec, 72℃ 4 sec (50 cycles). 10 pmole Beet-RdDP-nF/nR primers were used in each PCR. Lane 1 (positive control), 1 ng standard DNA pTOP-Beet-RdDp315 was used as initial template; lane 2 (positive control), 0.01 ng In pTOP-Beet-RdDp315. lane 3~10, each 1 μL of reisdual DNA from 8 commercial honey. Left-high, amplification curves of 10 PCRs; Righthigh, CT and Tm; Left-low, melting-point analysis; Right-low, peak-curve (dF/dT). Based on Tm value, lane 3, 5 indicate as positive, and lane 7 might be positive (amplification of beet-specific gene).
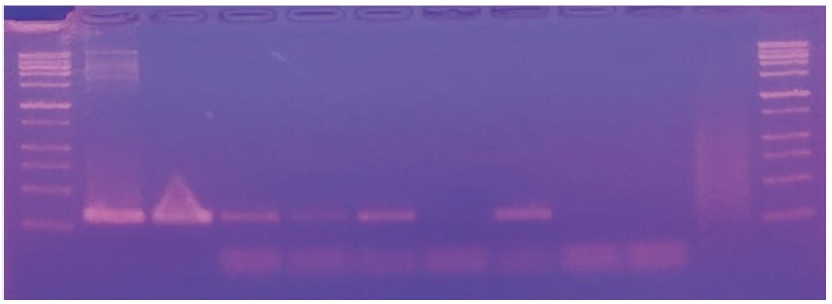
Beet-specific Detection PCR products using 8 commercial honey products. 10 Beet-specific Detection PCR products (Supplement Figure S2) were separatedly migrated in 2% Agarose gel, 1×TAE buffer, 50 volt, 60 min long. Left and right DNA size marker shows 100, 200, 300, 400 bp, etc from lowest band. As positive control, 160 bp long Beet-specific Detection PCR products were shown in lane 1 and 2. Also, same sized bands were observed in lane 3, 4, 5 and 7 (positive).
