
다양한 조건의 살충제에 노출된 꿀벌에서의 qRT-PCR을 위한 안정 발현 reference 유전자 선정
Abstract
Pesticides have been considered to be a major factor that leads to a significant decline in honey bee populations, including colony collapse disorder. To understand pesticide detoxification physiology in honey bees, it is essential to identify the gene expression in different pesticide-exposed conditions. For the accurate determination of target gene quantification through quantitative real-time polymerase chain reaction (qRT-PCR), reference gene stably expressing across different samples should be selected. Therefore, in this study, to select the optimal reference genes, we analyzed the amplification stability of five candidate reference genes (RPS5, RPS18, GAPDH, ARF1, and RAB1A) from honey bees exposed to seven pesticides (acetamiprid, imidacloprid, flupyradifurone, fenitrothion, carbaryl, amitraz, and bifenthrin) across various condition of samples, including different tissues and exposure time and concentration of pesticides using four programs (geNorm, NormFinder, BestKeeper, and RefFinder). Although the stability values of genes varied depending on different analysis algorithms, our results suggest that RPS5 is the most appropriate reference gene for the identification of target gene expression levels in qRT-PCR assays for honeybees under various conditions of pesticide treatment.
Keywords:
Honey bee, Pesticide, Reference gene, qRT-PCR, Expression stability서 론
꿀벌은 꿀, 프로폴리스, 로얄젤리 등의 양봉산물 생산 뿐만 아니라 딸기, 사과, 참외, 고추 등의 대부분의 충매 작물의 화분 매개체로서 중요한 곤충이다 (Jung, 2008; Paudel et al., 2015). 이들의 농업 생태계에 대한 기여는 전 세계 농작물의 70% 이상이 꿀벌의 화분매개에 의존하고 있으며, 미국에서의 꿀벌의 화분매개로의 경제적 가치가 연간 140억 달러에 이르는 것으로 추정하고 있다 (Lee, 2008). 우리나라의 경우, 꿀벌 화분 매개가 50억 달러에 달하는 과채류 생산에 기여하는 것으로 생각된다 (Jung, 2008). 지구 생태계의 10만종 이상의 현화식물이 화분매개에 의존하고 있으며, 꿀벌을 비롯한 화분매개 곤충의 활동으로 식물상의 유지, 야생 동물의 먹이로 과실과 종자를 제공하는 등의 농업생태계 뿐만 아니라 자연 생태계에 기여하는 꿀벌이 비경제적 가치는 산출이 불가할 정도로 중요하다 (Jung, 2008). 꿀벌의 경제적, 공익적 가치에도 불구하고 최근 꿀벌은 그 개체수가 급속하게 감소하는 위기에 처해있다. 2006년 미국을 시작으로 봉군의 30~90%의 개체가 사라지는 봉군붕괴현상 (CCD, Colony Collapse Disorder)이 미국과 유럽 각지에서 보고되고 있으며 (Johnson, 2010), 바이러스와 곰팡이, 응애 발생 및 기후변화를 비롯한 다양한 요인이 CCD의 발생원인으로 생각되고 있으나 (VanEngelsdorp et al., 2007; Bromenshenk et al., 2010), 최근에는 imidacloprid와 clothianidin 등의 neonicotinoid계 살충제가 CCD의 주요한 요인인 것으로 이해되고 있다 (Johnson, 2010; Chen sheng et al., 2014). 아시아 국가에서는 아직 CCD가 보고되지 않았지만, 각국에서 꿀벌의 개체수가 급속히 감소하는 현상이 보고되고 있다 (Zee et al., 2014; Liu et al., 2016). 2022년 초 우리나라 남부지방을 중심으로 대규모의 월동 꿀벌 폐사 현상은 양봉 산업의 위기를 단적으로 보여주는 사례라 판단된다 (RDA, 2022). 우리나라에서의 월동 꿀벌 폐사 현상의 원인으로 급격한 기후/기상 변화, 드론을 이용한 농약 살포, 질병 발생, 불량 꿀벌 사료의 공급으로 인한 영양 불균형, 응애 방제의 실패 등 다양한 요인이 지목되고 있으나, 다양한 요인들이 복합적으로 작용하여 나타나는 현상이라는 것이 대부분 연구자들의 공통된 의견이다 (Choi et al., unpublished data; Lee et al., 2022). 농약이 CCD 현상의 주요한 요인으로 지목된 것을 고려할 때, 우리나라에서 발생하고 있는 봉군 개체수 감소의 원인 중 하나로 농약의 노출을 배재할 수 없다. 특히 외역활동 중 살포된 농업용 살충제에 비의도적으로 꿀벌이 노출될 가능성이 높고, 응애 방제를 위해 봉군에 직접 처리되는 살비제 등이 봉군의 잠재적 위험으로 판단된다 (VanEngelsdorp et al., 2009). 살충제 노출이 꿀벌의 면역 체계, 학습/기억 능력, 외역활동력, 후각인지능력에 영향을 미친다는 기연구결과에 근거할 때 (Desneux et al., 2007; Williamson and Wright, 2013), 살충제가 꿀벌의 다양한 생리학적 경로에 영향을 미침은 자명한 사실이다. 살충제 노출에 의한 꿀벌의 생리학적 변화를 연구하기 위해서는 관련 유전자의 발현양의 변화를 정확히 측정하고 산출해 내야한다.
유전자 발현을 정량화 하기 위해서는 빠른 반응 속도, 높은 감소, 높은 재현성 및 정확도를 담보하는 quantitative real-time polymerase chain reaction (qRT-PCR)을 가장 널리 활용하고 있으나 (Bustin et al., 2005; Zhai et al., 2014), qRT-PCR의 높은 반응 감도로 인해 primer의 PCR 증폭 효율, 주형 핵산의 양, 처리 조건 및 조직 등의 다양한 조건의 샘플들 간 유전자의 발현 정도를 정량하는 과정 중 심각한 오류가 발생할 가능성이 높다 (Ling and Salvaterra, 2011; Zhai et al., 2014; Moon et al., 2018a, b). 그래서 다양한 조건의 샘플에서도 안정적으로 발현되는 reference 유전자의 발현량을 기준으로 대상 유전자의 발현량을 정량화 하는 과정이 필수적으로 수행되어야 한다 (Ling and Salvaterra, 2011). Reference 유전자는 대부분 보편적인 세포 생리학적 기능을 하는 housekeeping gene을 주로 사용하는데, 곤충에서는 ribosomal proteins (RP) (Mamidala et al., 2011; Cardoen et al., 2012; Lu et al., 2012; Kim et al., 2014; Lee and Kim, 2017), glyceraldehyde-3-phosphate dehydrogenase (GAPDH) (Reim et al., 2013), ADP-ribosylation factor (ARF), Ras-related protein Rab (RAB) (Kim et al., 2022a), actin (ACT) (Seong et al., 2012; Zhai et al., 2013), tubulin (TUB) (Swarup and Verheyen, 2011; Zhai et al., 2014), TATA binding protein (TBP) (Zhai et al., 2014), elongation factor 1 beta (EF-1β) (Scharlaken et al., 2008), heat shock protein (HSP) (Ling and Salvaterra, 2011; Xie et al., 2012), arginine kinase (AK) (Horňáková et al., 2010) 등의 유전자를 qRT-PCR의 reference 유전자로 활용해 왔다. 그러나 많은 연구에 의하면 이들 housekeeping 유전자가 샘플의 다양한 조건에 따라 그 발현량이 항상 일정적으로 유지되지 않는 것으로 나타났고 (Lourenço et al., 2008; Scharlaken et al., 2008; Ling and Salvaterra, 2011; Reim et al., 2013; Zhai et al., 2014), 이는 특정 조건의 샘플을 대상으로 유전자 발현량을 측정하기에 앞서 qRT-PCR 실험을 위해 안정적으로 발현되는 reference 유전자를 검증하고 선택하는 과정이 필수적으로 수행되어야 함을 의미한다. 다수의 연구에서 곤충을 대상으로 reference 유전자들의 발현 안정성을 꿀벌 (Apis mellifera) (Lourenço et al., 2008; Scharlaken et al., 2008; Reim et al., 2013; Moon et al., 2018a, b; Jeon et al., 2020; Kim et al., 2021), 노랑초파리 (Drosophila melanogaster) (Kim et al., 2019; Kim et al., 2020), 벗초파리 (Drosophila suzukii) (Zhai et al., 2014), 거짓쌀도둑거저리 (Tribolium castaneaum) (Lord et al., 2010), 빈대 (Cimex lectularius) (Mamidala et al., 2011), Rhodnius prolixus (Majerowicz et al., 2011), 금파리 (Lucilia cuprina) (Bagnall and Kotze, 2010), 그리고 호주솔개메뚜기 (Chortoicetes terminifera) (Chapuis et al., 2011) 등에서 평가했다.
특히 꿀벌의 경우에도 다양한 발달 단계별 (Reim et al., 2013; Kim et al., 2021), 내외역봉간 (Moon et al., 2018b), 세균 접종 (Scharlaken et al., 2008), 연간 월별 (Jeon et al., 2020) , 살충제 처리 (Wieczorek et al., 2020; Kim et al., 2022a) 등의 다양한 환경 조건에서 안정적으로 발현되는 reference 유전자를 선발하는 연구가 수행되어 왔다. 최근 본 연구팀은 7가지 살충제 (acetamiprid, imidacloprid, flupyradifurone, fenitrothion, carbaryl, amitraz, bifenthrin)의 다양한 농도와 다양한 처리 시간 조건에서 RPS5를 적합한 reference 유전자로 제안하였으나 (Kim et al., 2023), 이들 살충제 노출 후 5가지 조직 (head, thorax, gut, fat body, carcass)에서는 RAB1A와 RPS18 두 유전자를 혼합하여 reference로 활용하는 것이 정확한 표적 유전자의 발현량을 산출할 수 있을 것으로 제안하였다 (Kim et al., 2022b). 동일한 살충제를 처리한 꿀벌에서도 노출 조건에 따라 제안되는 안정 발현 유전자가 상이하여 비교 대상 꿀벌의 샘플 조건이 다른 경우 표적 유전자의 발현량을 명확히 정량하기에 한계가 있다고 판단된다. 그러므로 본 연구에서는 위 7가지 살충제에 노출된 다양한 조건 (농도, 노출시간, 조직)의 꿀벌 샘플에서 공통으로 적용이 가능한 qRT-PCR 용 reference 유전자를 선발하였다.
재료 및 방법
1. 실험 곤충 및 살충제 처리
실험 곤충은 경북대학교 실험 양봉장 (36°36ʹ69.09ʺN, 128°11ʹ70.42ʺE)에서 사육하는 양봉꿀벌 (Apis mellifera L.) 성충을 사용하였고, 실험에 사용된 7가지 살충제 (acetamiprid, imidacloprid, flupyradifurone, fenitrothion, carbaryl, amitraz, bifenthrin)는 Sigma-Aldrich (Saint Louis, MO, USA)에서 구매하였다. 다양한 농도의 살충제에 노출된 꿀벌 샘플 확보를 위해 각 살충제의 LD5, 10, 20, 30, 40, 50 (μg/bee) 농도를 sucrose solution (10 μL)과 함께 구강에 제공하여 섭식하도록 하였고 (Kim et al., 2023), 각 꿀벌 샘플은 살충제 노출 24시간 후에 확보하였다 (Kim et al., 2023). 다양한 시간 동안 살충제에 노출된 샘플 확보를 위해 LD20 농도의 살충제 처리 0, 3, 6, 12, 24, 48, 72시간 후에 샘플을 확보하였다 (Kim et al., 2023). 살충제에 노출된 꿀벌의 조직 샘플을 확보하기 위해, LD20 농도의 살충제에 24시간 노출된 생존한 꿀벌을 해부하여 5가지 조직 (head, thorax, gut, fat body, carcass)으로 분리하였다 (Kim et al., 2022b).
2. RNA 추출 및 qRT-PCR
각 살충제의 다양한 농도와 다양한 노출 시간으로 처리된 꿀벌 샘플은 총체 1마리를 1반복군으로 설정하여 총 5반복으로 RNA를 추출하였다 (Kim et al., 2023). 살충제 노출 후 확보한 각 조직 샘플은 5마리를 해부하여 pooling 한 후 1반복군으로 활용하였고, 총 3반복으로 RNA를 추출하였다 (Kim et al., 2022b). 준비된 꿀벌 샘플은 yesRTM total RNA extraction kit (GenesGen, Busan, Korea)의 buffer RXL과 함께 파쇄한 후, total RNA를 추출하였고, RNA 추출 과정 중 genomic DNA는 제거하였다. 1 μg의 total RNA에서 ReverTra AceTM (Toyobo, Osaka, Japan)를 이용하여 cDNA를 합성한 후, CFX Connect Real-Time PCR Detection System (Bio-Rad, Hercules, CA, USA) 장비를 이용하여 SYBR GREEN 방법을 통해 qRT-PCR을 실시하였다 (Kim et al., 2022b; Kim et al., 2023). 본 연구에서 검증하는 유전자는 5개로 다음과 같다 [40S ribosomal protein S5 (RPS5), 40S ribosomal protein S18 (RPS18), Glyceraldehyde-3-phosphate dehydrogenase (GAPDH), ADP-ribosylation factor 1 (ARF1), Ras-related protein Rab1A (RAB1A)]. 이들 유전자 증폭을 위한 primer들은 이전 연구에서 증폭 효율과 증폭 대상 유전자에 대한 특이성을 검증하였다 (Kim et al., 2022b; Kim et al., 2023). qRT-PCR은 Thunderbird SYBR qPCR Master Mix (Toyobo)를 이용하였고, 다양한 조건의 꿀벌 샘플로부터 준비된 cDNA는 10배 희석한 후 동일하게 5 μL씩 분주하여 다음과 같은 조건으로 qRT-PCR 을 실시하였다. 95°C 1분, (95°C-15초; 56°C-15초; 72°C-30초)×40 cycles. 또한 PCR 산물은 melting curve 분석 (65°C~95°C in 0.5°C increments)을 통해 그 특이성을 확인하였다. Quantification cycle (Cq) 값은 모든 샘플에 동일하게 fluorescence threshold (0.1)에서 측정하였다 (Kim et al., 2022b; Kim et al., 2023).
3. reference 유전자의 발현 안정성 평가
7가지 살충제를 다양한 농도로 처리한 꿀벌, 다양한 처리 시간의 샘플 (Kim et al., 2023) 및 살충제 처리 후 5가지 조직을 대상 (Kim et al., 2022b)으로 실시한 5개 유전자의 각각의 Cq 값은 이전 연구에서 획득하였다. 본 연구에서는 각 샘플에 대한 각 유전자의 Cq 값을 통해 후보 reference 유전자의 발현 안정성을 Cq 분포도 (Jeon et al., 2020; Kim et al., 2021; Kim et al., 2022b)와 NormFinder (version 0.953) (Andersen et al., 2004), BestKeeper (version 1) (Pfaffl et al., 2004), geNorm (version 3.2) (Vandesompele et al., 2002), RefFinder (Xie et al., 2012)를 포함하는 4가지 프로그램을 통해서 분석하였다. Cq 분포도 분석을 통해서 arithmetic means (AM), standard deviation (SD)과 coefficient of variation (CV) value를 다음과 같이 산출하였다 (CV=SD/AM) (Fig. 1). NormFinder 프로그램은 각 유전자의 샘플 조건에 따른 전반적인 발현 변동값에 근거하여 stability value 수치를 산출하게 되고, stability value 값이 낮을수록 더 안정적인 유전자를 의미한다 (Andersen et al., 2004) (Fig. 2). BestKeeper 프로그램은 각 유전자 Cq 값의 기하평균 (geometric mean)과 표준편차 값 (SD)에 근거하여 발현 안정도를 평가하게 되고, SD 값이 낮을수록 더 안정적이라고 인정된다 (Pfaffl et al., 2004) (Fig. 3). geNorm 프로그램 또한 기하평균에 근거하여 각 유전자의 expression stability (M) 값을 산출하고 더 낮은 M 값을 가지는 유전자가 안정 발현 유전자로 판단된다 (Fig. 4). RefFinder는 앞선 3가지 프로그램 (geNorm, NormFinder, BestKeeper)의 분석 결과와 delta-Ct 분석 방법을 종합하여 geomean ranking values 값을 계산하여 안정 발현 유전자의 순위를 지정해 준다 (Xie et al., 2012) (Fig. 5).
결과 및 고찰
1. Cq 분포도 분석
7가지 살충제를 처리한 후 5가지 조직을 대상으로 실시한 5가지 유전자의 Cq 값 (Kim et al., 2022b)과, 각 살충제를 다양한 농도로 처리한 꿀벌 및 다양한 노출 시간으로 처리된 꿀벌 샘플에서 획득한 각 유전자의 Cq 값 (Kim et al., 2023)을 통합하여 Cq 분포도 분석을 실시하였다 (Fig. 1). 본 연구에서는 통합된 각 유전자의 Cq 값을 근거로 AM, SD, CV 값을 재계산하였다. 살충제 처리의 다양한 조건에 따른 SD 값은 ARF1이 0.92로 가장 낮은 편차를 보였고, 다음으로 RPS5 (1.06), RAB1A (1.07), RPS18 (1.18)로 산출되었고, GAPDH의 SD 값은 2.46로 가장 높은 편차를 보였다 (Fig. 1A). SD 값에 AM 값을 나눈 CV 값으로 각 유전자의 발현 안정성을 평가한 결과, ARF1이 0.04로 가장 낮은 수치를 획득하였고, RAB1A (0.05), RPS5 (0.06), RPS18 (0.06), GAPDH (0.14) 순으로 산출되었다 (Fig. 1A). CV 값이 1 미만일 때 그 유전자의 발현 변이가 크지 않다고 판단할 때 (Ospina and Marmolejo-Ramos, 2019), 5가지 후보 유전자 모두 qRT-PCR의 reference 유전자로 선정 가능성이 높다고 생각된다. 5가지 유전자의 CV 값에 근거하여 발현 안정성을 비교하면, ARF1이 다양한 살충제 노출 조건에서 가장 안정적으로 발현되는 reference 유전자로 생각되는 반면, GAPDH는 가장 불안정하게 발현되는 것으로 보인다 (Fig. 1A). 살충제 처리 후 다양한 조직을 대상으로 실시한 Cq 분포도 분석에서도 ARF1이 가장 낮은 SD 값과 CV 값을 나타내었고, GAPDH에서 가장 높은 SD 값과 CV 값을 나타냈다 (Fig. 1B) (Kim et al., 2022b). 뿐만 아니라 다양한 농도의 살충제 처리 조건과 다양한 노출 시간 조건에서도 역시 ARF1과 GAPDH가 각각 가장 안정적인 유전자와 가장 불안정적인 유전자로 분석되었다 (Fig. 1C) (Kim et al., 2023). 이를 통해 살충제 처리 조건이 다양한 샘플에서 ARF1의 발현은 5개 유전자 중 가장 낮은 변이를 보이는 것으로 사료된다.
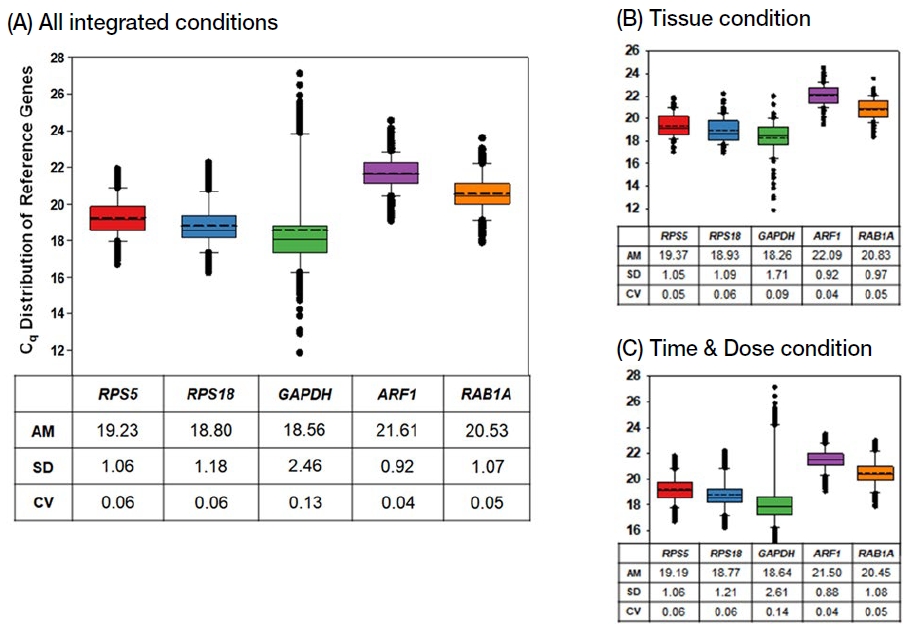
Distribution of quantification cycle (Cq) values of five candidate reference genes in honey bee samples exposed to seven pesticides. The all Cq values were integrated (A) from different tissues (B) and different treatment conditions of pesticides, such as exposure time and concentration of pesticides (C). The horizontal lines in the box indicate the 25th, 50th, and 75th percentile values. The dotted lines in the box show the mean median. The error bars denote the maximum and minimum values. The arithmetic mean (AM), standard deviation (SD), and coefficient of variation (CV) values were calculated (CV=SD/AM). Image of (B) was reproduced with permission from Kim et al. (2022b). Copyright 2022 PLoS ONE. Image of (C) was reproduced with permission from Kim et al. (2023). Copyright 2023 Entomological Research.
2. NormFinder을 통한 유전자 발현 안정성 분석
각 살충제 처리한 꿀벌의 조직 (Kim et al., 2022b)과 다양한 살충제 처리 조건 (농도 및 노출시간) (Kim et al., 2023)에 따른 각 5가지 후보 reference 유전자들의 발현 안정성을 통합하여 NormFinder를 통해 분석하였다 (Fig. 2). NormFinder에서는 stability value 수치를 산출하고 그 값이 0.15 미만일 경우, 유전자의 발현 안정성을 담보한다 (McMillan and Pereg, 2014; Julian et al., 2016). 위 기준으로 볼 때 5가지 유전자들은 qRT-PCR 분석에 안정적인 reference 유전자로 사용 가능하다고 판단된다 (Fig. 2A). 그러나 각 유전자의 stability value을 상호 비교한 결과, RPS5의 stability value가 가장 낮은 값 (0.005)으로 산출되었고, 다음으로 RAB1A (0.006), RPS18 (0.008), ARF1 (0.034), GAPDH (0.087) 순으로 확인되었다 (Fig. 2A). Cq 분포도 분석 (Fig. 1A)에서 가장 불안정한 유전자로 선정되었던 GAPDH가 NormFinder에서도 가장 불안정한 stability value를 나타내었고 (Fig. 2A), 살충제 처리 꿀벌의 조직 샘플 (Fig. 2B) (Kim et al., 2022b)과 살충제처리조건 (농도 및 노출시간)에 따른 꿀벌 샘플 (Fig. 2) (Kim et al., 2023)에서도 가장 불안정한 발현 안정성을 보였다. Cq 분포도 (Fig. 1A)에서 가장 안정적인 유전자로 분석되었던 ARF1은 NormFinder에서는 두번째로 불안정한 유전자로 선정되었고 (Fig. 2A), 조직 샘플 (Fig. 2B)과 농도/시간 샘플 (Fig. 2C)에서도 동일한 결과가 도출되었다. 조직 샘플 (Fig. 2B)과 농도/시간 샘플 (Fig. 2C)에서는 RAB1A가 각각 가장 안정적인 유전자로 분석되었으나, 두 조건을 통합하였을 때 (Fig. 2A)는 RPS5가 가장 안정적인 유전자로, RAB1A가 두 번째로 안정적인 유전자로 선정되었다.
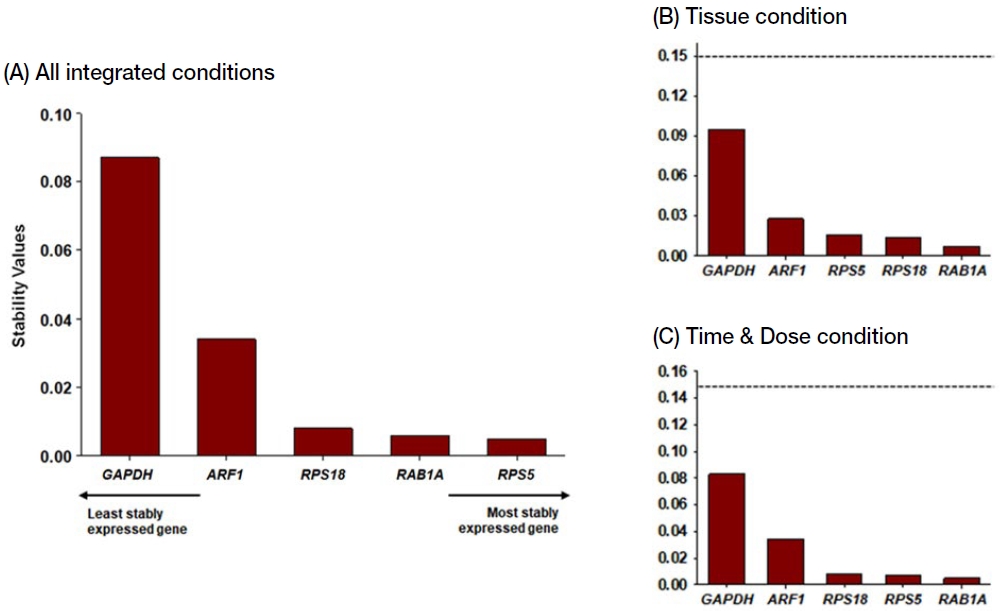
The expression stability values of five candidate reference genes in honey bee following seven pesticides treatment under different treatment conditions, such as different tissues of honey bee treated with pesticides (B), and various treatment time and concentration of pesticides (C), and all integrated conditions (A). The data were analyzed by NormFinder. The dotted lines indicate the cut-off value for the suggested reference genes. Image of (B) was reproduced with permission from Kim et al. (2022b). Copyright 2022 PLoS ONE. Image of (C) was reproduced with permission from Kim et al. (2023). Copyright 2023 Entomological Research.
3. BestKeeper를 통한 유전자 발현 안정성 분석
7가지 살충제의 다양한 처리 조건 (농도 및 노출시간) (Kim et al., 2023)과 살충제 노출 꿀벌의 조직 (Kim et al., 2022b)에 따른 reference 유전자의 발현 안정성을 통합하여 BestKeeper로 분석하였다 (Fig. 3). Cq 분포도 분석 (Fig. 1A)에서와 동일하게 ARF1이 가장 안정적인 유전자로 선정되었고, RAB1A, RPS5, RPS18, GAPDH 순으로 그 안정성이 분석되었다 (Fig. 3A). 살충제 처리의 다양한 조건을 통합한 각 유전자의 안정성 순위는 조직 샘플 (Fig. 3B) (Kim et al., 2022b)과 농도/노출시간 변이 샘플 (Fig. 3C) (Kim et al., 2023)과 동일하게 분석되었다. BestKeeper은 각 유전자 Cq 값의 geometric mean과 SD에 근거하여 발현 안정도를 평가하게 되는데 (Pfaffl et al., 2004), Cq 값의 SD와 CV 값으로 안정성을 평가하는 Cq 분포도 분석법과 유사한 기준으로 유전자 발현 안정도를 평가하기에 두 분석법의 결과가 유사한 것으로 판단된다. 앞선 두 분석법 (Figs. 1, 2)에서와 동일하게 GAPDH는 가장 불안정한 유전자로 BestKeeper에서도 선정되었으며, SD 값이 1.0 미만일 때 안정한 reference 유전자로 제안하는 BestKeeper 기준으로 근거할 때 (Chechi et al., 2012), GAPDH는 살충제 노출 꿀벌의 다양한 샘플을 대상으로 실시하는 qRT-PCR에 적합한 reference 유전자로 제안되지 않는다.

The standard deviation values of five candidate reference genes in honey bee following seven pesticide treatment under different treatment conditions, such as different tissues of honey bee treated with pesticides (B), and various treatment time and concentration of pesticides (C), and all integrated conditions (A). The data were analyzed by BestKeeper. The dotted lines indicate the cut-off value for the suggested reference genes. Image of (B) was reproduced with permission from Kim et al. (2022b). Copyright 2022 PLoS ONE. Image of (C) was reproduced with permission from Kim et al. (2023). Copyright 2023 Entomological Research.
4. geNorm을 통한 유전자 발현 안정성 분석
다양한 살충제 처리조건 (Kim et al., 2022b; Kim et al., 2023)에 대한 각 reference 유전자 발현 안정성을 본 연구에서 통합하여 geNorm으로 분석하였다 (Fig. 4). geNorm에서는 M 값을 산출하고 M 값이 0.5 미만일 때 그 유전자가 샘플들 간 안정적으로 발현된다고 평가받는다 (Hellemans et al., 2007; Liu et al., 2014). M<0.5 기준으로 근거할 때, 본 연구에서는 GAPDH의 M 값이 0.954로 적당한 reference 유전자로 제안되지 않는다 (Fig. 4A). 이는 조직 샘플 (Fig. 4B) (Kim et al., 2022b)과 살충제 농도/노출시간 샘플 (Fig. 4C) (Kim et al., 2023)에서도 동일하게 GAPDH가 M<0.5 기준을 상외하는 결과에서 유래한 것으로 판단된다. GAPDH를 제외하고 나머지 4개의 유전자는 M<0.5 기준에 부합하여 살충제 처리 꿀벌 샘플 간 비교 연구를 위한 안정적인 reference 유전자로 활용 가능할 것으로 사료된다 (Fig. 4A). 그 중 RPS5의 M 값은 0.319로 가장 안정적인 유전자로 제안되었고, RPS18 (0.319), RAB1A (0.357), ARF1 (0.420) 순으로 그 안정성이 검증되었다. geNorm 분석에 따른 안정 유전자의 순위 결과는 NormFinder의 것과 가장 유사하게 나타났다 (Fig. 2A, 4A).
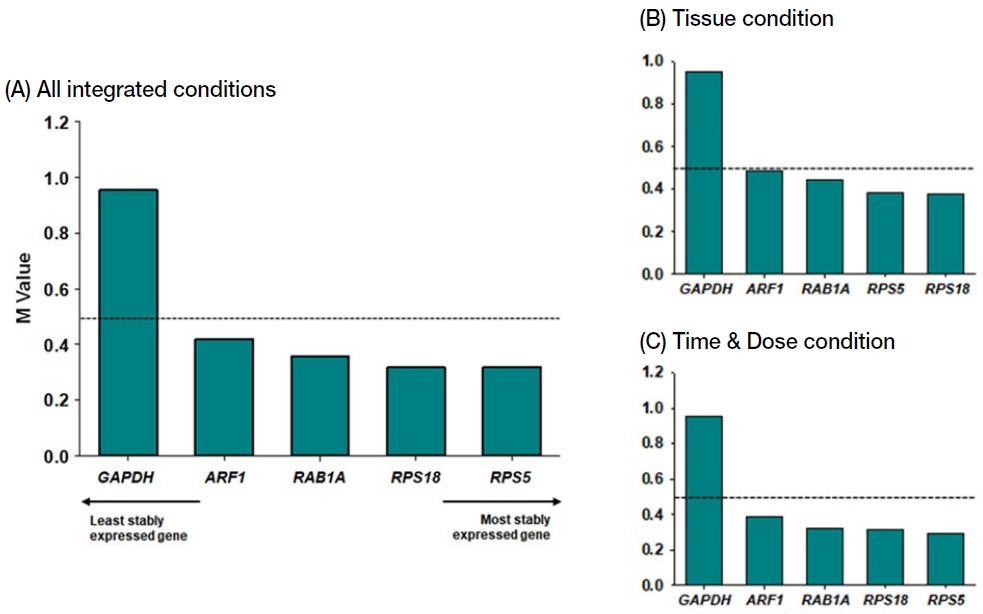
The expression stability values (M) of five candidate reference genes in honey bee following seven pesticide treatment under different treatment conditions, such as different tissues of honey bee treated with pesticides (B), and various treatment time and concentration of pesticides (C), and all integrated conditions (A). The data were analyzed by geNorm. The dotted lines indicate the cut-off value for the suggested reference genes. Image of (B) was reproduced with permission from Kim et al. (2022b). Copyright 2022 PLoS ONE. Image of (C) was reproduced with permission from Kim et al. (2023). Copyright 2023 Entomological Research.
5. RefFinder를 통한 유전자 발현 안정성 분석
Cq 분포도 분석 (Fig. 1), NormFinder (Fig. 2), BestKeeper (Fig. 3), geNorm (Fig. 4)로 분석한 각 유전자의 발현 안정도가 분석법에 따라 조금 상이함에 따라, RefFinder에서는 이들 분석 결과와 delta-Ct 분석 방법을 종합하여 geomean ranking values 값을 계산하여 안정 발현 유전자의 순위를 지정하게 된다 (Xie et al., 2012). 살충제 처리 조건 (농도/노출시간) (Kim et al., 2023)와 살충제 처리 꿀벌의 조직 (Kim et al., 2022b)의 데이터 값을 종합하여 RefFinder로 분석한 결과, RPS5가 가장 안정적인 유전자로 선정되었고, 이후 RPS18, RAB1A, ARF1, GAPDH 순으로 순위가 결정되었다 (Fig. 5A). 조직 샘플과 다양한 살충체 처리 조건의 샘플에서는 RAB1A가 가장 안정 유전자로 선정되었지만 (Fig. 5B, C) (Kim et al., 2022b; Kim et al., 2023), 종합적 분석에서는 RPS5가 가장 안정 유전자로, RAB1A는 세번째로 안정적인 유전자로 분석되었다 (Fig. 5A). RefFinder로 분석한 결과의 안정 유전자 순위는 geNorm의 것 (Fig. 4A)와 동일하게 분석되었고, NormFinder (Fig. 2A)와는 첫 번째, 네 번째, 다섯 번째 안정 발현 유전자가 동일하게 분석되었다.
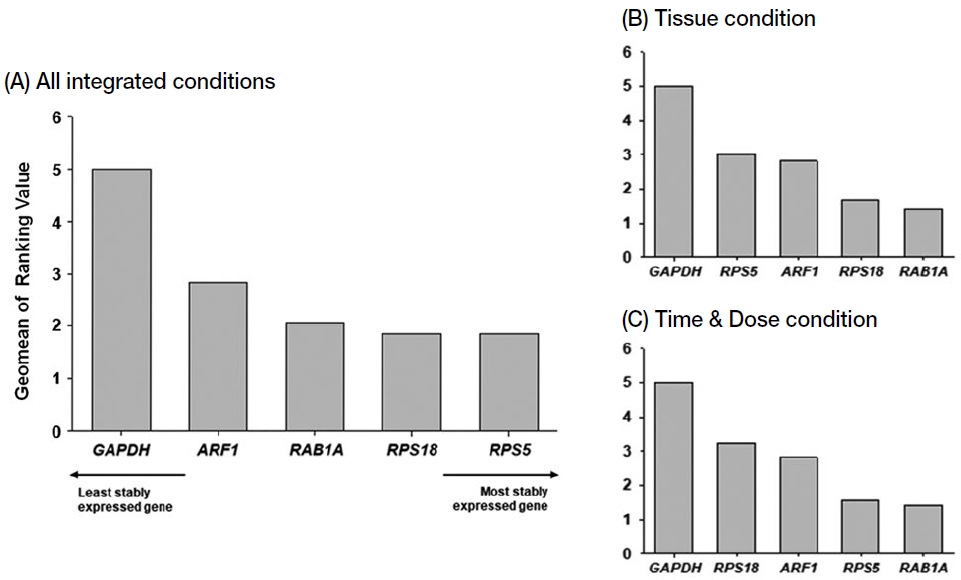
The geomean of the ranking values of five candidate reference genes in honey bee following seven pesticide treatment under different treatment conditions, such as different tissues of honey bee treated with pesticides (B), and various treatment time and concentration of pesticides (C), and all integrated conditions (A). The data were analyzed by RefFinder. Image of (B) was reproduced with permission from Kim et al. (2022b). Copyright 2022 PLoS ONE. Image of (C) was reproduced with permission from Kim et al. (2023). Copyright 2023 Entomological Research.
6. 후보 reference 유전자 발현 안정성의 종합적 분석
최근 본 연구팀은 7가지 살충제 (acetamiprid, imidacloprid, flupyradifurone, fenitrothion, carbaryl, amitraz, bifenthrin)를 다양한 농도와 시간으로 처리한 꿀벌 샘플과 이들 살충제를 처리한 꿀벌의 5가지 조직 (head, thorax, gut, fat body, carcass)에서의 안정 발현 reference 유전자를 평가한 바가 있다 (Kim et al., 2022b; Kim et al., 2023). 본 연구에서는 앞선 연구를 종합하여 5가지 후보 reference 유전자 (RPS5, RPS18, GAPDH, ARF1, RAB1A)의 발현 양상을 Cq 분포도 분석 (Fig. 1) 및 4가지 통계분석 알고리즘 (NormFinder, BestKeeper, geNorm, RefFinder)을 이용하여 조사하였다 (Figs. 2~5). 이전 연구에서와 동일하게, 본 연구에서도 분석 방법에 따라 유전자의 안정성 순위에는 조금씩 차이가 있었다 (Moon et al., 2018b; Wang et al., 2019; Jeon et al., 2020; Kim et al., 2020; Köhsler et al., 2020; Kim et al., 2022a). 그러나 공통적으로 모든 분석법에서 GAPDH의 순위는 최하위였고, 유전자 발현 안정성 기준값을 제시하는 BestKeeper (Fig. 3A)와 geNorm (Fig. 4A) 분석에서는 GAPDH가 그 기준 값을 상회하는 결과 값이 도출되었다. NormFinder에서는 그 기준 값을 초과하지 않았지만, GAPDH가 여전히 최하위의 순위를 기록하였다 (Fig. 2A). 각 분석법을 통합하여 분석한 RefFinder (Xie et al., 2012)에서도 GAPDH는 최하위의 유전자로 분석된 것을 종합할 때, 살충제에 노출된 꿀벌을 대상으로 표적 유전자의 발현량을 qRT-PCR로 분석할 경우, GAPDH는 최적의 reference 유전자로 활용될 수 없는 것으로 생각된다. GAPDH는 해당과정에서 중요한 가능을 하는 효소이며 (Colell et al., 2009), 최근 연구에서 해독과정에 관여한다는 보고 (Chen et al., 2016)는 살충제에 노출된 꿀벌이 살충제 해독 과정에서 에너지 요구량의 변화가 발생하고 이에 따라 GAPDH의 발현양의 변화가 발생하는 것으로 판단된다. GAPDH를 제외한 나머지 4개 유전자들은 mean stability<0.15 (NormFinder) (McMillan and Pereg, 2014; Julian et al., 2016), SD value<1.0 (Best-Keeper) (Chechi et al., 2012), M<0.5 (geNorm) (Hellemans et al., 2007; Liu et al., 2014) 기준에 근거할 때, 그 발현이 안정적이고 살충제 노출 꿀벌을 대상으로 실시하는 연구에 활용 가능한 reference 유전자로 선정 가능하다고 판단된다. 그러나 분석 방법에 따라 각 유전자의 안정성 순위에서 조금의 차이가 있으나, RefFinder를 통한 종합적 분석에서는 RPS5가 가장 높은 순위를 기록하였다 (Fig. 5A). RPS5는 NormFinder (Fig. 2A), geNorm (Fig. 4A)에서도 가장 높은 순위를 기록하였고, BestKeeper (Fig. 3A)와 Cq 분포도 분석 (Fig. 1A)에서도 안정 발현 유전자 기준 수치 값보다 낮은 안정성을 보였다. 이에 본 분석 결과를 종합할 때, 다양한 살충제에 노출된 꿀벌을 대상으로 연구하고자 하는 유전자의 발현량을 qRT-PCR로 분석하고자 할 때, RPS5가 가장 적합한 reference 유전자로 활용할 수 있다고 제안한다.
적 요
살충제는 봉군붕괴현상을 포함하여 꿀벌 개체수를 크게 감소시키는 주요 요인으로 간주된다. 꿀벌의 살충제 해독 생리를 이해하기 위해서는 다양한 살충제 노출 조건에서 유전자의 발현 변화를 확인하는 것이 필수적이다. Quantitative real-time polymerase chain reaction (qRT-PCR)을 통한 표적 유전자 발현량을 정량하기 위해서는 다양한 조건에서 안정적으로 발현하는 reference 유전자를 선택해야 한다. 따라서 본 연구에서 기존 연구를 기반으로 최적의 reference 유전자를 선정하기 위해 7종의 살충제 (acetamiprid, acetamiprid, imidacloprid, flupyradifurone, fenitrothion, carbaryl, amitraz, bifenthrin)에 노출된 꿀벌의 다양한 조직, 살충제 처리 시간 및 농도 조건의 꿀벌 시료를 대상으로 후보 reference 유전자의 발현 안정성을 4가지 프로그램 (geNorm, NormFinder, BestKeeper 및 RefFinder)을 사용하여 분석하였다. 비록 분석 알고리즘마다 각 유전자의 발현 안정성 수치 값의 차이가 있지만, 본 연구에서 실시한 종합적 분석 결과, RPS5가 다양한 조건으로 살충제에 노출된 꿀벌에 대한 qRT-PCR 분석에서 표적 유전자 발현 수준을 정량화 하는데 가장 적합한 기준 유전자임을 확인하였다.
Acknowledgments
본 연구는 농촌진흥청 공동연구과제 (과제번호: J015778012023)의 지원에 의해 이루어진 것임.
References
-
Andersen, C. L., J. L. Jensen and T. F. Ørntoft. 2004. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64: 5245-5250.
[https://doi.org/10.1158/0008-5472.CAN-04-0496]

-
Bagnall, N. and A. Kotze. 2010. Evaluation of reference genes for real-time PCR quantification of gene expression in the Australian sheep blowfly, Lucilia cuprina. Med. Vet. Entomol. 24: 176-181.
[https://doi.org/10.1111/j.1365-2915.2010.00866.x]

-
Bromenshenk, J. J., C. B. Henderson, C. H. Wick, M. F. Stanford, A. W. Zulich, R. E. Jabbour, S. V. Deshpande, P. E. McCubbin, R. A. Seccomb and P. M. Welch. 2010. Iridovirus and microsporidian linked to honey bee colony decline. PLoS One 5: e13181.
[https://doi.org/10.1371/journal.pone.0013181]

-
Bustin, S., V. Benes, T. Nolan and M. Pfaffl. 2005. Quantitative real-time RT-PCR-a perspective. J. Mol. Endocrinol. 34: 597-601.
[https://doi.org/10.1677/jme.1.01755]

-
Cardoen, D., U. R. Ernst, B. Boerjan, A. Bogaerts, E. Formesyn, D. C. de Graaf, T. Wenseleers, L. Schoofs and P. Verleyen. 2012. Worker honeybee sterility: a proteomic analysis of suppressed ovary activation. J. Proteome Res. 11: 2838-2850.
[https://doi.org/10.1021/pr201222s]

-
Chapuis, M.-P., D. Tohidi-Esfahani, T. Dodgson, L. Blondin, F. Ponton, D. Cullen, S. J. Simpson and G. A. Sword. 2011. Assessment and validation of a suite of reverse transcription-quantitative PCR reference genes for analyses of density-dependent behavioural plasticity in the Australian plague locust. BMC Mol. Biol. 12: 7.
[https://doi.org/10.1186/1471-2199-12-7]

-
Chechi, K., Y. Gelinas, P. Mathieu, Y. Deshaies and D. Richard. 2012. Validation of reference genes for the relative quantification of gene expression in human epicardial adipose tissue. PLoS One 7: e32265.
[https://doi.org/10.1371/journal.pone.0032265]

-
Chen, J., M. Yoshinaga, L. D. Garbinski and B. P. Rosen. 2016. Synergistic interaction of glyceraldehydes-3-phosphate dehydrogenase and ArsJ, a novel organoarsenical efflux permease, confers arsenate resistance. Mol. Microbiol. 100: 945-953.
[https://doi.org/10.1111/mmi.13371]

- Chensheng, L., K. M. Warchol and R. Callahan. 2014. Sub-lethal exposure to neonicotinoids impaired honey bees winterization before proceeding to colony collapse disorder. Bull. Insectology 67: 125-130.
-
Colell, A., D. Green and J. Ricci. 2009. Novel roles for GAPDH in cell death and carcinogenesis. Cell Death Differ. 16: 1573-1581.
[https://doi.org/10.1038/cdd.2009.137]

-
Desneux, N., A. Decourtye and J.-M. Delpuech. 2007. The sublethal effects of pesticides on beneficial arthropods. Annu. Rev. Entomol. 52: 81-106.
[https://doi.org/10.1146/annurev.ento.52.110405.091440]

-
Hellemans, J., G. Mortier, A. De Paepe, F. Speleman and J. Vandesompele. 2007. qBase relative quantification framework and software for management and automated analysis of real-time quantitative PCR data. Genome Biol. 8: R19.
[https://doi.org/10.1186/gb-2007-8-2-r19]

-
Horňáková, D., P. Matoušková, J. Kindl, I. Valterová and I. Pichová. 2010. Selection of reference genes for real-time polymerase chain reaction analysis in tissues from Bombus terrestris and Bombus lucorum of different ages. Anal. Biochem. 397: 118-120.
[https://doi.org/10.1016/j.ab.2009.09.019]

-
Jeon, J. H., K. Moon, Y. Kim and Y. H. Kim. 2020. Reference gene selection for qRT-PCR analysis of season-and tissue-specific gene expression profiles in the honey bee Apis mellifera. Sci. Rep. 10: 13935.
[https://doi.org/10.1038/s41598-020-70965-4]

- Johnson, R. 2010 Honey bee colony collapse disorder. 1-20. Congressional Research Service, Washington.
-
Julian, G. S., R. W. d. Oliveira, S. Tufik and J. R. Chagas. 2016. Analysis of the stability of housekeeping gene expression in the left cardiac ventricle of rats submitted to chronic intermittent hypoxia. J. Bras. Pneumol. 42: 211-214.
[https://doi.org/10.1590/S1806-37562015000000133]

- Jung, C. 2008. Economic value of honeybee pollination on major fruit and vegetable crops in Korea. J. Apic. 23: 147-152.
-
Kim, S., S. Cho and S. H. Lee. 2022a. Selection of stable reference genes for real-time quantitative PCR in honey bee pesticide toxicity studies. J. Apic. Res. 61: 26-36.
[https://doi.org/10.1080/00218839.2021.1950972]

-
Kim, Y., J. Cha, H. Kim, S. H. Lee and Y. H. Kim. 2023. Validation of reference genes for quantitative real-time polymerase chain reaction assay of honeybee under various pesticide treatment conditions. Entomol. Res. 53: 82-92.
[https://doi.org/10.1111/1748-5967.12634]

-
Kim, Y., H. Kim, J. Cha, S. H. Lee and Y. H. Kim. 2022b. Validation of quantitative real-time PCR reference genes and spatial expression profiles of detoxication-related genes under pesticide induction in honey bee, Apis mellifera. PLoS One 17: e0277455.
[https://doi.org/10.1371/journal.pone.0277455]

-
Kim, Y., H. Kim and Y. H. Kim. 2021. Evaluation of quantitative real-time PCR reference genes for the investigation of gene expression profiles in honeybee developmental stages. J. Apic. 36: 251-259.
[https://doi.org/10.17519/apiculture.2021.11.36.4.251]

-
Kim, Y., Y. Kim, D. Kim, S. Kim, G. Seo, S. Shin, J. Lee and Y. H. Kim. 2019. Validation of reference genes for quantitative real-time polymerase chain reaction in Drosophila melanogaster exposed to two chemicals. Entomol. Res. 49: 277-283.
[https://doi.org/10.1111/1748-5967.12365]

-
Kim, Y., Y. Kim and Y. H. Kim. 2020. Evaluation of reference genes for gene expression studies using quantitative real-time PCR in Drosophila melanogaster after chemical exposures. J. Asia-Pac. Entomol. 23: 385-394.
[https://doi.org/10.1016/j.aspen.2020.01.008]

-
Kim, Y. H., D. H. Kwon, H. M. Ahn, Y. H. Koh and S. H. Lee. 2014. Induction of soluble AChE expression via alternative splicing by chemical stress in Drosophila melanogaster. Insect Biochem. Mol. Biol. 48: 75-82.
[https://doi.org/10.1016/j.ibmb.2014.03.001]

-
Köhsler, M., D. Leitsch, N. Müller and J. Walochnik. 2020. Validation of reference genes for the normalization of RT-qPCR gene expression in Acanthamoeba spp. Sci. Rep. 10: 10362.
[https://doi.org/10.1038/s41598-020-67035-0]

- Lee, S.-H. 2008 꿀벌의 실종. The Korea Beekeeping Bulletin, Korea. 329: 28-29.
-
Lee, S.-J., S.-H. Kim, J. Lee, J.-H. Kang, S.-M. Lee, H. J. Park, J. Nam and C. Jung. 2022. Impact of ambient temperature variability on the overwintering failure of honeybees in South Korea. J. Apic. 37: 331-347.
[https://doi.org/10.17519/apiculture.2022.09.37.3.331]

-
Lee, S. H. and Y. H. Kim. 2017. Comparative proteome analysis of honey bee workers between overwintering and brood-rearing seasons. J. Asia-Pac. Entomol. 20: 984-995.
[https://doi.org/10.1016/j.aspen.2017.07.011]

-
Ling, D. and P. M. Salvaterra. 2011. Robust RT-qPCR data normalization: validation and selection of internal reference genes during post-experimental data analysis. PLoS One 6: e17762.
[https://doi.org/10.1371/journal.pone.0017762]

-
Liu, J., Q. Wang, M. Sun, L. Zhu, M. Yang and Y. Zhao. 2014. Selection of reference genes for quantitative real-time PCR normalization in Panax ginseng at different stages of growth and in different organs. PLoS One 9: e112177.
[https://doi.org/10.1371/journal.pone.0112177]

-
Liu, Z., C. Chen, Q. Niu, W. Qi, C. Yuan, S. Su, S. Liu, Y. Zhang, X. Zhang and T. Ji. 2016. Survey results of honey bee (Apis mellifera) colony losses in China (2010-2013). J. Apic. Res. 55: 29-37.
[https://doi.org/10.1080/00218839.2016.1193375]

-
Lord, J. C., K. Hartzer, M. Toutges and B. Oppert. 2010. Evaluation of quantitative PCR reference genes for gene expression studies in Tribolium castaneum after fungal challenge. J. Microbiol. Methods 80: 219-221.
[https://doi.org/10.1016/j.mimet.2009.12.007]

-
Lourenço, A. P., A. Mackert, A. dos Santos Cristino and Z. L. P. Simões. 2008. Validation of reference genes for gene expression studies in the honey bee, Apis mellifera, by quantitative real-time RT-PCR. Apidologie 39: 372-385.
[https://doi.org/10.1051/apido:2008015]

-
Lu, Y., Y.-P. Pang, Y. Park, X. Gao, J. Yao, X. Zhang and K. Y. Zhu. 2012. Genome organization, phylogenies, expression patterns, and three-dimensional protein models of two acetylcholinesterase genes from the red flour beetle. PLoS One 7: e32288.
[https://doi.org/10.1371/journal.pone.0032288]

-
Majerowicz, D., M. Alves-Bezerra, R. Logullo, A. Fonseca-de-Souza, J. Meyer-Fernandes, G. Braz and K. Gondim. 2011. Looking for reference genes for real-time quantitative PCR experiments in Rhodnius prolixus (Hemiptera: Reduviidae). Insect Mol. Biol. 20: 713-722.
[https://doi.org/10.1111/j.1365-2583.2011.01101.x]

-
Mamidala, P., S. P. Rajarapu, S. C. Jones and O. Mittapalli. 2011. Identification and validation of reference genes for quantitative real-time polymerase chain reaction in Cimex lectularius. J. Med. Entomol. 48: 947-951.
[https://doi.org/10.1603/ME10262]

-
McMillan, M. and L. Pereg. 2014. Evaluation of reference genes for gene expression analysis using quantitative RT-PCR in Azospirillum brasilense. PLoS One 9: e98162.
[https://doi.org/10.1371/journal.pone.0098162]

-
Moon, K., S. H. Lee and Y. H. Kim. 2018a. Evaluation of reference genes for quantitative real-time PCR to investigate seasonal and labor-specific expression profiles of the honey bee abdomen. J. Asia-Pac. Entomol. 21: 1350-1358.
[https://doi.org/10.1016/j.aspen.2018.10.014]

-
Moon, K., S. H. Lee and Y. H. Kim. 2018b. Validation of quantitative real-time PCR reference genes for the determination of seasonal and labor-specific gene expression profiles in the head of Western honey bee, Apis mellifera. PLoS One 13: e0200369.
[https://doi.org/10.1371/journal.pone.0200369]

-
Ospina, R. and F. Marmolejo-Ramos. 2019. Performance of some estimators of relative variability. Front. Appl. Math. 5: 43.
[https://doi.org/10.3389/fams.2019.00043]

-
Paudel, Y. P., R. Mackereth, R. Hanley and W. Qin. 2015. Honey bees (Apis mellifera L.) and pollination issues: Current status, impacts, and potential drivers of decline. J. Agric. Sci. 7: 93.
[https://doi.org/10.5539/jas.v7n6p93]

-
Pfaffl, M. W., A. Tichopad, C. Prgomet and T. P. Neuvians. 2004. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations. Biotechnol. Lett. 26: 509-515.
[https://doi.org/10.1023/B:BILE.0000019559.84305.47]

- RDA. 2022. 전국 양봉농가 월동 꿀벌 피해 민관 합동 조사 결과. https://www.rda.go.kr/board/board.do?boardId=farmprmninfo&prgId=day_farmprmninfoEntry&currPage=1&dataNo=100000777725&mode=updateCnt&searchSDate=&searchEDate=&totalSearchYn=Y, Accessed 8 Sep. 2022. Accessed
-
Reim, T., M. Thamm, D. Rolke, W. Blenau and R. Scheiner. 2013. Suitability of three common reference genes for quantitative real-time PCR in honey bees. Apidologie 44: 342-350.
[https://doi.org/10.1007/s13592-012-0184-3]

-
Scharlaken, B., D. C. de Graaf, K. Goossens, M. Brunain, L. J. Peelman and F. J. Jacobs. 2008. Reference gene selection for insect expression studies using quantitative real-time PCR: The head of the honeybee, Apis mellifera, after a bacterial challenge. Insect Sci. 8: 33.
[https://doi.org/10.1673/031.008.3301]

-
Seong, K. M., Y. H. Kim, D. H. Kwon and S. H. Lee. 2012. Identification and characterization of three cholinesterases from the common bed bug, Cimex lectularius. Insect Mol. Biol. 21: 149-159.
[https://doi.org/10.1111/j.1365-2583.2011.01118.x]

-
Swarup, S. and E. M. Verheyen. 2011. Drosophila homeodomain-interacting protein kinase inhibits the Skp1-Cul1-F-box E3 ligase complex to dually promote Wingless and Hedgehog signaling. Proc. Natl. Acad. Sci. 108: 9887-9892.
[https://doi.org/10.1073/pnas.1017548108]

-
Vandesompele, J., K. De Preter, F. Pattyn, B. Poppe, N. Van Roy, A. De Paepe and F. Speleman. 2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 3: research0034.0031.
[https://doi.org/10.1186/gb-2002-3-7-research0034]

-
VanEngelsdorp, D., J. D. Evans, C. Saegerman, C. Mullin, E. Haubruge, B. K. Nguyen, M. Frazier, J. Frazier, D. Cox-Foster and Y. Chen. 2009. Colony collapse disorder: a descriptive study. PLoS One 4: e6481.
[https://doi.org/10.1371/journal.pone.0006481]

- VanEngelsdorp, D., D. C. Foster, M. Frazier, N. Ostiguy and J. Hayes 2007 “Fall-dwindle Disease”: Investigations Into the Causes of Sudden and Alarming Colony Losses Experience by Beekeepers in the Fall of 2006. Florida Department of Agriculture, Harrisburg
-
Wang, Z., Q. Meng, X. Zhu, S. Sun, S. Gao, Y. Gou and A. Liu. 2019. Evaluation and validation of reference genes for quantitative real-time PCR in Helopeltis theivora Waterhouse (Hemiptera: Miridae). Sci. Rep. 9: 13291.
[https://doi.org/10.1038/s41598-019-49479-1]

-
Wieczorek, P., P. Frçckowiak and A. Obrępalska-Stęplowska. 2020. Evaluation of the expression stability of reference genes in Apis mellifera under pyrethroid treatment. Sci. Rep. 10: 16140.
[https://doi.org/10.1038/s41598-020-73125-w]

-
Williamson, S. M. and G. A. Wright. 2013. Exposure to multiple cholinergic pesticides impairs olfactory learning and memory in honeybees. J. Exp. Biol. 216: 1799-1807.
[https://doi.org/10.1242/jeb.083931]

-
Xie, F., P. Xiao, D. Chen, L. Xu and B. Zhang. 2012. miRDeepFinder: a miRNA analysis tool for deep sequencing of plant small RNAs. Plant Mol. Biol. 80: 75-84.
[https://doi.org/10.1007/s11103-012-9885-2]

-
Zee, R. v. d., R. Brodschneider, V. Brusbardis, J.-D. Charriere, R. Chlebo, M. F. Coffey, B. Dahle, M. M. Drazic, L. Kauko and J. Kretavicius. 2014. Results of international standardised beekeeper surveys of colony losses for winter 2012-2013: analysis of winter loss rates and mixed effects modelling of risk factors for winter loss. J. Apic. Res. 53: 19-34.
[https://doi.org/10.3896/IBRA.1.53.1.02]

-
Zhai, Y., Q. Lin, X. Zhou, X. Zhang, T. Liu and Y. Yu. 2014. Identification and validation of reference genes for quantitative real-time PCR in Drosophila suzukii (Diptera: Drosophilidae). PLoS One 9: e106800.
[https://doi.org/10.1371/journal.pone.0106800]

-
Zhai, Y., J. Zhang, Z. Sun, X. Dong, Y. He, K. Kang, Z. Liu and W. Zhang. 2013. Proteomic and transcriptomic analyses of fecundity in the brown planthopper Nilaparvata lugens (Stal). J. Proteome Res. 12: 5199-5212.
[https://doi.org/10.1021/pr400561c]
