꿀벌 병원성 바이러스 Deformed Wing Virus의 4종 특이 단백질들의 대량 생산
Abstract
Deformed Wing Virus (DWV) is one of viral pathogens in honeybee and caused a wing deformity and a premature death of honeybee. In this study, capsid protein VP1, capsid protein VP1-VP3 complex, RNA dependent RNA Polymerase (RdRP) and C3G peptidase (C3G) originated from DWV were successfully over-expressed using E. coli expression system, respectively. Each recombinant proteins using pET32a(+) or pMAL-C2 system was purified using affinity-column chromatography under optimum expression conditions, Four specific proteins in this study would be use as antigens to generate specific antibodies against DWV. Especially, recombinant RdRP and recombinant C3G might be useful materials to studies on enzymatic analysis and inhibitory assay of DWV.
Keywords:
Deformed wing virus, DWV, RdRP, C3G Peptidase, VP1, VP3서 론
Deformed Wing Virus(DWV)는 꿀벌(Apis mellifera L.)의 다양한 병원성 바이러스 중 꿀벌의 날개를 변형시킨다는, 형태적으로 분명한 병징을 보이며, 꿀벌 응애(Varroa destructor)의 매개에 의하여 감염이 증폭되는 병원체로 알려져 있다(Anderson, D.L. and J.W.H. Trueman, 2002; Bailey, L. and B.V. Ball, 1991; Ball, B.V. and M.F. Allen, 1989; Ball et al., 1988).
DWV를 포함하여, 알려진 꿀벌의 모든 병원성 바이러스들은, 양성의 단일가닥 RNA(positive single strand RNA)를 유전체로 가지고 있으며, 다른 Retrovirus들의 경우와 달리, 꿀벌의 유전체(DNA)에 삽입되어 DNA로 변형되지 아니하고("no DNA-stage"), 꿀벌에 감염 이후, 스스로 바이러스의 복제형인 음성 단일가닥 RNA (negative single strand RNA)를 만들고, 이를 주형으로 대량의 바이러스 유전체(양성 단일가닥 RNA)를 생산하는 것으로 추측되고 있다.
RNA의존 RNA 중합효소(RdRP; RNA dependent RNA Polymerase)는 많은 RNA 바이러스 중 "no DNA-Journal stage"의 바이러스에서만 발견되는 효소로써, DWV에서는 양성가닥 RNA로부터 음성가닥 RNA를, 그리고 음성가닥 RNA로부터 다시 양성가닥 RNA(유전체)를 생산하는 기능을 담당한다. 그러나 DNA의존 DNA 중합효소(복제), DNA의존 RNA 중합효소(전사), 그리고 RNA의존 DNA중합효소(역전사효소)의 경우와 달리, RdRP의 기능에 대한 분자적 기작은 어느 하나 분명한 것이 밝혀지지 못하고 있다.
한편, C3G Peptidase는 바이러스의 양성가닥 RNA 유전체가, 꿀벌의 mRNA처럼 기능하여 만들어 낸 거대 polypeptide의 특정 아미노산 서열 부분들을 인식하고, 절단하는 효소이다. 이후 절단된 작은 polypeptide들은 각기 바이러스의 구조 단백질(VP1, VP3 등), 비구조 단백질(Non-structural proteins; RdRP, C3G)의 개별 단백질로 기능하게 되나, 이 C3G의 특이 인식/절단 부위는 현재 여러 가설만 제기된 수준이며, 거대 polypeptide가 최소 6개 이상의 개별 polypeptide로 각기 절단되어 각기 특이 단백질로 기능하게 하는데 그 절단부위의 보존 서열 등 많은 의문점들이 아직 미해결로 남아 있다.
현재까지 DWV에 대항하기 위한 연구들은 주로 DWV 특이 유전자 검출과 검량에 집중되어 왔으며, Ultra-rapid Real-time PCR(Lim et al., 2013), Ultra-Fast High-Performance PCR(Lim, 2013), Real Time PCR(Lee et al., 2005), Loop-mediated Isothermal Amplification(LAMP; No et al., 2010) 등이 그것들이라 할 것이다. 이들 방법들은 모두 유전자 기반의 분자진단법으로 실험실 환경에서 매우 민감한 결과를 보이고 있으나, 양봉 현장에서 즉석 진단에 사용하기에는 어려움이 많은 수준이다. 현재의 기술수준에서 현장진단에 가장 근접한 방법은 Immunochromatography법이라 생각되며, 이를 위한 개별 바이러스에 대한 특이 항원/항체의 개발은 또한 시급한 요구라 판단된다.
따라서 본 연구에서는 대장균 발현시스템을 이용하여DWV의 특이 단백질들 가운데 구조 단백질 2종(VP1과 VP3)과 비구조 단백질 2종(RdRP와 C3G)의 대량생산을 목표로 하였다. 이들 4종의 특이 단백질들은 각기 특이 항체 생산을 위한 항원들로 사용될 수 있을 뿐 아니라, DWV의 생존 및 감염기작을 밝힐 수 있는 연구재료로써 중요하다. 또한 대상으로 한 2종의 효소들은 효소기능 및 그 저해에 대한 연구를 통하여, 꿀벌에 대한 DWV 감염을 저해시킬 수 있는 항바이러스 저해제 개발에서 주요 역할을 할 것으로 기대하였다.
재료 및 방법
시료의 확보 및 RNA의 순수분리
DWV에 감염되었다고 의심되는 꿀벌(Apis mellifera L.) 시료를 경기대학교 양봉장에서 확보를 하였으며, DWV 감염사실은 추후 특이 RT-PCR의 방법으로 확인하였다. RNA 순수분리는 꿀벌 시료 3수에 Total RNA Extraction kit(Intron Bio Inc., Korea)의 1ml lysis buffer와 200μl Chloroform (Daejung Inc., Korea)를 첨가 후, MagNA Lyser Green Beads (Roche, Switzerland)를 사용하여 6,000RPM에서 60초의 조건으로 파쇄를 수행함으로 시작하였다. 이 후 과정은 Total RNA Extraction kit(Intron bio, Korea)를 사용하여 제작자의 지시에 따라 수행하였고, 추출된 전체 RNA는 OD 260nm에서 농도를 측정하고, Reverse Transcription 과정에 사용하였으며, 잔여RNA는 초저온냉동고( 70°C 이하)에 보관하였다.
Reverse Transcription
추출된 전체 RNA중 3,000ng을 사용하여 cDNA를 합성하였다. 3,000ng의 전체 RNA가 포함된 용액에 100pmol oligo-dT를 첨가하여 65°C에서 10분간 정치하였다. 정치 후, 즉시 ice로 옮겨 5분간 정치하였고, 이 반응액에 100mM DDT, 10mM dNTP(각 2.5mM), RNase inhibitor, Rocket Script Reverse Transcriptase(10,000U), 5X reaction buffer를 첨가하여 42°C에서 60분간 정치하여 역전사반응을 수행하였다.
Molecular cloning of each protein gene from DWV
GenBank에 등록된 DWV의 유전체 서열(Accession No. JX878305.1)을 바탕으로 각 단백질 부분의 유전자가 증폭되도록 특이 프라이머를 설계하였고, BLAST(http://www.ncbi.nlm.nih.gov/BLAST)를 통하여 특이성 및 다른 염기서열과의 상동성을 비교하였다. 각 oligonucleotide의 제작은 바이오닉스(Bionics, Korea)에 의뢰하였다(Table 1).
역전사반응을 통해 제작된cDNA를 주형으로, 이들 primer쌍을 사용하여 DWV의 각 유전자들을 PCR 증폭하였다. 이때 각 PCR의 조성은, 2.5U Taq DNA Polymerase(GeneClone, Korea), 2.5mM dNTPs, 10×PCR buffer(25mM MgCl2), 각 10pmol primer set로 하였고, 각 PCR의 조성은 94°C에서 5분간 pre-denaturation 후, 94°C에서 30초, 58°C에서 30초, 72°C에서 1분을 1cycle로 하여, 총 35cycle 반복수행하고, 마지막으로 72°C에서 10분간 post-extension을 수행하였다.
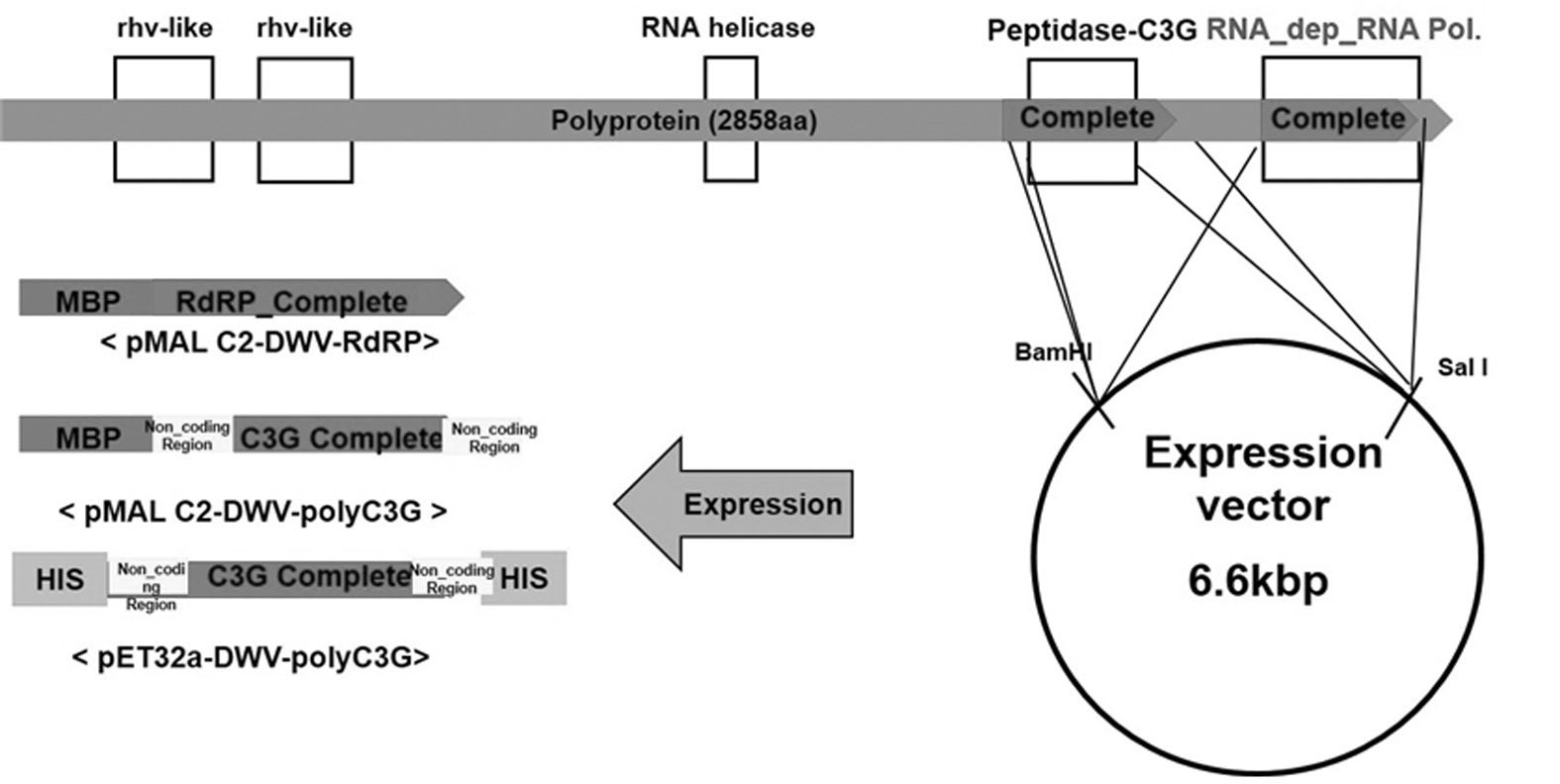
증폭된 각 유전자들은 아가로오스 젤 전기영동을 통하여 그 PCR 산물의 크기를 확인하였으며, 크기가 확인된 PCR산물들은 각기 pBlueXcmⅠ vector(Gene Clone, Korea)에 삽입하여 T-vector cloning을 수행하였다. 선별된 재조합 DNA들은 DNA 염기 서열 분석을 의뢰하였으며(SolGent, Korea), 염기서열이 확인된 재조합 DNA들은, 제한효소로 절단하여, 각기 발현 vector인 pET32a(+) (Novagen, Germany) 그리고 pMALC2(NEB, UK) Expression vector에 삽입시켰다(Fig. 1, Fig. 2).
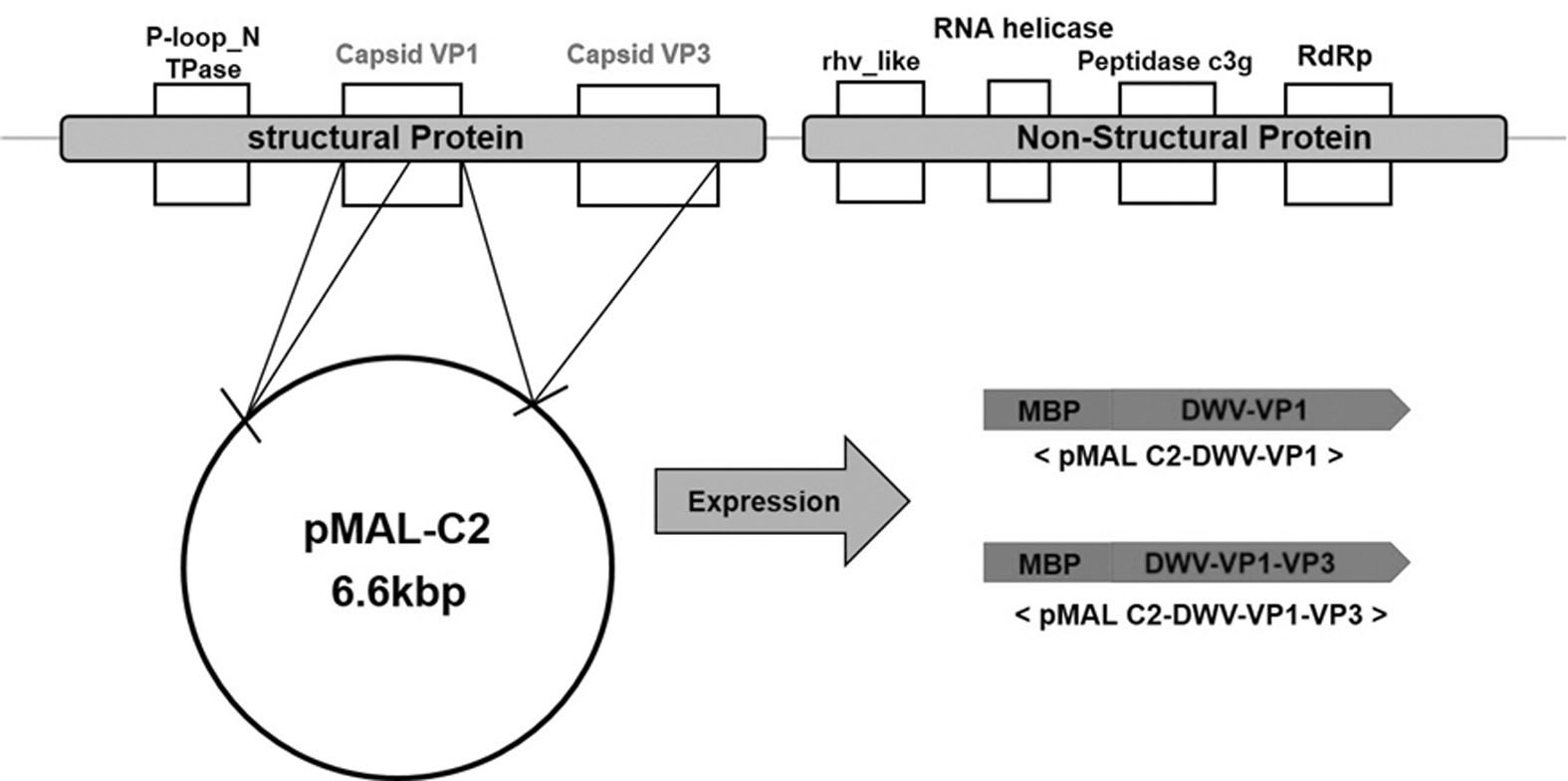
The genetic map of Deformed Wing Virus and basic strategy of molecular cloning for DWV VP1 / DWV VP1 VP3 using pMAL C2 vector.
재조합 DWV 단백질들의 발현
재조합된DWV의 각 유전자 DNA 염기서열과 추론된 아미노산 서열들을 GenBank에 보고된 DWV의 유전체 서열(Accession No. JX878305.1)과 비교 검토한 후, DWV-VP1, DWV-VP1-VP3, DWV-polyC3G, DWVRdRP라 명명하였다. 이들 유전자들은 각기 pMAL-c2 vector와 pET32a vector에, 제한효소 절단 및 연결을 통하여 재조합시켰으며 기본 발현조건으로 재조합 단백질 발현을 유도하였다.
각 재조합 단백질들의 발현용 대장균 숙주는 Escherichia coli BL21(DE3)을 사용하였다. 해당 재조합 단백질의 발현 유도는 정체기에 들어선 각 대장균 배양액(5ml)을 새로운 배지와 1/25의 배율로 희석한 후 Sub-culture를 통하여 OD-600값이 0.6에 도달하였을 때 발현 유도체인 isopropyl-1-thio-β-D-galactopyranoside(IPTG)을 최종농도 0.6mM이 되도록 첨가하였다. 이후37°C에서150RPM의 진탕조건으로 6시간 호기 상태에서 유도 배양하였다.
유도 배양된 각 대장균 배양액은 50ml 원심분리관에 분주하여, 3,000RPM에서 15min, 4°C의 조건에서, S750-4B rotor를 사용하여 원심 분리하였으며(Union 32R, Hanil Inc., Korea), 각 tube당 40ml PBS를 사용하여 2회의 washing 과정을 거친 후, 상층액을 완전히 제거하고, 최종적으로 10ml PBS로 부유시켰다. 이 부유액은 초음파 분쇄기인 VCX 500 (Sonic & Materials Inc., USA)에 S&M 1000 rode를 장착하고, 40% Amplitude, pulse on 0.5 sec, pulse off 0.5 sec, running time 1min 30sec의 조건으로 세포를 분쇄하였으며, 이 조건에서 세포 부유액이 맑아진 것을 확인하였다.
세포 분쇄액은 4°C의 조건에서 3,000RPM에서 15분간 S750-4B rotor를 사용하여 재차 원심 분리하였으며(Union 32R, Hanil Inc., Korea), 이 중 상층액을 새로운 튜브에 옮기고 단백질 정제에 사용하였다. 이중 일부는 발현 여부 확인을 위하여, 5× probe buffer를 가한 후, 99°C에서 10분간 열처리하고, 12% SDS-polyacrylamide gel electrophoresis (SDS-PAGE)를 사용하여 전기영동 분석을 수행하였다.
DWV 재조합 단백질의 정제
SDS-PAGE에서 과발현이 확인된 재조합 단백질(pMAL-DWV-VP1, pMAL-DWV-VP1-VP3, pMALDWV-polyC3G, pMAL-DWV-RdRP, pET32a-DWVpolyC3G)들은 각기 대량생산 및 단백질 순수분리를 수행하였다.
pET32a-DWV-polyC3G의 경우, 재조합 단백질의 N-말단 및 C-말단에 His tagging이 되어 있기에, Ni+column(His trap HP, 1ml, GE Healthcare, USA)을 사용하여 정제를 진행을 하였다.
먼저 His trap HP를 AKTA Start system(GE Healthcare, USA)에 장착하고, 20ml His-washing solution(20mM sodium phosphate, 0.5M NaCl, 40mM imidazole, pH 7.4)을 flow rate 0.5ml/min의 속도로 system내에 주입하였으며, His trap HP를 통과하여 나온 His-washing solution이 UV-detector의 관찰하에 평형에 이르게 됨을 확인하였다. 이후 10ml 세포 분쇄액을 flow rate 0.5ml/min의 속도로 system내에 주입하였으며(His trap HP를 통과하여 나온 여과액은 수집하고 재사용), 이어 동량 10ml의 His-washing solution으로 세척하였다. 이후 동량 10ml의 His-Elution solution(20mM sodium phosphate, 0.5M NaCl, 500mM imidazole, pH 7.4)을 투입하였으며, 추출되는 단백질들은 UV detector의 관찰 하에, 각 1ml씩 fraction collector로 수집하였다. 각 fraction의 단백질 양은 UV detector로 관찰할 때, 일반적으로 3ml(3번 fraction)에서 5ml(5번 fraction)까지 급히 증가하였으나, 이후 급히 감소하였다.
pMAL-DWV-VP1, pMAL-DWV-VP1-VP3, pMALDWV-polyC3G, pMAL-DWV-RdRP의 경우, 발현된 재조합 단백질의 N-말단 부위에 Maltose binding protein(MBP)가 있고, 재조합체는 이와 융합단백질의 형태로 연결되기에, Maltose 및 MBP Column(MBP trap HP, 1ml, GE Healthcare, USA)을 사용하여 정제를 진행하였다.
먼 저 MBP trap HP를 AKTA Start system(GE Healthcare, USA)에 장착하고, 20ml MBP-washing solution(20mM Tris-HCl, 200mM NaCl, 1mM EDTA, pH 7.4)을 flow rate 0.5ml/min의 속도로 system내에 주입하였으며, MBP trap HP를 통과하여 나온 MBP-washing solution이 UV-detector의 관찰하에 평형에 이르게 됨을 확인하였다. 이후 10ml 세포분쇄액을 flow rate 0.5ml/min의 속도로 system내에 주입하였으며(MBP trap HP를 통과하여 나온 여과액은 수집하고 재사용), 이어 동량 10ml의 MBP-washing solution으로 세척하였다. 이후 동량 10ml의 MBP-Elution solution(20mM Tris-HCl, 200mM NaCl, 1mM EDTA, 10mM maltose, pH 7.4)을 투입하였으며, 추출되는 단백질들은 UV detector의 관찰하에, 각 1ml씩 fraction collector로 수집하였다. MBP-trap의 경우도 각 fraction의 단백질양은 UV detector로 관찰할 때, 일반적으로 3ml(3번 fraction)에서 5ml(5번 fraction)까지 급히 증가하였으나, 이후 급히 감소하였다.
결과 및 고찰
DWV유전자들에서 유래된 재조합 단백질의 염기서열 분석
DWV의 각 유전자들은 cDNA 주형 및 해당 특이 primer쌍(Table 1)을 사용하여 PCR 증폭함으로 얻을 수 있었다. 이들 PCR산물은 각기 T-vector cloning하여 유전자를 확보하였으며, 각기 pBX-DWV라 명명하고, 각기 염기서열 결정을 의뢰하였다.
pBX-DWV clone들의 염기서열은 각기 primer 제작시 사용했던 DWV Complete sequence(Accession No. JX878305.1)와 비교 분석을 수행하였으며, 그 결과 확보된DWV 구조단백질인 Capsid protein VP1의 염기서열 (GenBank accession No. KP739938.1)은 GBJX878305.1의 그것과 97%(791/818)의 상동성을 보였으며, 이 염기서열을 근거로 추론된 아미노산 서열의 비교 분석에서는 100%의 상동성을 가지는 것으로 나타났다(Fig. 3, Fig. 7).
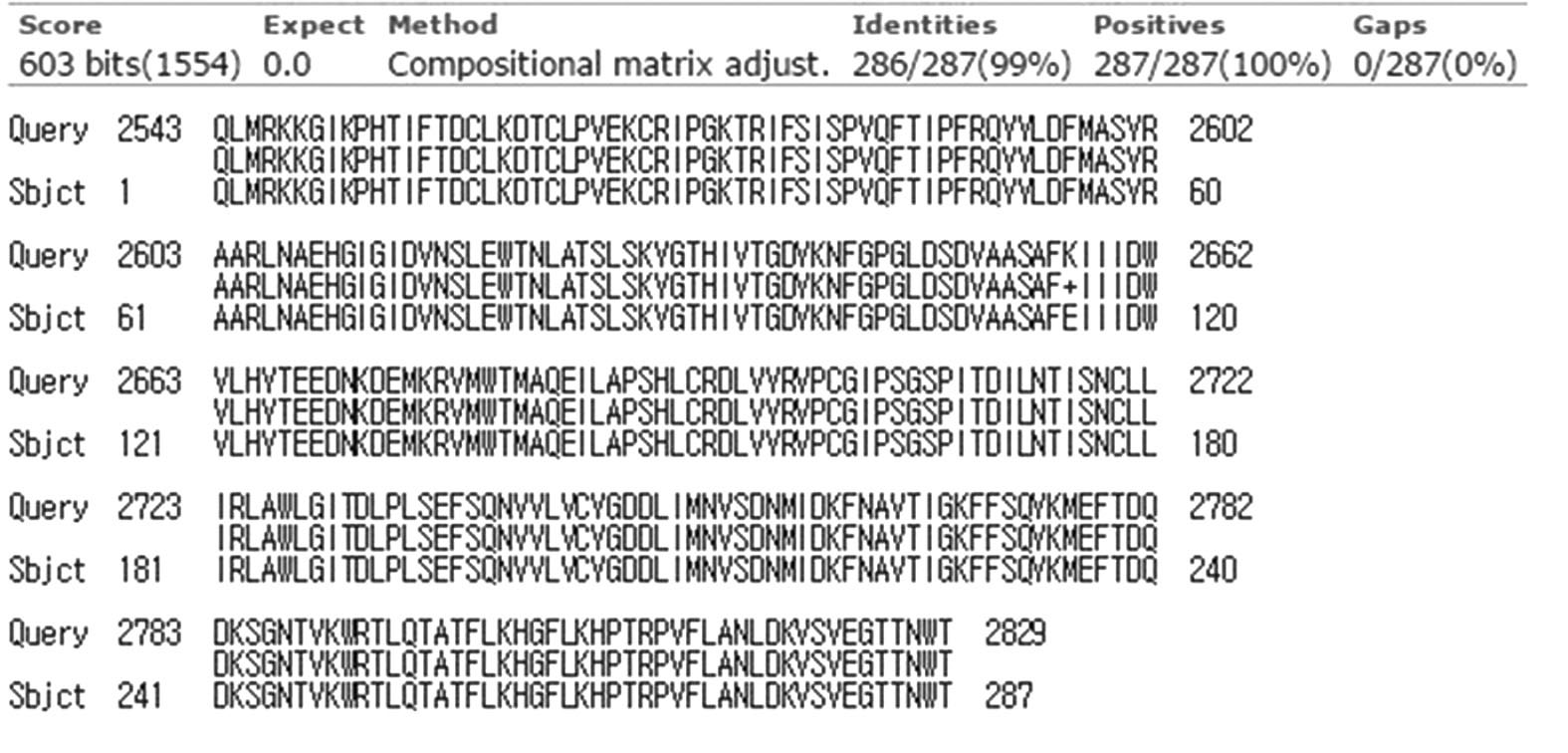
확보된 Capsid protein VP1+VP3 (GenBank accession No. KP751410.1)의 경우, GB-JX878305.1의 그것과 98%(1145/1167)의 염기서열 상동성을 보였으며, 이염기서열을 근거로 추론된 아미노산 서열의 비교 분석에서는 99%의 상동성을 가지는 것으로 나타났다(Fig. 4, Fig. 8).
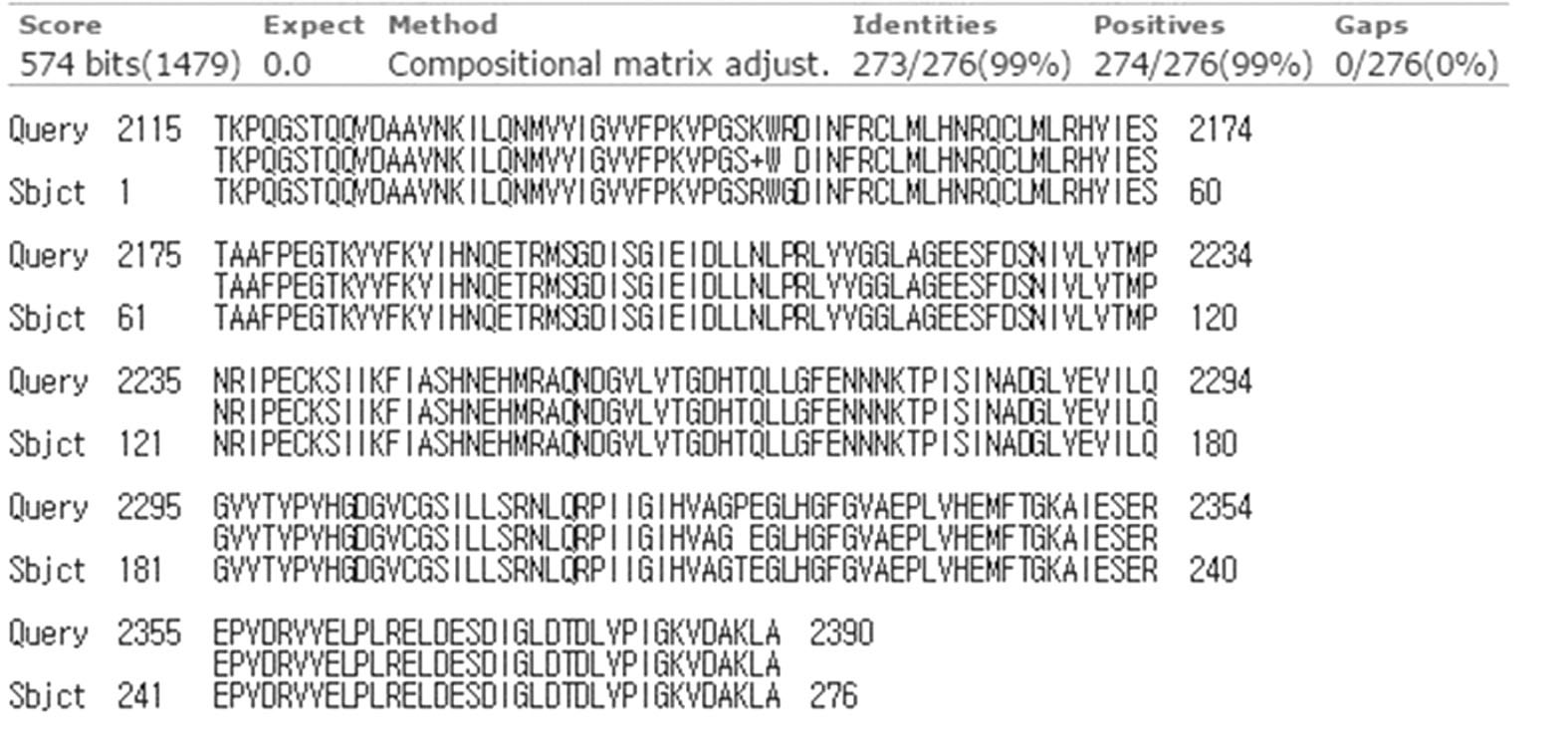
한편, 확보된 DWV 비구조단백질인 RdRP (RNA dependent RNA polymerase)의 염기서열 (GenBank accession No. KP739937.1)은 GB-JX878305.1의 그것과 96%(832/863)의 상동성을 보였으며, 이 염기서열을 근거로 추론된 아미노산 서열의 비교 분석에서는 99%의 상동성을 가지는 것으로 나타났다(Fig. 5, Fig. 9).
또한, 확보된 DWV 비구조단백질인 C3G(C3G peptidase)의 경우, GB-JX878305.1의 그것과 100%(828/828)의 염기서열 상동성을 보였으며, 이 염기서열을 근거로 추론된 아미노산 서열의 비교 분석에서는 99%의 상동성을 가지는 것으로 나타났다(Fig. 6, Fig. 10).

DNA sequence homology between JX878305.1 and pBX DWV VP1. JX878305.1 is complete nucleotides of DWV deposited in GenBank. Alignment analysis was revealed 97% (791/818) homology between nucleotide sequences of pBX DWV VP1 (GenBank accession No. KP739938.1) and 2993 3810 of JX878305.1.

DNA sequence homology between JX878305.1 and pBX DWV VP1 VP3. JX878305.1 is complete nucleotides of DWV deposited in GenBank. Alignment analysis was revealed 98% (1145/1167) homology between nucleotide sequences of pBX DWV VP1 VP3 (GenBank accession No. KP751410.1) and 5420 4618 of JX878305.1.

DNA sequence homology between JX878305.1 and pBX DWV RdRP. JX878305.1 is complete nucleotides of DWV deposited in GenBank. Alignment analysis was revealed 96% (832/863) homology between nucleotide sequences of pBX DWV RdRP (GenBank accession No. KP739937.1) and 8756 9618 of JX878305.1.

DNA sequence homology between JX878305.1 and pBX DWV polyC3G. JX878305.1 is complete nucleotides of DWV deposited in GenBank. Alignment analysis was revealed 100% (828/828) homology between nucleotide sequences of pBX DWV polyC3G and 7472 8299 of JX878305.1.

Amino acid sequence homology between JX878305.1 and pBX DWV VP1. The deduced amino acid sequences based on pBX DWV VP1 (GenBank accession No. KP739938.1) was aligned with the deduced amino acid sequences on 2993 3810 of JX878305.1 The homology between both amino acid sequences was calculated as 100% (273/273).

Amino acid sequence homology between JX878305.1 and pBX DWV VP1 VP3. The deduced amino acid sequences based on pBX DWV VP1 VP3 (GenBank accession No. KP751410.1) was aligned with the deduced amino acid sequences on 5420 4618 of JX878305.1 The homology between both amino acid sequences was calculated as 99% (388/389).
Amino acid sequence homology between JX878305.1 and pBX DWV RdRP clone. The deduced amino acid sequences based on pBX DWV RdRP (GenBank accession No. KP739937.1) was aligned with the deduced amino acid sequences on 8756 9618 of JX878305.1 The homology between both amino acid sequences was calculated as 99% (286/287).
DWV 재조합 단백질들의 발현 확인
재조합 DNA의 염기서열들과DWV의 각 유전자간 상동성 여부를 확인 한 후, 4개 유전자들을 각기 발현 vector system인 pET32a(+) 및 pMAL-C2에 subcloning을 하여 각 재조합 단백질들을 대장균 내에서 과발현시키고자 하였다. 과발현이 확인된 재조합체는 5종이었으며, 이들은 각기 DNA 재조합 프로그램(pDRAW32)을 사용하여 예측한 재조합 단백질의 예상 크기인 pMAL-DWV-VP1, 75kDa; pMAL-DWVVP1-VP3, 93kDa; pMAL-DWV-polyC3G, 81kDa; pMAL-DWV-RdRP, 81kDa;, pET32a-DWV-polyC3G, 50kDa에 근접한 과발현 단백질 밴드를 보여주었다(Fig. 11).
발현유도를 위한 최적 IPTG농도들은 모든 발현계에서 0.1~0.3mM에 나타났으며, 본 연구에서 사용한 유전자들(DWV-VP1, -VP1-VP3, RdRP, C3G)은 거의 모든 경우에서 pMAL vector에 의한 MBP와 융합단백질을 이루어 과발현됨을 보여 주었다. 한편, pET vector에 의하여 C3G의 발현이 확인되었을 뿐 나머지 재조합체에 의한 과발현은 발견되지 않았다(Fig. 11E).

Expression of DWV proteins depends on IPTG concentr ation. All recombinant DNA in E. coli BL21 was cultured until OD600 reach to 0.6 and induced by IPTG to express recombinant DWV proteins. Bacterial cell was collected at 6 hours after induction, respectively. Panel A. pMAL DWV VP1, Panel B. pMAL DWV VP1 VP3, Panel C. pMAL DWV RdRp, Panel D. pMAL DWV polyC3G, and Panel E. pET32a DWV polyC3G. Lane M was protein size marker (T&I). Lane N were cultures without IPTG induction. Lanes 1 to 5 were induced culture with 0.1, 0.3, 0.6, 1.0 and 2 mM of IPTG, final concentration, respec tively.
DWV 단백질의 정제
pET32a-DWV-polyC3G은 Ni+ column을 사용하여 정제를 진행하였으며, pMAL-DWV-VP1, pMAL-DWVVP1-VP3, pMAL-DWV-RdRP pMAL-DWV-polyC3G의 경우에는 MBP column을 사용하여 재조합 단백질들을 각기 순수분리하였다. 재조합 단백질의 순수분리 후 정제된 단백질은 12% SDS-PAGE를 사용하여 결과를 재확인하였다(Fig. 12).
이후 Bradford assay를 통하여 각 정제된 단백질들을 정량하였으며, 그 결과 pMAL-DWV-VP1, pMALDWV-VP1-VP3, pMAL-DWV-polyC3G, pMAL-DWVRdRP, pET32a-DWV-polyC3G의 단백질량은 각각 2.71mg/ml(total 8.13mg of recombinant protein /125ml culture), 4.71mg/ml(total 14.13mg of recombinant protein/125ml culture), 2.07mg/ml(total 6.21mg of recombinant protein/125ml culture), 2.57mg/ml(total 7.71mg of recombinant protein/125ml culture), 1.04mg/ml(total 3.12mg of recombinant protein/125ml culture)로 나타났다.
DWV(Deformed Wing Virus)는 날개불구병이라는 이름으로국내에도 널리 알려진 바이러스이며, 2013년 꿀벌질병관리센터(Center for Honeybee disease control)의 조사에 의하면 국내 꿀벌의 바이러스 질병 중 3번째로 많이 발견되는 바이러스이다(Yoo et al., 2014).
국내의 꿀벌 질병 중 DWV를 포함한 꿀벌 바이러스에 관한 연구는, 병원체의 분자적 검출에 크게 집중되어 있고, 그것도 실험실적 유전자 진단에 초점이 맞추어져 있다고 판단된다. 바이러스 질병을 보다 근본적으로 극복하기 위하여 바이러스에 관한 연구, 즉 바이러스의 특정 단백질들의 특성연구 또는 바이러스의 감염 및 전파에서 바이러스의 각 단백질들의 기능 등에 대한 연구는 불가결할 것이라 생각되나, 국내에서는 이러한 연구들이 미진하다.
본 연구는 DWV의 각 개별 단백질에 대한 연구를 위하여, 대표적 구조단백질인 VP1, VP3와 대표적 비구조단백질이며, 바이러스의 생존에 불가결한 RdRP, C3G를 재조합 DNA방법을 통한 재조합 단백질들로 생산하고자 하였으며, 이는 바로 개별 단백질의 연구를 위하여 연구재료를 생산하고자 하는 것이었다.
본 연구를 통하여 재조합 단백질로 생산된 RdRP(RNA dependent RNA polymerase)는 DWV 유전체를 복제하는 기능의 중요 단백질 효소이나, 이 단백질의 기능에 대한 납득할 만한 가설, 즉 어떻게, 어디서부터, positive-RNA(유전체)를 주형으로 negative RNA(복제형)를 만들고, 또한 negative RNA(복제형)로부터 positive RNA(유전체)를 대량 생산할 수 있는가는 성립되지도 않고 있다.
근래 RdRP는 식물체에서도 발견되어, 식물체의 바이러스 침입 등에 대한 방어 작용인 RNA 간섭(interference; RNAi) 현상에 관여하는 것으로 관심을 모으고 있으며, 국내에 큰 영향을 미친 바 있는 구제역 바이러스(FMDV; Foot-and-Mouth Disease Virus)와 메르스를 유발하는 코로나 바이러스(MERS-CoV; Middle East Respiratory Syndrome Coronavirus) 등도 동일 기능의 RdRP유전자를 가지고 있어, 이 기능에 의하여 바이러스의 자체 복제가 진행된다. 이런 바이러스의 생존에 불가결한 효소들은 항 바이러스제 발굴을 위한 효소저해제 개발에 매우 좋은 대상이 된다. 따라서 본 연구에서 개발된 DWV-RdRP의 생산법은 이 효소를 이용한 저해제 발굴 및 항바이러스제의 개발과 인체에 무해한 꿀벌바이러스에서 유래한 이 RdRP 단백질은 바이러스 연구에 있어서 많은 장점을 보여줄 것으로 기대한다. 또한, 본 연구를 통하여 재조합 단백질로 생산된 C3G(C3G peptidase)는 polyprotein의 형태로 1차 생산된 DWV의 단백질체를 개별 단백질들로 절단, 분리, 활성화시키는 단백질 절단효소이다. 이 역시 DWV의 생존에 불가결한 기능을 수행하여야 하며, 그 절단부위 및 특성에 관한 연구는 새로운 항바이러스제 개발을 위한 신약개발과정에서 시급히 요구된다.
한편, 본 연구를 통하여 재조합 단백질로 생산된 구조단백질 VP1 및 VP3은 바이러스의 외피를 구성하는 주요 단백질들로, 고등동물의 방어기작인 항원/항체반응에서, 일반적으로 우수한 항원결정기(epitope)로 기능한다. 현재의 꿀벌바이러스의 연구에서 꿀벌바이러스의 보다 쉬운 현장 검색을 위한 immunochromatography법의 개발은 매우 시급히 요구되는 것이며, 이를 위한 우수한 항원과 항체의 개발은 선결과제라 판단된다. 본 연구에서 개발된 구조단백질 VP1 및 VP3의 대량생산법은 이런 기대에 부응하는 것이라 판단되며, 이들을 이용한 특이항체의 개발이 바로 이어지기를 기대한다.
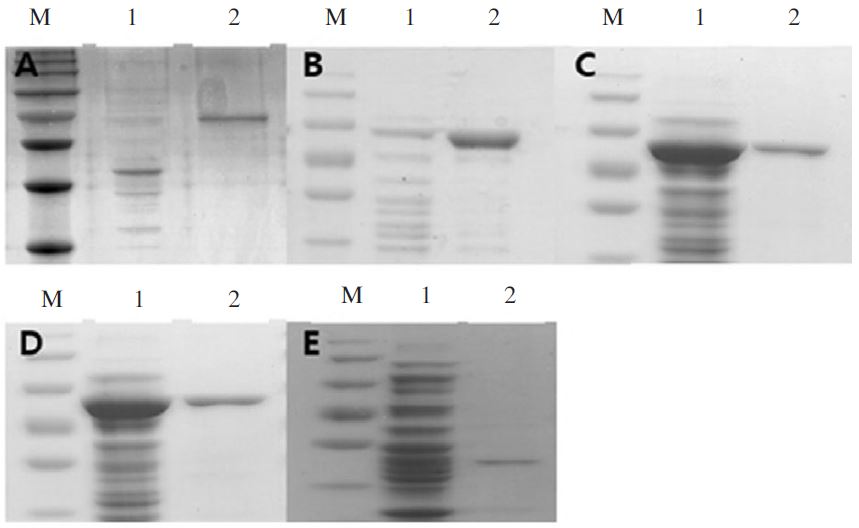
The purifications of recombinant DWV proteins. Panel A. pMAL DWV VP1, Panel B. pMAL DWV VP1 VP3, Panel C. pMAL DWV RdRp, Panel D. pMAL DWV polyC3G, Panel E. pET32a DWV polyC3G. Lane M in each panel was protein size marker (T&I). Lane 1 of each panel were total proteins from each culture induced IPTG, lane 2 of each panel were affinity column purif ied recombinant proteins from each culture.
Acknowledgments
본 연구는 2014학년도 경기대학교 학술연구비(일반연구과제) 지원에 의하여 수행되었음.
Reference
-
Anderson, D. L., and J. W. H. Trueman, (2002), Varroa jacobsoni (Acari:Varroidae) is more than one species, Exp. Appl. Acarol, 24, p165-189.
[https://doi.org/10.1023/A:1006456720416]

- Bailey, L., and B. V. Ball, (1991), Honey bee pathology, 2nd ed, Academic Press, London, England.
-
Ball, B. V., and M. F. Allen, (1988), The prevalence of pathogens in honey bee (Apis mellifera) colonies infested with the parasitic mite Varroa jacobsoni, Ann. Appl. Biol, 113, p237-244.
[https://doi.org/10.1111/j.1744-7348.1988.tb03300.x]

- Ball, B. V., (1989), Varroa jacobsoni as a virus vector, In: Cavalloro, R. Ed, Present Status of Varroa tosis in Europe and Progress in the Varroa Mite Control, OYce for OYcial Publications of the European Communities, Luxembourg, p241-244.
- Lee, H. M., D. B. Lee, S. H. Han, M. L. Lee, Y. K. Lim, B. S. Yoon, (2005), Identification of Deformed Wing Virus from the Honeybee in Korea and Establishment of PCR Detection Method, J. Apicul, 20, p85-94.
- Lim, H. Y., (2013), Development of Novel Rapid Detection Method for Deformed Wing Virus (DWV) using Ultra Fast High Performance PCR (UF HP PCR), J. Apicul, 28, p237-244.
- Lim, H. Y., and B. S. Yoon, (2013), Rapid and Sensitive detection of Deformed Wing Virus (DWV) in Honeybee using Ultra rapid Real time PCR, J. Apicul, 28, p121-129.
- No, J. N., Nguyen Van Phu, M. S. Yoo, Y. H. Park, B. S. Yoon, (2010), Simple and Rapid Method for Detection of Deformed Wing Virus (DWV) by Loop mediated Isoth ermal Amplification (LAMP), J. Apicul, 25, p211-216.
- Yoo, M. S., Y. H. Kim, N. H. Kim, H. N. Jung, Thi, B., Reddy, K., S. C. Jung, S. W. Kang, (2014), Molecular Detection of Honeybee Disease in Apis mellifera and Apis cerana in Korean apiaries, 2013, The 29th Conference of the Apicultural Society of Korea, 2013, p66.