
사탕무(Beta vulgaris) 설탕으로 제조된 사양꿀에서 사탕무 고유 유전자의 검출
Abstract
We could detect the specific gene of sugar beet (Beta vulgaris) from honey produced by sugar of sugar beet using the ultra-rapid real-time PCR (UR-qPCR). In our knowledge, it is the first report in the world that PCR detection of sugar beet-specific gene from adulterated honey. Through extracting the DNA (using CTAB method) from sugar and honey of sugar beet and using the UR-qPCR, sugar beet-specific DNA sequence could be amplified quantitatively until 100 molecules of initial template. By using nested PCR and DNA sequencing, its specificity was confirmed. DNA sequence was matched 100% with mitochondria gene of sugar beet. This finding that trace DNA in adulterated honey could be genetically analysed, would be used as a decisive evidence for the authenticity test of honey.
Keywords:
Sugar beet, Beta vulgaris, Adulterated honey, Detection, Ultra-rapid real-time PCR서 론
현재 사양꿀과 천연꿀을 판별하는 방법은 탄소동위원소비(C13/C12)를 토대로 한 EA-IRMS (Elemental Analyser Isotope Ratio Mass Spectrometry)법과 LC-IRMS(Liquid Chromatography Isotope Ratio Mass Spectrometry) 법이 주로 사용되고 있으며, 이외에 SCIRA (Stable Carbon Isotope Mass Spectrometry), FT-IR (reflectance-Fourier Transform Infrared spectroscopy), NMR spectroscopy, HPLC-ECD (High Performance Liquid Chromatography with Electrochemical Detection)법 등이 사양꿀 판별법으로 보고되었다(Elflein and Raezke, 2008; Bertelli et al., 2012; Rios-Corripio et al., 2012; Sobrino-Gregorio et al., 2017).
그러나, C4 식물군인 사탕수수(Saccharum officinarum)의 설탕으로 생산된 사양꿀의 경우, 탄소동위원소비, 즉 d13C값이 -11‰ 수준으로 측정되어 천연꿀과의 구분에 수월한 반면, C3 식물군인 사탕무(Beta vulgaris)의 설탕으로 생산된 사양꿀의 경우, -22 ~ -30‰으로 측정되며 그 값이 천연꿀과 유사하기에, 탄소동위원소비를 사용한 방법에 의한 사탕무 사양꿀 판별은 불가한 것으로 알려져 있다(유등, 2010).
지금까지 고도의 정제과정을 통해 생산된 설탕에서 사탕수수 또는 사탕무 고유 유전자의 검출은 가능하지 않은 것으로 생각되었고, 설탕을 사용하여 생산된 사양꿀에서 이들 고유 유전자의 검출은 더욱이 불가할 것이라 생각되어 왔다.
그러나, 설탕을 사용한 사양꿀에서는 아니나, 일반 벌꿀에서 화분유전자를 이용하여 주 밀원 유전자를 검출하고자 하는 DNA 분리법과 PCR 증폭법은 이미 보고되었으며(Jain et al., 2013), 같은 목적으로 rice syrup과 rice molasses를 사용한 사양꿀에서 해당 식물체 유전자를 검출할 수 있는 PCR 방법이 제시된 바 있다(Sobrino-Gregorio et al., 2018).
한편, PCR (Polymerase Chain Reaction)법에 의한 DNA 증폭기술이 발전을 거듭함에 따라, 하나의 DNA 주형 분자만으로도 PCR에 의한 증폭이 가능함을 보여주고 있으며, 특히 초고속 PCR은 40회전 이상에서도 DNA 증폭이 계속되어 민감도를 보다 증대할 수 있었다(김 등, 2017). 이런 높은 민감성은 높은 수준의 정제를 거친 설탕에서도, 또한 이 설탕으로 제조한 사양꿀에서도 사탕수수 또는 사탕무 고유 유전자가 초극미량이라도 잔류한다면, PCR을 통해 그 존재를 입증할 수 있을 것이라는 가능성을 높여 주었다.
따라서, 본 연구는 민감도가 높은 PCR법을 적용한다면, 고도로 정제된 설탕에서도, 그리고 이를 사용한 사양꿀에서도 원 식물체의 고유 유전자 존재를 증명할 수 있을 것을 기대하게 되었으며, 현재 사용되고 있는 사양꿀 판별법인 탄소 동위원소비 측정법을 무력하게 만드는 사탕무 사양꿀을 대상으로 해당 사양꿀에서의 사탕무 고유 유전자의 존재를 입증하고자 하였다.
공시재료는 경기대학교에서 2008년 9월 3일 제작된 사탕무 사양꿀과 2008년 8월 11일 한국양봉농협에서 공여받은 사탕무 설탕을 사용하였으며, 생체 시료는 사탕무 씨앗(Sugar Beet seed, 터키산, 2018년 3월; 아시아 종묘[주] 판매)을 사용하였다. 사탕무 씨앗에서 미토콘드리아 일부 유전자를 증폭시켜 molecular cloning하였으며, 재조합된 DNA는 pSugarBeet-mt라 명명하였다. 이에 탑재된 유전자 염기서열은 Genbank에 보고된 사탕무 미토콘드리아 유전자(Accession No. BA000024.1)와 100% 일치됨을 확인하였으며, 본 연구에서 표준 특이 유전자로 사용하였다.
사탕무 설탕물과 사탕무 사양꿀에서 DNA의 추출은 계면활성제인 Cetyl Trimethyl Ammonium Bromide(CTAB)와 phenol/chloroform 혼합액을 사용하여 추출 정제하는 CTAB법을 이용하였다 (윤 등, 2014). 이를 약술하면, 꿀 1ml에 30μl 10% SDS와 3μl proteinase K(20mg/ml)를 넣고 37°C waterbath에서 1시간동안 정치한 후, 100μl 5M NaCl을 넣고 혼합하였으며, 이에 80μl CTAB/NaCl 용액을 추가하고 혼합 후, 65°C에서 10분간 정치시켰다. 700μl phenol/chloroform (24:1)을 추가하고 진탕한 후, 12,000 rpm으로 실온에서 1분간 원심분리하였다. 그런 뒤, 상층부 용액만을 취하여 새로운 tube에서, 0.6배 양의 isopropanol을 넣고 혼합 후, 15,000 rpm으로 실온에서 15분간 원심분리하고 상등액을 제거하였다. 침전에 1ml의 75% ethanol을 가하여 녹여준 뒤, 5분간 정치하고 15,000 rpm으로 실온에서 15분간 원심분리하고 상층액을 제거하였다. DNA 침전은 30분간 자연건조 후 100μl TE buffer에 용해시켰으며, DNA 용액은 Biophotometer (Eppendorf, Germany)를 통해 농도를 측정하고 -20°C에서 보관하며 사용하였다.
사탕무 고유 유전자 검출을 위해서 초고속 PCR 기기인 Genechecker II (GENESYSTEM Co., Korea)를 사용하였으며, PCR 조건은 초기변성 95°C에서 30초 진행한 후, 95°C 변성 3초, 53°C 혼성 3초, 72°C 중합 3초를 1 cycle로 하였으며, 총 cycle 수는 50 cycle로 설정하였다. 각 PCR 반응액은 상기 DNA 용액 중 1μl를 주형으로 사용하였으며, 사탕무 고유 유전자의 검출을 위해 사용된 primer는 Forward primer: 5’-CTCGCTTTATCTCTTTCTACCGG-3’, Reverse primer: 5’-GAAATCTCCTTCAGGTTCAGTCG-3’이며, 각각 최종농도 1μM로 조정하였다.
결과로써, 사탕무 사양꿀 시료에서 추출된 DNA를 통해 사양꿀에 사탕무 고유 유전자가 잔류하고 있음을 확인할 수 있었으며, 이는 증폭곡선, 융점 분석 및 아가로스 전기영동을 통하여 사탕무 고유 유전자임을 입증할 수 있었다(Fig. 1).
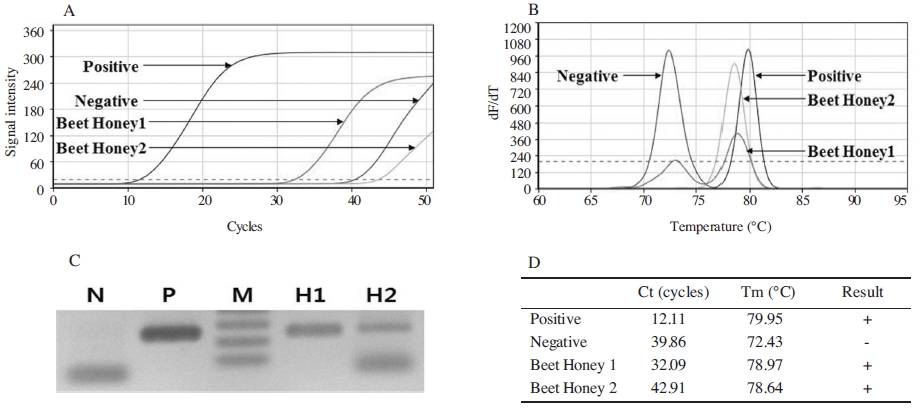
Detection of sugar beet-specific gene using DNA from sugar beet honey. Panel A is the fluorescent graph of amplified target gene. Panel B, a peak graph of Tm against detection of sugar beet-specific gene in honey. Panel C, Lane M is the DNA size marker (100bp, 200bp, 300bp); lane N, non-template; lane P, PCR with pSugarBeet-mt as positive (250bp); lane H1, PCR with DNA from sugar beet honey sample 1; lane H2, from sugar beet honey sample 2. Panel D shows each Ct (Threshold Cycles) and Tm (mid point of melting temperature) of PCRs.
pSugarBeet-mt는 사탕무 고유 염기서열(250bp)을 탑재한 플라스미드로써, 분광광도계에 의해 정량적으로 측정된 플라스미드 용액은 단위부피당 분자수를 정확히 계산할 수 있다. 이를 연속희석하여 각기 PCR의 주형으로 사용하였으며, 이 결과를 분석하여 표준 정량선으로 나타내었다. DNA추출과정에서 사양꿀 1g로부터 100μl의 DNA를 획득하였으며, PCR에 사용된 DNA는 1μl로, 이는 사양꿀 10mg에서 획득한 DNA에 해당된다. 이를 상기 정량선에 대입하여, 꿀 1g에 존재하는 사탕무 고유 유전자의 분자수를 계산하였다.
그 결과, 사양꿀 시료 1의 경우 8.19×103 분자, 사양꿀 시료 2의 경우 1.49×101 분자를 함유하는 것으로 계산되었다.
이처럼 사양꿀 내에 존재하는 사탕무 고유 유전자의 양을 계산할 수 있으며, 이는 천연꿀과 사탕무 사양꿀의 혼합비를 밝힐 수 있는 정량법으로의 정립이 가능할 것으로 사료된다(Fig. 2).
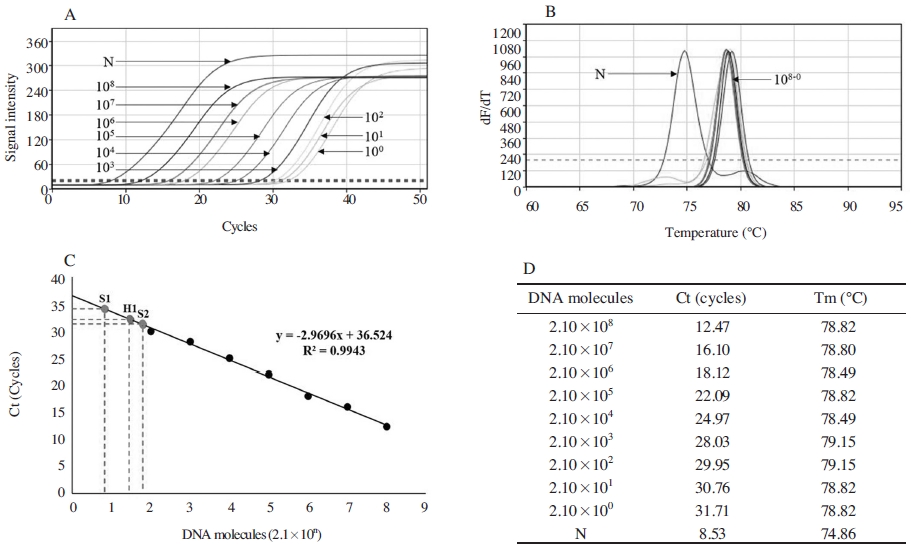
Quantitative detection using pSugarBeet-mt against sugar beet-specific gene. Panel A, the fluorescent graph of amplified target gene according to the serially diluted initial templates. Panel B, a peak graph of Tm. Panel C, the regression line according to the PCRs of panel A. And S1 (Sugar 1), S2 (Sugar 2) and H1 (honey 1) were indicated. Panel D shows each Ct and Tm of PCRs.
사탕무 꿀에서의 사탕무 고유 유전자의 검출 가능한 것을 확인함에 따라, 그 유전자가 사탕무 설탕을 통해 함입되었는지 여부와 사탕무 설탕에서의 사탕무 유전자 증폭을 확인하고자 하였다.
결과로는 사탕무 설탕 시료에서 추출된 DNA를 통해 설탕에 사탕무 고유 유전자의 DNA가 잔류되어 있음을 확인할 수 있었으며, 이는 증폭곡선, 융점 분석 및 아가로스 전기영동으로 입증되었다(Fig. 3).
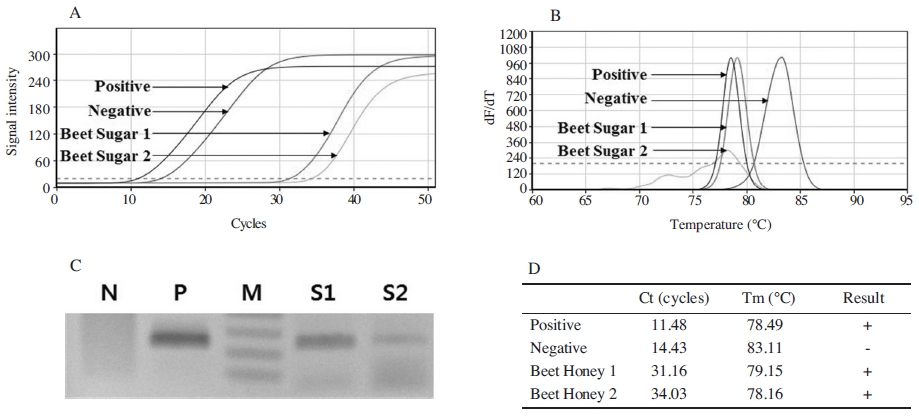
Detection of sugar beet-specific gene using DNA from sugar by sugar beet. Panel A, the fluorescent graph of amplified target gene. Panel B, a peak graph of Tm against detection of sugar beet-specific gene in sugar. Panel C, Lane M is the DNA size marker (100bp, 200bp, 300bp); lane N, non-template; lane P, PCR with pSugarBeet-mt as positive (250bp); lane S1, PCR with DNA from sugar beet sugar sample 1; lane S2, from sugar beet sugar sample 2. Panel D shows each Ct and Tm of PCRs.
각 설탕 0.5g으로부터 100μl의 DNA를 획득하였으며, PCR에 사용된 DNA는 1μl로, 이는 설탕 5mg에서 획득한 DNA에 해당된다. 이를 상기 정량선에 대입하여, 설탕 0.5g에 존재하는 사탕무 고유 유전자의 분자수를 계산하였으며, 설탕 시료 1의 경우 1.36×104 분자와 설탕 시료 2의 경우 1.45×103 분자를 내재하는 것으로 계산되었다.
보다 분명하게 사탕무 고유 유전자의 검출임을 확인하기 위해 1차적으로 증폭된 PCR 산물을 재증폭하는 nested PCR법으로 검증하였다. 주형으로는 사탕무 특이 primer를 사용하여 증폭한 PCR 산물을 1/1,000로 희석하여 사용하였다. 동일한 Genechecker II 기기를 사용하였으며, nested PCR 조건은 초기변성 95°C에서 30초 진행한 후, 95°C 변성 3초, 57°C 혼성 3초, 72°C 중 합 3초를 1 cycle로 하였다. 또한, Ct 값이 30 cycle 이상일 경우 융점분석에서 값이 비특이적 증폭으로 측정되어 유효하지 않다고 판단해 총 cycle 수는 30 cycle로 설정하였다. 각 PCR 반응액은 상기 DNA 용액 중 1μl를 주형으로 사용하였으며, 사탕무임을 검증하기 위해 사용된 primer는 Forward primer: 5’-GAATTGCTGCTTGAAAGTTTTC-3’, Reverse primer: 5’-CAATTCTTCCTATTCTCCTGC-3’이며, 각각 최종농도 1μM로 조정하였다.
결과로는 설탕(Beet Sugar 1, Beet Sugar 2)과 꿀(Beet Honey 1, Beet Honey 2) 시료에서 증폭한 산물이 내부 염기서열을 통해 증폭됨을 확인하였으며, 이는 증폭곡선, 융점 분석 및 아가로스 전기영동에서 증폭산물이 사탕무 고유 유전자임을 확증할 수 있었다(Fig. 4).
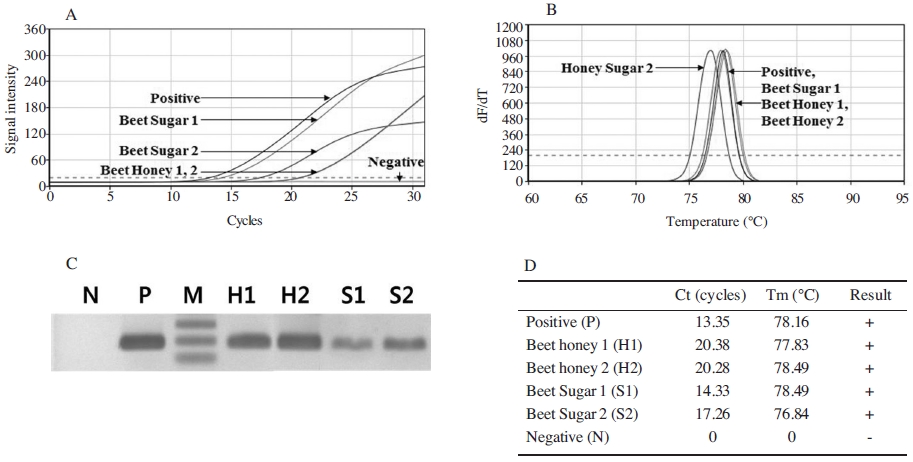
Detection of sugar beet-specific gene using nested PCR. Panel A, the flourescent graph of amplified target gene through nested PCR. Panel B, a peak graph of Tm. Panel C, Lane M is the 100bp size marker (100bp, 200bp, 300bp); lane N, non-template; lane P, PCR with pSugarBeet-mt as positive (196bp); lane S1, nested PCR product with DNA from sugar sample 1; lane S2, sugar sample 2; lane H1, from sugar beet honey sample 1; lane H2, honey sample 2. Panel D shows each Ct and Tm of PCRs.
보다 명확한 사탕무 설탕과 사탕무 사양꿀에서의 사탕무 유전자 검출을 입증하기 위해, PCR을 통해 생산된 PCR 산물을 정제하여 염기서열분석을 수행하였다. 사탕무 씨앗과 사탕무 설탕, 사탕무 사양꿀에서 증폭된 PCR 산물과 nested PCR을 증폭해 얻은 PCR 산물은 Megaquick-spin™ Total fragment DNA purification kit (iNtRON, Korea)를 이용하여 정제한 뒤, Bionics사에 염기서열분석을 의뢰하였다.
Sanger sequencing을 통해 염기서열을 분석한 결과, 사탕무 씨앗, 사탕무 설탕, 사탕무 꿀에서 검출된 증폭산물은 사탕무 미토콘드리아 유전자(GenBank accession No. BA000024.1)와 모두 100%의 일치함을 보여 사탕무 고유 유전자임을 확인하였다. 또한, Nested PCR product을 이용한 염기서열 분석에서는 6번째 염기서열인 아데닌의 결손이 존재했으며, 상동성은 99.2%로 나타났다. 이를 통해, 각각의 사탕무 씨앗과 사탕무 설탕, 사탕무 꿀에서 검출된 DNA는 사탕무 고유 유전자임을 입증할 수 있었으며, 고도의 정제과정을 거친 사탕무 설탕과 사탕무 설탕을 이용해 생산된 꿀에서 주원료가 된 사탕무 유전자가 잔류함을 알 수 있었고, 잔류 DNA는 검출될 수 있음을 증명하였다(Fig. 5).

Sequence alignment about PCR product from seed, sugar and honey of sugar beet. BA000024.1 is Beta vulgaris subsp. vulgaris mitochondrial DNA, complete genome from Sapporo, Japan. Sugar beet seed is DNA sequence of its PCR product. Sugar beet sugar, Sugar beet honey and Nested PCR product were generated in this study. All of the detected DNAs were confirmed to be mitochondrial genes of sugar beet.
일반적으로 사용되는 PCR기기에서 본 검출법이 적용될 수 있는지 실험하고자, 일반 PCR기기인 PTC-200 Peltier thermal cycler (MJ Research, USA)를 사용하여 사탕무 고유 DNA 증폭을 시행하였다. PCR 조건은 초기변성 95°C에서 5분 진행한 후, 95°C 변성 30초, 53°C 혼성 30초, 72°C 중합 30초를 1 cycle로 하였으며, 총 cycle 수는 40 cycle로 설정하였다. 각 PCR 반응액은 DNA 용액 중 1μl를 주형으로 사용하였으며, 사탕무 고유 유전자의 검출을 위해 사용된 primer는 Forward primer: 5’-CTCGCTTTATCTCTTTCTACCGG-3’, Reverse primer: 5’-GAAATCTCCTTCAGGTTCAGTCG-3’이며, 각각 최종농도 1μM로 조정하였다.
사탕무 씨앗과 사탕무 설탕, 사탕무 꿀에서 증폭된 산물은 전기영동을 통해 사탕무 고유 유전자임(250bp)을 확인하였고 더불어, 각 증폭 band는 nested PCR로도 확인되었으며, 예상된 196bp의 band를 관찰할 수 있었다(자료미제시). 이 결과는 사탕무 고유 유전자의 증폭으로 판단되며, 각 시료의 증폭을 확인함으로써 일반 PCR기기에서도 본 검출법이 충분히 적용가능함을 보여주었다(Fig. 6).
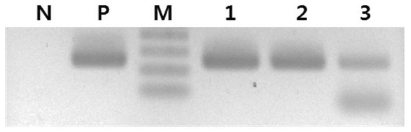
Conventional PCR of sugar beet-specific gene in seed, sugar, honey. Lane M is the 100bp ladder size marker (100bp, 200bp, 300bp, 400bp); lane N, non-template; lane P, amplification of PCR with pSugarBeet-mt as positive (250bp); lane 1, PCR with DNA from sugar beet seed sample; lane 2, from sugar beet sugar sample; lane 3, from sugar beet honey sample. Sugar beet-specific gene were well amplified with different DNA of sources using conventional PCR.
본 연구는 초고속 PCR법과 nested PCR법 그리고 Sanger sequencing을 이용하여 사탕무 꿀과 사탕무 설탕에서 사탕무 고유 유전자기 특이 검출될 수 있음을 증명한 것이며, 사탕무 고유 유전자의 양은 정량될 수 있음을 보여주었다.
벌꿀에 잔류된 밀원 등의 화분 유전자를 PCR법을 이용하여 특이 검출한 연구, 즉 벌꿀에서 화분 유전자의 프로파일링의 연구들은 보고된 바 있었으나(Jain et al., 2013; Sobrino-Gregorio et al., 2018), 이들 연구결과는 직접적으로 사양꿀 판별법으로 제시될 수 없었으며, 본 연구와 같이 고도의 정제과정을 거친 설탕을 당원으로 제조한 사양꿀에서 해당 당원, 즉 설탕의 특이 유전자를 특이 검출한 것은 아니었다.
사탕무 설탕과 사탕무 사양꿀 속에 존재하는 사탕무의 고유 유전자는 초극미량이며, 이를 정확히 정량/정성 분석을 하기 위하여, 사양꿀에서 보다 효과적인 DNA 분리법이 요구되고 있으며, 또한 대량의 벌꿀시료들을 보다 빠르게 사탕무 사양꿀 판별을 수행하기 위하여, 본 정량 PCR을 사용하는 자동화 사탕무 사양꿀 판별방법도 개발되어야 할 것이다. 본 연구에 의하여 발견된 사탕무 사양꿀에서 사탕무 고유 유전자의 존재 입증은 국내외 사탕무 사양꿀의 진위 판정법으로 크게 활용되기를 기대한다.
Acknowledgments
본 연구는 2018학년도 농촌진흥청 연구사업(세부과제번호: PJ01408002) 지원에 의하여 수행되었으며, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 첨단생산기술개발사업(115102-03)과 중소기업청에서 지원하는 2017년도 산학연협력 기술개발사업(C0563751)에 의한 연구 결과물임을 밝힙니다.
References
-
김정민, 임수진, Truong A Tai, 홍기정, 윤병수, (2017), 초고속 유전자 증폭법을 이용한 벌집꼬마밑빠진벌레(Aethina tumida)의 신속한 검출 기법 개발, J. Apiculture, 32, p119-131.
[https://doi.org/10.17519/apiculture.2017.06.32.2.119]

- 유응철, 공영건, 윤병수, (2010), 불량꿀 검사를 위한 개선된 분석 방법에 관한 연구, J. Apiculture, 25, p63-76.
- 윤병수, 한상훈, 임희영, (2014), 분자생물학 연구방법론 Ⅳ, 경기대학교 출판부, p95-97.
-
Bertelli, D., M. Lolli, G. Papotti, L. Bortolotti, G. Serra, and M. Plessi, (2010), Detection of adulteration by sugar syrups using one-dimensional and two-dimensional high-resolution Nuclear Magnetic Resonance, J. Agric. Food Chem, 58, p8495-8501.
[https://doi.org/10.1021/jf101460t]

-
Boffo, E. F., L. A. Tavares, A. C. T. Tobias, M. M. C. Ferreira, and A. G. Ferreira, (2012), Identification of components of Brazilian honey by H NMR and classification of its botanical origin by chemometric methods, LWT - Food Sci. Technol, 49, p55-63.
[https://doi.org/10.1016/j.lwt.2012.04.024]

-
Elflein, L., and K. Raezke, (2008), Improved detection of honey adulteration by measuring differences between 13C/12C stable carbon isotope ratios of protein and sugar compounds with a combination of elemental analyser e isotope ratio mass spectrometry and liquid chromatography - isotope ratio mass spectrometry (δ13C-EA/LC-IRMS), Apidologie, 39, p574-587.
[https://doi.org/10.1051/apido:2008042]

-
Jain, S. A., F. T. d. Jesu, G. M. Marchioro, and E. D. d. Araujo, (2013), Extraction of DNA from honey and its amplification by PCR for botanical identification, Food Sci. Technol. Campinas, 33, p753-756.
[https://doi.org/10.1590/s0101-20612013000400022]

-
Ohmenhaeuser, M., Y. B. Monakhova, T. Kuballa, and D. W. Lachenmeier, (2013), Qualitative and quantitative control of honeys using NMR spectroscopy and chemometrics, Analytical Chemistry, 2013, p1-9.
[https://doi.org/10.1155/2013/825318]

-
Riberio, R. d. O. R., E. T. Marsico, C. d. S. Carneiro, M. L. G. Monteiro, C. C. Junior, and E. F. O. d. Jesus, (2014), Detection of honey adulteration of high fructose corn syrup by low field Nuclear Magnetic Resonance (LF1H NMR), J. Food Eng, 135, p39-43.
[https://doi.org/10.1016/j.jfoodeng.2014.03.009]

-
Rios-Corripio, M. A., M. Rojas-Lopez, and R. Delgado-Macuil, (2012), Analysis of adulteration in honey with standard sugar solutions and syrups using attenuated total reflectance-Fourier transform infrared spectroscopy and multivariate methods, CyTA - J. Food, 10, p119-122.
[https://doi.org/10.1080/19476337.2011.596576]

-
Sobrino-Gregorio, L., S. Vilanova, J. Prohens, and I. Esriche, (2018), Detection of honey adulteration by conventional and real-time PCR, Food control, 95, p57-62.
[https://doi.org/10.1016/j.foodcont.2018.07.037]

-
Tosun, M., (2013), Detection of adulteration in honey samples added various sugar syrups with 13C/12C isotope ratio analysis method, Food chem, 138, p1629-1632.
[https://doi.org/10.1016/j.foodchem.2012.11.068]
