Multi-point PCR법을 이용한 Black Queen Cell Virus (BQCV) 검출법 개발
Abstract
BQCV multi-point PCR was developed as a rapid multiplex detection method for BQCV, one of the viral pathogens of honeybees. It could detect BQCV specific genes qualitative as well as quantitative detection based on ultra-rapid PCR. Three primer pairs (RNA dependent RNA polymerase, capsid protein, 3C like protease) were specifically designed for accurate the detection and were optimized for minimizing the detection time and increasing the sensitivity. Our advanced diagnostic system have the accuracy by lowering the concern about the variation in the BQCV detection site. In addition, it should be an opportunity to identify mutations that are mixed with other viruses.
Keywords:
Ultra-Rapid Real-time PCR (UR-qPCR), Black Queen Cell Virus (BQCV), Honeybee, Apis mellifera ( A. mellifera), Muti-point PCR, Reverse transcription (RT)서 론
국내에서 2007년 최초로 Black Queen Cell Virus (BQCV)가 발견된 이후 (유 등, 2007), 꿀벌에서의 BQCV 감염 수준은 국내외로 극심하였다. 하지만, 대부분의 꿀벌 바이러스 질병과 같이 뚜렷한 증상이 알려져 있지 않아 감염을 인지하기 어려우며, 강군 양성 외에는 특별한 치료법이 개발되어 있지 않아 질병의 사전 예방과 효과적인 차단을 위해 정확하고 신속한 진단이 절실히 필요한 실정이다.
또한, 꿀벌 바이러스에서 유전자 변이 및 다른 유사 바이러스와의 융합 (fusion)을 통해 꿀벌에 감염되는 사례가 다수 밝혀지고 있다 (Dalmon et al., 2017). 특히, Deformed Wing Virus (DWV)의 경우, 꿀벌 응애에 감염되는 바이러스인 Varroa destructor Virus-1 (VDV-1)과 융합되어 서양종 꿀벌 (Apis mellifera)에서 검출되어지고 있어 (Wang et al., 2013), DWV 이외의 바이러스에서도 바이러스 간의 유전자 교차가 의심되고 있다. 더불어, BQCV의 유전자형을 분석한 선행연구에서는 균주간의 유전자가 상이함을 밝힌 바 있어 BQCV를 검출하는 과정에서 고도로 정교화된 검출법이 요구되고 있다 (Tapaszti et al., 2009).
최근 신속한 검출을 위해 꿀벌 질병검사에서 주로 Ultra-rapid Real-time PCR (UR-qPCR)이 사용되고 있다. UR-qPCR은 Rapi:PCR칩이 넓은 표면적을 이용하여 PCR 조성액에 효과적인 열전달을 통해 각 PCR 단계의 시간을 최소화할 수 있게 만들어졌으며, 2008년 꿀벌 병원성 질병인 American Foulbrood (AFB) 검출법에 적용되어 처음으로 소개되었다 (Han et al., 2008). 그 이후로도 UR-qPCR을 이용한 검출법은 꾸준히 연구되어지고 있다 (김 등, 2017a; 김 등, 2017b; 김 등, 2017c; 임 등, 2017;김 등, 2018).
그동안 Black Queen Cell Virus의 진단을 위해 Real-time PCR을 이용한 많은 연구들이 수행되어졌으며 (유 등, 2008), 최근에는 Ultra-rapid Real-time PCR을 이용한 다중 바이러스 질병 진단에서 한 가지 병원체로써 보고되었다 (Wang et al., 2016). 그러나 이는 다중 PCR 증폭으로 인한 BQCV 단일 질병에 대한 정확한 진단이 불가피하고 한 쌍의 특이 프라이머를 사용하여 변이가 존재하는 바이러스의 검출이 힘들었다.
본 논문에서 BQCV의 다양한 특이 프라이머를 이용한 multi-point URRT-qPCR을 개발하였으며, one-step PCR을 이용해 20분 이내의 빠른 BQCV 진단을 가능하게 하였다. 개발된 BQCV의 진단법은 높은 민감도와 신속성을 가지고 있어 질병 감염 초기단계에서의 진단이 정확하게 이루어질 수 있음을 보고자 한다. 더불어, BQCV 내부 유전자 변이가 일어나 검출이 어려운 경우에도 특이 프라이머 3쌍을 이용하여 검출할 수 있으며 다른 virus와의 혼재된 변이를 확인할 수 있는 계기가 될 것이다.
재료 및 방법
꿀벌 시료 확보 및 Total RNA의 추출
본 연구에 사용된 BQCV 감염 의심 시료는 제주도 및 경기도 수원시 양봉장, 전라남도 여수 양봉장에서 각각 확보되었으며 이를 이용하여 RNA 추출을 진행하였다. RNA 추출은 R&A Blue Kit (Takara, Japan)를 사용하여 제작사의 방법에 따라 수행하였다. 날개가 제거된 꿀벌 1마리 (90 μg)를 통해 total RNA를 확보하였으며, 추출된 total RNA는 spectrophotometer로 정량하였고 이는 -70℃에 보관하였다.
BQCV 특이 유전자의 재조합 DNA 제작 및 프라이머의 선별
재조합 DNA는 pBQ-RdRp, pBQ-3CL, pBQ-VP로 3가지를 제작하였다. 이들의 사용목적은 꿀벌 시료에서 BQCV 검출시 Tm값을 비교하여 검출여부를 판정하는 표준 시료로써 사용되었다 (Fig. 1). GenBank Accession No. EF517515의 RNA dependent RNA pol ymerase (RdRp), 3C like protease (C3G), capsid pro tein (VP)을 target으로 제작되었으며 각 크기는 831 bp (4457-5287), 318 bp (3452-3769), 2,415 bp (5848-8262)이다.
Determination of annealing and polymerization time in multi-point URRT-qPCR against BQCV. Panel A is minimum of annealing time for BQCV URRT-qPCR. Panel B, minimum of polymerization time for BQCV-URRT-qPCR.
BQCV에 대한 특이 PCR 프라이머 3쌍은 BQCV (GenBank Accession No. EF517515)의 유전체 내부 open reading frame (ORF) 중 capsid protein, RNA-dependent RNA-polymerase (RdRp), 3C like protease를 암호화 하는 위치에서 각각 특이 프라이머 쌍을 설계하였다 (Table 1). 각 프라이머 제작은 bionics사 (Korea)에 의뢰하였다.
BQCV 재조합 DNA를 이용한 검출한계 및 PCR 단계 최소화
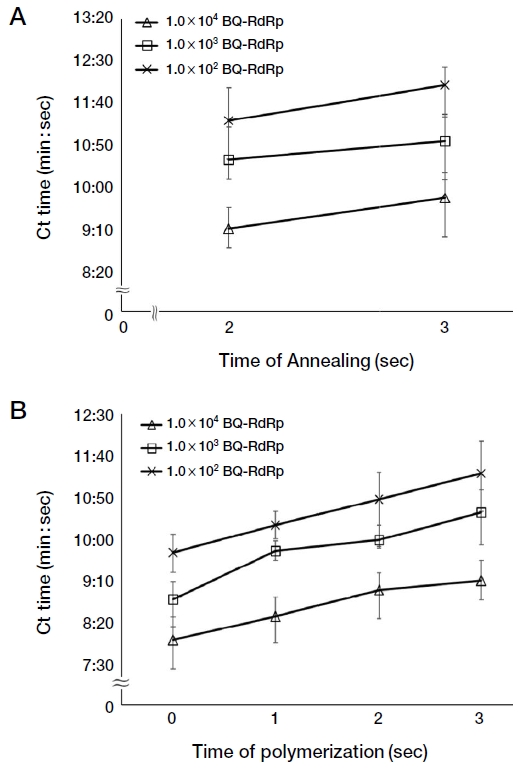
Taq DNA polymerase가 높은 온도에 노출되는 시간을 최소화하여 Taq DNA polymerase 손상을 줄이고 BQCV의 빠른 검출을 위해 최소 검출 시간을 측정하고자 초고속 PCR의 각 cycle 중 혼성시간을 3초에서 1초까지 감소시켜 각 온도와 시간 조건에 따른 Ct (Threshold cycles) 값과 Ct 값이 측정되는 시간 (Ct time)을 측정하였고 중합시간은 3초에서 0초까지 감소시켜 최소 검출 가능 시간을 측정하였다.
BQCV 특이 유전자 초고속 검출을 위한 Multi-PCR 조건 최적화
재조합 DNA에 대한 프라이머쌍의 혼성 온도와 농도가 최적일 때, URRT-qPCR의 특이성 및 신속성에서 강점을 가지기 때문에 혼성 온도와 농도의 최적화 실험을 수행하였다.
초고속 PCR은 GenecheckerTM (Genesystem Co., Korea)를 사용하였으며, 이때, 초고속 PCR의 시간 조건은 앞선 실험을 통해 조정된 시간으로 설정되었다. 혼성 온도는 45~65℃의 범위에서 진행하였으며 프라이머의 농도는 1 μM로 통일하여 비교하였다. 각 부위별 특이 프라이머 1쌍과 2×Rapi Master mix (Genesystem Co., Korea), BQCV 재조합 DNA로 총 10 μL의 반응액을 조성하여, Ultra-rapid PCR을 진행하고 각 Ct 값과 최대 형광값을 측정하여 최적 조건을 설정하였다.
BQCV multi-point URRT-qPCR을 위한 Reverse transcription 시간 최적화
BQCV의 초고속 검출에 있어서 병원체의 신속한 cDNA 합성은, PCR에서의 목적 유전자 증폭을 더 빠르게 시작할 수 있게 하며, 결과적으로는 전체 검사 소요시간을 단축할 수 있게 하는 중요 요소이다. 따라서 PCR 검출 결과에 영향을 주지 않으며, 초고속 PCR에 적용 가능한 역전사 시간은 어느 정도인지, 또한 시간에 따라 역전사 효율이 어떤 양상으로 변화하는지 확인하고자, 그 시간을 10분, 7분, 5분, 3분, 1분, 0분으로 50℃의 reverse transcription을 각기 수행하였으며, BQCV 감염 시료 (경기도 수원, 제주도 제주시)로부터 추출한 30 ng의 total RNA를 주형으로 반응시켜 각 조건에서 Ct 값을 비교·분석하여 정량을 실시하고 Ct time을 상호 비교하였다. RT 시간을 제외한 조건은 앞선 실험을 통해 최적화된 조건으로 사용되었다.
현장 적용을 위한 BQCV 감염 검체를 사용한 확정 시험
BQCV multi-point URRT-qPCR의 유효성을 평가하기 위하여, 경기도 수원, 전라남도 여수, 제주도 시료로부터 추출한 Total RNA 중 30 ng을 주형으로 BQCV 검출을 실시하였다. 하나의 시료에 대해 RdRp, 3C like protease, capsid protein을 특이적으로 검출하는 3쌍의 프라이머로 모두 검사하였으며 PCR 반응 조건은 앞선 실험을 통해 최적화된 조건으로 사용되었다. 모든 실험은 3번 이상 반복되었다.
결과 및 고찰
BQCV 재조합 DNA를 이용한 검출한계 및 PCR 단계 최소화
초기변성은 15초, 변성은 1초로 고정하였다. 이 측정에서는 재조합 DNA를 1.0×108 분자부터 1.0×100 분자까지 연속 희석하여, 초고속 PCR의 주형으로 사용하였으며, 초고속 PCR의 각 단계에서 시간 조건을 가감하여, BQCV multi-point URRT-qPCR의 최소 검출 시간을 도출하였다.
1.0×103 분자인 pBQ-RdRp를 기질로 사용한 초고속 PCR 기준에서 각 cycle의 혼성시간을 3초에서 1초까지 조정한 결과, 각 Ct 값은 3초에서 30.07 cycles (Ct time; 10분 14초), 2초에서 29.98 cycles (Ct time; 9분 45초)로 측정되었으며 BQCV 특이 유전자의 증폭을 확인할 수 있었다. 혼성시간이 1초일 경우, DNA 증폭을 나타내는 형광곡선이 1.0×108 분자부터 1.0×100 분자까지 유사한 Ct 값인 6.50±0.18 cycles에서 동시에 증폭됨을 볼 수 있어 정량적 측정이 불완전하다고 판단하였다 (자료 미제시). 같은 조건에서 각 혼성시간만 3초와 2초로 감소시킨 초고속 PCR과 비교한 결과, 혼성시간 2초에서 29초의 감소세를 보였다. 혼성시간을 3초에서 1초로 조정하여 비교하여 검출 가능 최소 혼성시간은 2초로 판단하였다 (Fig. 1A).
1.0×103 분자인 pBQ-RdRp를 기질로 사용한 초고속 PCR에서 각 cycle의 중합시간을 3초에서 0초까지 조정한 결과, 각 Ct 값은 3초에서 29.98 cycles (Ct time; 9분 45초), 2초에서 31.79 cycles (Ct time; 9분 50초), 1초에서 32.77 cycles (Ct time; 9분 34초), 0초에서 29.91 cycles (Ct time; 8분 14초)로 측정되었으며 BQCV 특이 유전자의 증폭을 확인할 수 있었다. Ct time은 중합시간 0초와 1초를 비교했을 때, 1분 20초 차이로 큰 감소세를 보였다. 중합시간을 3초에서 0초로 조정하여 비교하였을 때의 검출 가능 최소 중합시간은 0초로 판단되었다 (Fig. 1B).
변성, 혼성, 중합을 각 1초, 3초, 3초로 설정한 기본조건과 변성, 혼성, 중합을 각 1초, 2초, 0초로 설정한 최단조건과의 비교를 진행하였다. cycle 시간의 감소에 따른 Ct 값 차이인 dCt 값은 오차가 1.71 cycles로 크게 차이를 보이지 않아 각 step당 cycle 시간의 감소가 검출에서 불리하게 작용하지 않았다. 각 step 시간 감소에 따른 Ct time의 차이를 비교한 결과, 각 분자 수에 따라 Ct time의 차이는 최단조건이 기존조건보다 108 분자일 때, 69.74초 빠르게 나타났고, 107 분자에서 검출한계인 102 분자까지 77.06초, 64.49초, 63.92초, 90.14초, 119.92초, 115.35초 빠르게 나타났다. 108~102 분자에서 1초, 2초, 0초가 평균 85.80±23.53초로 빠르게 나타났다. 기존조건인 1초, 3초, 3초와 비교하였을 때, 검출에서 더욱 유리하게 나타났다 (Fig. 2).
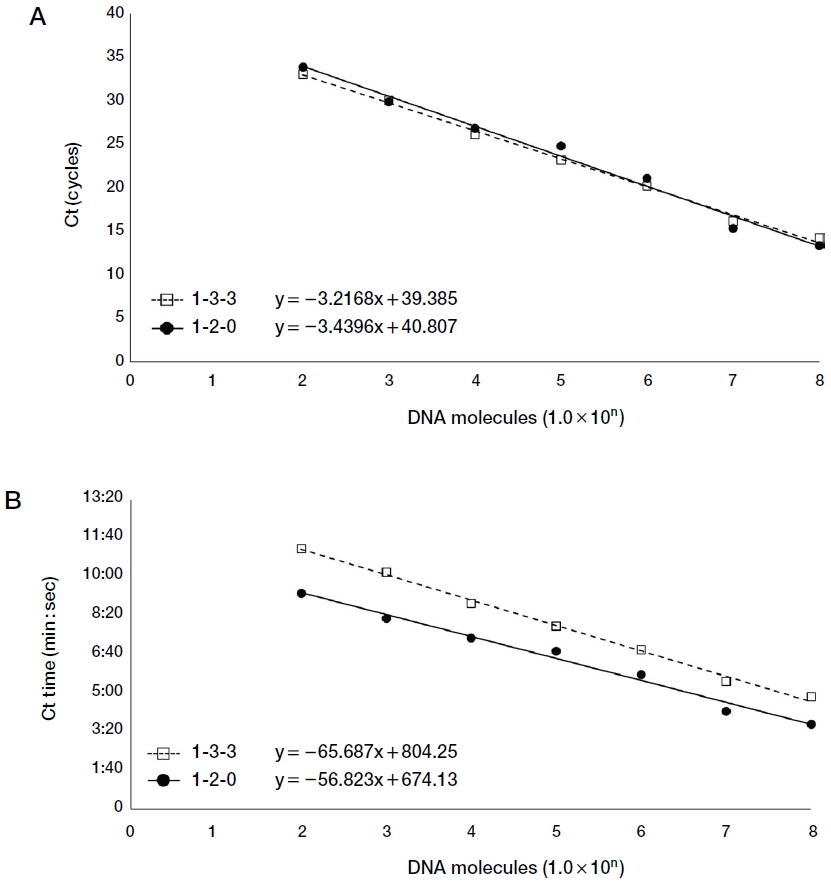
Comparison between the shortest condition (1 sec-2 sec-0 sec) and the stable condition (1 sec-3 sec-3 sec). Panel A is comparison about Ct (cycles) between the shortest and the preexistence condition. Panel B, Comparison about Ct time (min : sec) between the shortest and the stable condition.
한편, 중합시간을 0초까지 내렸음에도 불구하고 증폭그래프의 양상이 기존조건의 양상과 유사하게 나타났다. Taq DNA polymerase의 이전 연구에 따르면 70℃에서 60 nt/sec이상의 효율을 나타내었으며, 55℃에서는 24 nt/sec의 효율을 나타내었으며, 90℃ 이상의 높은 온도에서는 매우 적은 양의 DNA 합성이 이루어졌다고 밝혔다 (Innis et al., 1990). 이를 통해 55℃에서 95℃로 온도를 올리는 동안 Taq DNA polymerase가 작동 가능한 점을 보아 온도를 올리는 Ramping time인 2초만으로도 충분히 중합 (100~300 bp)이 가능하다는 점을 보여주었다. 또한 일반 PCR의 경우, Taq DNA polymerase의 손상으로 PCR cycle이 30~45 cycles로 제한되어있으며, 이는 PCR의 발달을 제한하는 한 요소였다. 낮은 분자 수는 보통 40 cycles 이상에서 검출되기 때문에 일반 PCR의 경우 검출의 어려움이 있다 (김 등, 2017). 본 연구에서는 최소 검출시간을 설립해나가면서 Taq DNA polymerase의 손상을 최소화하여 PCR을 50 cycles까지 가능하게 하였으며, 이를 통해 저분자 (102~100 분자)를 검출하게 하였다.
BQCV 특이 유전자 초고속 PCR을 위한 PCR 조건 확립
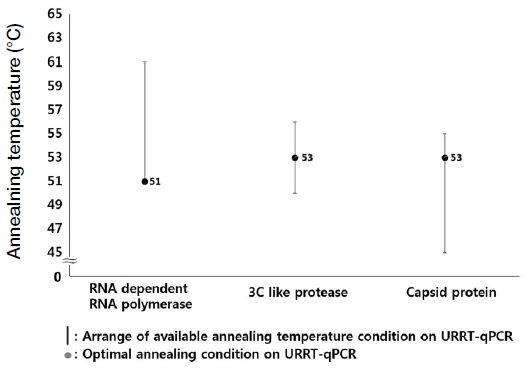
BQCV의 RdRp 부분 검출을 위한 초고속 PCR의 검출가능 혼성온도는 51~61℃의 범위이며, 범위 이외의 온도에서는 정량적 검출이 어려워 최적조건의 정립에서 배제하였다 (자료 미제시). 혼성온도가 51℃에서 102 분자일 때, Ct 값은 34.5 cycles로 가장 빠르게 측정되었으며, PCR 소요시간인 Ct time은 10분 13초이었다. 따라서 최적 혼성온도는 51℃로 확립하였으며, 최소 검출 시간 (10분 13초) 내 우수한 DNA 증폭량 (F; 212)을 확인할 수 있었다.
BQCV의 3C like protease 부분은 50~56℃의 범위이며, BQCV의 capsid protein 부분은 45~55℃의 범위이다. 이 중 3C like protease은 102 분자일 때, 53℃에서 34.14 cycles로 가장 빠르게 측정되었으며, capsid protein은 53℃에서 102 분자일 때, Ct 값은 35.18 cycles로 가장 빠르게 측정되었다.
BQCV의 multi-point 검출을 위해서는 혼성온도의 통합이 필수적임으로 두 부위 (3C like protease, capsid protein)에서 최적으로 검출된 53℃를 혼성온도로 통일하였다 (Fig. 3).
BQCV 초고속 RT-PCR을 위한 Reverse transcription 시간 최적화
cDNA 합성 및 PCR에 영향을 주지 않는 최적의 역전사 시간을 도출하고자 시료의 Total RNA 30 ng을 이용하여 역전사 시간 10분 이하로 단축하여 실험을 진행하였을 때, 측정된 Ct 값은 5분에서 24.38 cycles로 가장 빠르게 측정되었다. Ct 값을 토대로 목적 유전자의 분자 수를 계산한 결과, 10분에서 1.20×104 분자가 검출되었으며, 7분에서 1.07×104 분자, 5분에서 6.03×104 분자, 3분에서 1.51×104 분자, 1분에서 5.01×103 분자, 0분에서 3.55×102 분자로 0분에서 5분까지는 RT시간과 분자 수는 비례하는 것으로 확인하였으며, RT시간 7분과 10분에서 오히려 Ct 값과 검출된 분자 수에서의 감소세를 보였다 (Fig. 4).
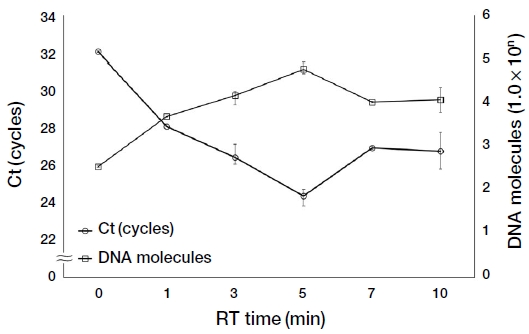
Optimum time of reverse transcription for BQCV detection according to Ct value and molecules of DNA. The optimal reverse transcription (RT) time was determined at 5 minutes for BQCV multi-point URRT-qPCR.
역전사 반응 시간과 합성되는 cDNA의 양은 비례적인 것으로 생각되나, 5분 이상의 역전사 반응은 크게 효과적이지 않으며, 0분의 역전사에서도 cDNA합성이 충분히 이루어지며, PCR을 통한 목적 유전자의 검출이 가능하였으므로, 초고속 PCR에서 역전사 반응을 진행하지 않고도 BQCV의 검출이 가능한 것으로 판단되었다.
제안하는 BQCV 특이 초고속 역전사 실시간 PCR은 기존의 PCR 기법과는 다르게 역전사 시간을 크게 단축할 수 있음을 정량적으로 입증하였으며, 결과적으로 역전사 반응시간을 주지 않았을 때, 검출 한계치인 102 분자 이상의 cDNA를 제작해 낼 수 있음을 확인하였다. 본 연구를 통하여 RT 반응은 역전사 효소를 넣어준 뒤, 반응 시작 후 초기 변성온도인 95℃로 올라가는 시간만으로도 역전사반응이 가능함을 보였다. 또한, RT시간 0분의 조건에서 9분 18초의 BQCV의 검출 시간을 보이며, 가장 빠르게 BQCV 감염을 검출할 수 있는 것으로 확인하였다. 하지만 보다 안정적이고 극미량의 BQCV의 검출을 위해 다량의 BQCV를 검출할 수 있는 5분으로 RT시간을 설정하였다.
현장 적용을 위한 꿀벌 시료를 사용한 확정 시험
BQCV 특이 유전자 초고속 PCR의 유효성을 평가하기 위하여, 경기도 수원, 전라남도 여수, 제주도에서 제공받은 시료를 통해 Total RNA를 추출하였으며, 이를 주형으로 BQCV의 3 point URRT-qPCR을 수행하였다.
BQCV 특이 프라이머 Tm 값의 유효범위는 RdRp의 경우 76.69±1.46℃이며, capsid protein의 경우 78.96±0.81℃이며, 3C like protease의 경우 80.55±0.52℃로 확립되었다. 이를 토대로 실제 양봉가에서 사멸한 꿀벌을 대상으로 BQCV를 검출 및 분석한 결과, 시료 6곳 중 5곳에서 BQCV의 검출이 가능하였다. 시료 1은 전라남도 여수 시료로 VP 부분에서 73.94℃로 유효범위와 4.21℃의 오차를 보이고 있어 비특이적 증폭이라고 판단하였으며, RdRp와 3C like protease 부분에서는 각각의 유효범위에 포함되는 77.82℃, 80.42℃로 나타나 양성반응으로 판정되었다. 또한, 시료 2와 5에서는 RdRp와 VP 부분에서 음성반응을 보였으며, 3C like protease에서는 양성반응을 보였다. 시료 6에서는 모든 부분에서 음성반응을 보여 BQCV에 감염되지 않은 시료로 판정되었다. 시료 3, 4에서는 모든 부분의 양성반응을 보이며 BQCV에 감염되었으며 검출 부위에 변이가 없었던 것으로 사료되었다 (Fig. 5).
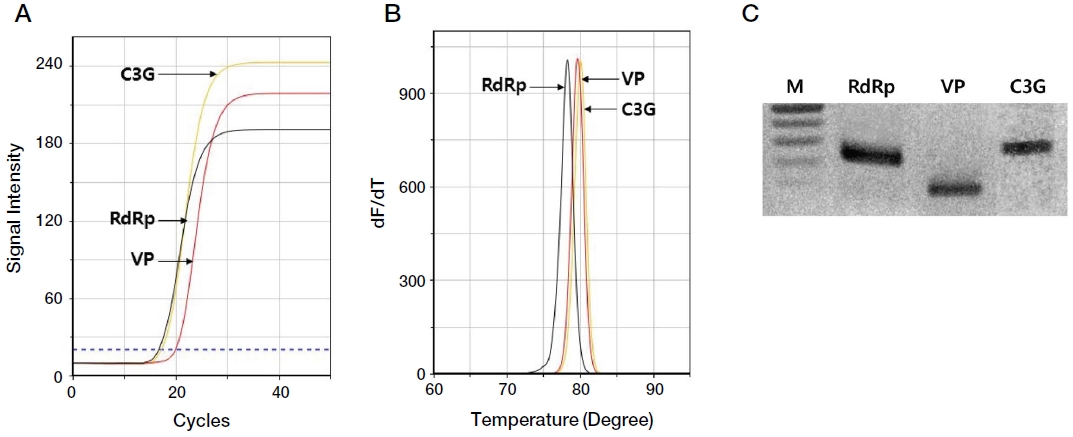
Representative data of multi-point URRT-qPCR for sample 4 from Suwon.Panel A, the fluorescent graph for each BQCV specific gene. Panel B, the value of Tm to amplified PCR product. Panel C, electrophoresis of amplified PCR for confirmation. lane M is the 100 bp DNA size marker (100 bp, 200 bp, 300 bp, 400 bp, 500 bp); lane RdRp, RdRp detection from sample 4 using 3 point-URRT-qPCR; lane VP, Vp detection; lane C3G, 3C like protease detection.
BQCV 특이 유전자 초고속 다중 PCR을 이용한 감염검사 확정 시험에서는 Tm 값을 통해 BQCV에 감염된 개체인지 미감염 개체인지 판단하기에 용이하였으며, 그 차이를 통해 비특이적 증폭인지 특이적 증폭인지 구분이 수월하였다. 시험에 사용한 시료 6곳 중 5곳에서 BQCV의 감염이 확인되었으며, 이를 통해 BQCV의 만연함을 확인할 수 있었다. 또한, BQCV의 서로 다른 ORF 세가지 부분을 검사함으로써 개체별 감염 양상과 BQCV의 유전자 변이에 대한 단서를 얻을 수 있었다.
또한, 실제 BQCV 감염 시료를 이용한 병원체 검출에서는, 한 마리당 3.57×104 분자의 바이러스 분자를 가진 시료 3과 한 마리당 1.55×108 분자에 해당하는 바이러스를 가진 시료 4로부터, cDNA 제작 과정인 역전사 과정부터 검출까지 각각 8분 58초, 5분 22초 만에 BQCV 유무를 감지할 수 있었으며, 시료마다 세 가지 유전자 중 검출되는 부위가 상이하여 BQCV 내부의 유전자 변이 가능성을 의심할 수 있었다 (Table 2).
결 론
본 연구에서 적용한 multi-point PCR 방법은 역전사 시간을 단축한 초고속 역전사 실시간 PCR 법으로 실제 시료 내 BQCV의 감염 여부를 가장 빠르고 정량적으로 검출할 수 있게 정립한 방법이다. 설계된 바이러스 특이 프라이머의 적용은 BQCV의 변이가능성을 의심할 수 있었으며 변이된 BQCV 또한 검출해낼 수 있는 BQCV 확정 검사를 가능하게 하였다. 본 연구에서 개발된 일련의 초고속 역전사 정량 PCR과 세 쌍의 특이 프라이머를 이용한 다중검사법이 다른 RNA 바이러스 병원체의 진단과 변이에 적용 및 발전될 수 있기를 기대한다.
Acknowledgments
본 연구는 2018학년도 농촌진흥청 연구사업 (세부 과제번호: PJ01408002) 지원에 의하여 수행되었으며, 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 농생명기술개발 사업 (과제번호 318093-03)과 수출전략기술개발사업 (과제번호 115067-02)에 의한 연구 결과물임을 밝힙니다.
References
- 김문정, 김정민, 김병희, 김소민, Truong A Tai, 윤병수, (2018), Acute Bee Paralysis Virus (ABPV)의 정확한 검출을 위한 Nested 초고속 PCR법 개발, J. Apiculture, 33, p165-180.
- 김소민, 임수진, 김정민, 임윤규, 윤병수, (2017a), Slow Bee Paralysis Virus (SBPV) 신속 검출을 위한초고속 역전사 중합효소 연쇄반응법의 개발, J. Apiculture, 32, p171-180.
- 김소민, 임수진, 김정민, 김병희, Truong A Tai, 윤병수, (2017b), 초고속 유전자 증폭법을 이용한 서양뒤영벌 의심병원체 Lysinibacillus fusiformis의 신속 검출법, J. Apiculture, 32, p181-189.
- 김정민, 임수진, Truong A Tai, 홍기정, 윤병수, (2017c), 초고속 유전자 증폭법을 이용한 벌집꼬마밑빠진벌레 (Aethina tumida)의 신속 검출 기법 개발, J. Apiculture, 32, p119-131.
- 왕지희, 이도부, 구수진, 백문철, 민상현, 임수진, 이칠우, 윤병수, (2016), 다중 PCR 증폭과 특이 DNA-chip을 활용한 꿀벌 주요 11종 병원체의 검출법 개발, J. Apiculture, 31, p133-146.
- 유미선, 김일욱, 강민희, 한상훈, 윤병수, (2008), Black Queen Cell Virus 진단을 위한 Real-Time PCR 진단법의 개발, J. Apiculture, 23, p37-42.
- 유미선, 이동우, 김일욱, 김동수, 권순환, 임윤규, 윤병수, (2007), 한국산 꿀벌에서 Black Queen Cell Virus의 검출, J. Apiculture, 22, p43-52.
- 임수진, 김정민, 이칠우, 윤병수, (2017), 꿀벌 6종 주요 병원체에 대한 초고속 다중 PCR 검출법의 개발, J. Apiculture, 32, p27-39.
-
Dalmon, A., C. Desbiez, M. coulon, M. Thomasson, Y. Le Conte, C. Alaux, J. Vallon, and B. Moury, (2017), Evidence for positive selection and recombination hotspots in Deformed wing virus (DWV), Sci. Rep., 7, 41045.
[https://doi.org/10.1038/srep41045]

-
Han, S. H., D. B. Lee, E. H. Kim, and B. S. Yoon, (2008), Ultra-rapid real-time PCR for the detection of Paenibacillus larvae, the causative agent of American Foulbrood (AFB), J. Invertebr. Pathol., 99, p8-13.
[https://doi.org/10.1016/j.jip.2008.04.010]

- Innis, M. A., D. H. Gelfand, J. J. Sninsky, and T. J. White, (1990), PCR Protocols: A Guide to Methods and Applications, ACADEMIC PRESS, INC, p131.
-
Tapaszti, Z., P. Forgach, C. Kovago, G. Topolska, N. Nowotny, M. Rusvai, and T. Bakonyi, (2009), Genetic analysis and phylogenetic comparison of Black queen cell virus genotypes, VETMIC, 139, p227-234.
[https://doi.org/10.1016/j.vetmic.2009.06.002]

-
Wang, H., J. Xie, T. G. Shreeve, J. Ma, D. W. Pallett, L. A. King, and R. D. Possee, (2013), Sequence Recombination and Conservation of Varroa desturctor Virus-1 and Deformed Wing Virus in Field Collected Honey Bees (Apis mellifera), PLoS One, 8, p1-9.
[https://doi.org/10.1371/journal.pone.0074508]