고추의 잔류 살충제 디노테퓨란 노출 후 꿀벌의 전사체 발현 패턴 분석
Abstract
Honey bees (Apis mellifera L.), recognized as vital pollinators in agriculture, are adversely effected by pesticides used to protect crops from various pests. Many farmers use neonicotinoids due to their reputed low toxicity to honey bees. To investigate the mechanisms underlying this low toxicity, the gene expression of worker bees was analyzed using RNA sequencing (RNA-seq). Approximately 10 GB of sequences were generated from each samples. Out of 250,928 genes analyzed, 3,017 genes were identified as differentially expressed genes (DEGs) with 1,516 up-regulated and 1,501 down-regulated. Among these DEGs, genes such as glutamate receptor, peroxiredoxin, multicopper oxidase, cytochrome P450 6a13, and acetylcholinesterase were revealed as potentially contributing to pesticide resistance in honey bees. This study provides significant insights into the genetic mechanisms underlying the effects of dinotefuran, a neonicotinoid insecticide, on honey bees.
Keywords:
Apis mellifera, Residue, Dinotefuran, Insecticide, RNA sequencing서 론
꿀벌 (Apis mellifera L.)은 농업에서 중요한 화분매개체 곤충으로써 전 세계적으로 가장 널리 이용되고 있다 (Ollerton et al., 2011; Sung et al., 2023). 이러한 꿀벌은 세계 식량의 90%를 차지하는 124개 경제 작물 가운데 70% 작물의 수분에 관여하며 커피 및 코코아와 같은 고부가가치 작물의 수분에 75%를 기여한다 (Klein et al., 2007; Gallai et al., 2009). 또한, 꿀벌에 의한 화분매개는 작물의 수확량과 품질에 영향을 미친다 (Giannini et al., 2015). 사과에 있어서 꿀벌에 의해 높은 화분매개율을 보인 과원에서는 사과 수확량과 당분의 함량이 각각 15%와 70% 이상 증가하였다 (Gallai et al., 2009). 전 세계적으로 꿀벌에 의한 화분매개의 경제적 기여 가치는 약 145억 달러에 달하며 유럽과 아시아지역에서 매년 2~5천억 달러의 경제적 가치를 가진다고 보고되었다 (Tirado et al., 2013; Potts et al., 2016). 따라서 꿀벌 밀도의 감소는 농산물의 감소와 이를 통한 식량안보에도 직접적인 영향을 미칠 수 있다.
농산물의 안정적인 생산증대를 위해서 농약의 사용은 필연적이다. 현재까지 세계적으로 다양한 종류의 농약이 작물의 생산성을 보장하기 위해 개발되고 판매되고 있다 (Hwang et al., 2020). 이 가운데 해충방제에 많이 활용되고 있는 네오니코티노이드 (Neonicotinoid)계 농약의 살충제는 니코틴과 유사한 신경 활성 살충제의 한 부류로써 1991년 Bayer사에 의해 최초로 계발된 침투이행성 살충제이다 (Hwang et al., 2020). 네오니코티노이드계 살충제는 곤충의 중추신경계에 nicotinic acetylcholine receptor (nAChR)를 표적으로 선택적 길항제로 작용하여 흥분을 전달하는 glutamate (Glu)와 억제자의 역할을 하는 γ-aminobutyric acid의 합성을 교란하여 신경전달을 저해함으로써 살충효과를 나타내며, 농산물에 잔류 가능성이 높은 농약이다 (Kim et al., 2014; Kim et al., 2015; Ihara and Matsuda, 2018).
2006년부터 2009년까지 미국에서는 30~90%에 달하는 꿀벌의 대규모 실종 사태가 발생하였다 (Mullin et al., 2010). 이러한 꿀벌집단의 급격한 밀도 감소는 꿀벌 군집 붕괴 (Colony Collapse Disorder, CCD)의 원인이 되고 있으며, CCD는 여왕과 유충의 감소 없이 성충일벌만 급격히 사라지는 것으로 특정된다 (vanEngelsdorp et al., 2007). 살충제는 오랫동안 꿀벌 감소의 잠재적인 원인으로 의심을 받아왔으며, 이 중 네오니코티노이드와 같이 저독성으로 개발된 일부 농약의 경우 꿀벌과 같은 비표적 곤충의 생태에 영향을 미쳐 피해를 주는 것으로 보고되었다 (Boncristiani et al., 2012; Dulin et al., 2014; Glavan et al., 2020). 네오니코티노이드계 농약은 꿀벌들의 집단 폐사와 방향 능력 상실에 영향을 주어 꿀벌 개체 수 감소의 잠재적인 원인으로 명확히 지적되어 전 세계적으로도 네오니코티노이드계 성분이 포함된 살충제에 대한 경각심이 높아졌다 (Lundin et al., 2015). 우리나라에서도 네오니코티노이드계 살충제가 벼와 과수 농사 등에 광범위하게 사용되면서 이들 농약의 잔류에 의한 농산물 안전성에 대한 우려가 제기되었다 (Hwang et al., 2020). 유럽연합에서는 클로티아니딘, 티아메톡삼 및 이미다클로프리드 등 3종의 네오니코티노이드계 농약의 사용이 금지되었으나, 동일한 계열의 다른 농약은 여전히 작물 보호를 위해 널리 사용되고 있다 (Simondelso et al., 2015). 꿀벌은 농약이 포함된 꽃가루와 꿀을 통해 농약에 노출될 수 있으며, 실제로 네오니코티노이드계 농약은 꽃가루와 꿀에서 높은 농도로 검출되어 꿀벌에게도 해로운 영향을 미칠 수 있다 (Krupke et al., 2012; Saleem et al., 2020). 비록 꿀벌은 해독효소인 cytochome oxidase P450 (CYP450)의 해독작용으로 네오니코티노이드계 살충제에 대해 저독성을 보여주고 있지만 (Matsumoto, 2013; Tsvetkov et al., 2023), 이 계열의 살충제에 만성적으로 노출된 꿀벌은 생리·생태적 기능이 교란되어 뇌와 중장 발육뿐만 아니라 학습, 먹이활동 및 귀소 능력 등 행동적으로도 영향을 받는다 (Peng and Yang, 2016; Li et al., 2017; Shi et al., 2018).
디노테퓨란 살충제는 광범위하고 체계적인 살충효과로 곤충 뇌의 nAChR에 강하게 결합하여 신경독성을 유발하는 3세대 네오니코티노이드계의 농약으로 알려져 있다 (Goulson, 2013). 따라서, 노지에서 네오니코티노이드계 디노테퓨란 살충제 살포가 비표적 곤충인 꿀벌에 미치는 영향성 평가와 상관관계 분석이 필요하다. 본 연구에서는 6월부터 7월 사이에 디노테퓨란 살충제가 살포된 경북 영양의 고추밭 주변에서 화분매개 활동을 하는 꿀벌집단과 동일한 약제에 노출되지 않은 경북 안동의 무처리 집단 간의 전사체 발현 수준을 비교 분석하였다.
재료 및 방법
1. 전사체 분석용 꿀벌 시료 채집
네오니코티노이드계 농약의 한 종류인 디노테퓨란 살충제에 노출된 꿀벌 (‘DINO’) 시료를 채집하기 위해서 2023년 6월 23일에 경북 영양의 고추밭 (36.7422N, 129.1872E)에 벌통을 설치한 후 당일부터 7월 28일까지 농약 (주성분 함량 10%)을 10 a 기준 1,000배 희석하여 일주일 간격으로 5회에 걸쳐 경엽처리하였다. 이후 1주일 뒤인 8월 4일에 벌통을 수거하여 외역 활동을 하는 꿀벌을 5마리씩 3반복으로 시료를 채집하였다. 무처리구인 디노테퓨란 농약에 노출되지 않은 꿀벌 (‘CON’)은 경북 안동의 유기농 고추 재배지 반경 5 km 이내에 있는 양봉장 (36.61875N, 28.8271E)에서 5마리씩 3반복으로 채집하여 사용하였다. 고추 재배지와 시험에 사용한 디노테퓨란 입상수화제는 판매상에서 구매하여 사용하였다. RNA를 추출하기까지 채집한 각 꿀벌 시료는 -80℃의 초저온 냉동고에 시료를 보관하였다.
2. RNA 추출 및 cDNA 라이브러리 제작
꿀벌의 mRNA는 3마리를 1.5 mL 튜브에 1 mL의 Trizol 용액 (Thermo scientiÀc, Massachusetts, USA)을 넣고 1회용 멸균 pestle을 이용하여 마쇄한 후 Trizol RNA 추출 키트 (Thermo Fisher Scientific, Massachusetts, USA)를 이용하여 추출하였다. RNA 시료는 3반복으로 추출하였으며, 추출된 mRNA의 펠렛은 DEPC가 처리된 증류수 100 μL에 녹여서 -80℃에 실험에 사용되기 전까지 보관되었다. 추출된 mRNA의 일부는 전사체 분석을 위해 Random primer를 이용한 TruSeq Stranded Total RNA Ribo-Zero H/M/R Gold (Illumina, California, USA)를 이용하여 cDNA 합성 후 cDNA 라이브러리를 제작하였다. 이후 qRT-PCR을 수행하기 위해 cDNA 라이브러리를 대상으로 Oligo dT primer가 포함된 Maxim RT premix kit (Intron, Seongnam, Korea)를 이용하여 cDNA를 합성하였다. 전사체 분석은 3반복으로 진행하였다.
3. 전사체 및 생물정보학적 분석
mRNA는 Novaseq (Illumina, California, USA)를 이용하여 염기서열 분석을 진행하였다. 분석된 염기서열은 Trimmomatic (version 0.38)을 이용하여 염기서열의 어댑터 부분을 편집하였으며, RNA 염기서열은 Trinity (Version trinityseq_r20140717, bowtie 1.1.2)를 이용하여 de novo 방식으로 조립하였다. RSEM (Version v1.3.1, bowtie 1.1.2.)을 이용하여 염기서열을 de novo 방식으로 조립된 염기서열을 mapping하여 RNA의 발현량을 확인하였다. RNA 염기서열은 Gene Ontology knowledgebase (http://geneontology.org/)와 BLAST v2.15.0+ (https://blast.ncbi.nlm.nih.gov/Blast.cgi)를 통해 확보한 데이터베이스를 이용하여 DIAMOND v0.9.21 (https://github.com/bbuchfink/diamond/releases?after=v0.9.21) 분석으로 유전자를 동정하였다. 이렇게 mapping된 데이터는 R의 edgeR 라이브러리를 이용하여 차등 발현 유전자 분석 (Differentially Expressed Genes, DEGs)을 진행하였다. DEG 분석을 위해서는 Fragmented per Kilobase of transcript per Million mapped reads (FPKM) 값을 이용하였다.
4. qRT-PCR
유전자 발현량 분석을 위한 qRT-PCR은 살충제 대사 및 꿀벌 생리에 관여하는 5개 유전자를 대상으로 특이적 프라이머 (Table 1)를 사용하여 ExicyclerTM Quantitative Thermal Block (Bioneer, Daejeon, Korea)을 이용하여 SYBR 그린 형광물질과 형광분석방법을 가미한 real time quantitative PCR (qRT-PCR) 기술로 측정하였다. qRT-PCR 반응은 Accupower GreenstarTM PCR premix (Bioneer, Daejeon, Korea)로 진행하였다. 이 반응물 (20 μL)은 1× GreenstarTM master mix, 10 mM MgCl2, 0.5 μM의 각 프라이머, 200 ng의 cDNA가 포함되었다. 반응조건은 Hotstart Taq DNA polymerase를 활성시키기 위해 95℃에서 15분간 가열하였으며, 이후 94℃에서 30초, 55℃에서 30초, 72℃에서 30초의 반응 순으로 40회 PCR 산물을 증폭시켰고, 증폭된 산물의 형광반응이 실시간으로 측정되었다. 각 처리는 3반복으로 각 반복은 독립된 반응으로 수행되었다. Reference 유전자는 Ribosomal protein L32 (RL32)를 이용하였다 (Jin et al., 2024). 이 유전자 증폭물을 각 유전자 반응의 정량적 표준화 대조구로 이용하였다. 얻어진 PCR 증폭물의 형광량을 ExicyclerTM 프로그램에 의해 실시간으로 도식화하였고, 정량적 임계 반응 횟수 (Critical Threshold, Ct)를 산출하였다. 다시 각 유전자의 Ct는 RL32 유전자 Ct에 대한 차이 (ΔCt)로 산출한 후, 이를 상대적 수치 (2-ΔΔCt)로 환산하여 비교하였다 (Livak and Schmittgen, 2001).
5. 디노테퓨란 표면처리를 통한 전사체 분석 결과 검정
전사체 분석 결과에 대한 유전자의 발현량 검정을 위해 국립안동대학교 양봉장에서 사육 중인 우화 후 3주가 지난 일벌을 대상으로 유전자 발현량을 조사하였다. 농약처리를 위해 0.01%의 Triton-X100 (Thermo Scientific, Massachusetts, USA)가 첨가된 물에 노지에 살포된 디노테퓨란과 동일한 농도인 1 ng/mL를 녹여서 사용했다. 약제 처리는 우화 후 15일 경과된 꿀벌을 대상으로 5마리씩 3반복으로 진행하였으며, 5 μL의 약액을 꿀벌의 등면 (dorsal)에 면봉으로 골고루 표면처리를 하였다. 그 뒤 꿀벌은 28℃의 인큐베이터에서 50%의 설탕물로 3일간 사육한 후 mRNA를 추출하였다.
6. 통계처리
그래프의 통계값은 평균±표준편차를 이용하여 나타내었다. 결과는 Graphpad prism 9 (Dotmatics, Boston, USA)을 사용하여 도표화하였다. 평균은 SAS 프로그램 (SAS Institute Inc, 1989)을 이용하여 one-way ANOVA 분석을 하였다. 처리 평균 간 비교는 LSD 검정을 사용하여 제1형 오류 확률 0.05를 기준으로 판별하였다.
결 과
1. RNA-seq 결과 분석
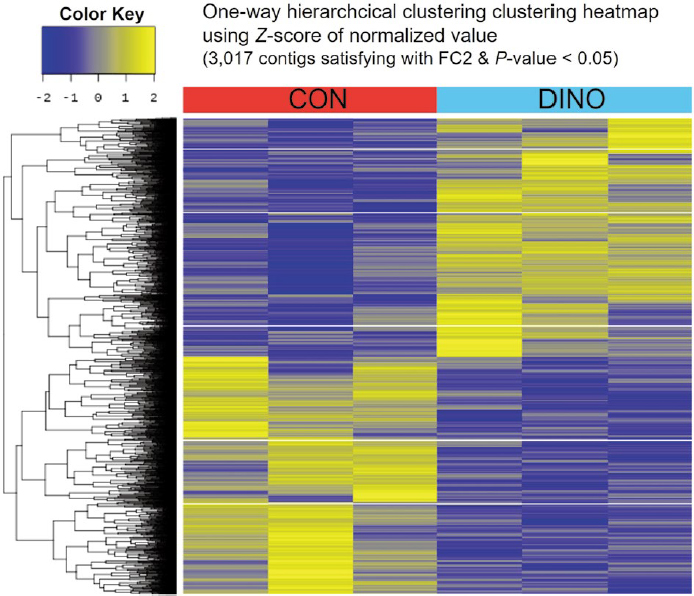
Novaseq 플랫폼을 통해 대략 10 GB 정도의 염기서열을 분석하였다. 각 염기서열을 101 bp의 크기로 편집한 뒤 100 KB 정도 크기의 염기서열 조각들을 얻었다. De novo 방식으로 mapping을 수행한 결과, CON과 DINO는 각각 75.00~82.12%와 75.73~83.57%의 염기서열이 매칭되었다 (Table 2). 총 250,928개의 유전자가 발굴되었으며, CON과 DINO의 유전자 차등 발현량은 Z-score로 변환 후 P-value를 이용하여 heatmap 분석을 통해 도출 결과를 시각화하였다 (Fig. 1). 또한 생산된 전사체 유전정보는 NCBI의 BioProject (https://www.ncbi.nlm.nih.gov/bioproject, accession number PRJNA1135696)에 등록하였다.

RNA-seq results of Apis mellifera workers, comparing dinotefuran-exposed (‘DINO’) and non-exposed (‘CON’) groups
2. 디노테퓨란 노출에 따른 꿀벌의 차등 발현 유전자 (DEG) 분석
DEG 결과를 바탕으로 유전자 발현이 2배 이상 차이가 나고 P-value가 0.05 미만인 유전자를 분류하기 위해 Volcano plot 분석을 수행하였다 (Fig. 2A). DINO는 CON에 비해 1,516개의 유전자에서 발현량이 2배 이상 증가하였으며, 1,501개의 유전자에서 발현량이 2배 이상 감소하였다 (Fig. 2B). Gene Ontology 분석 결과, biological process, cellular component, molecular process 부분을 분석하였다 (Fig. 2C). 세부적인 GO 분석 결과, biological process 부분에서 cellular process 그룹이 488개의 유전자로 가장 많이 검출되었으며, cellular components 부분에서 cell part 그룹이 471개의 유전자로 가장 많이 검출되었다. 그리고 molecular process 부분에서는 catalytic activity 그룹에서 512개의 유전자로 가장 많이 검출되었다.
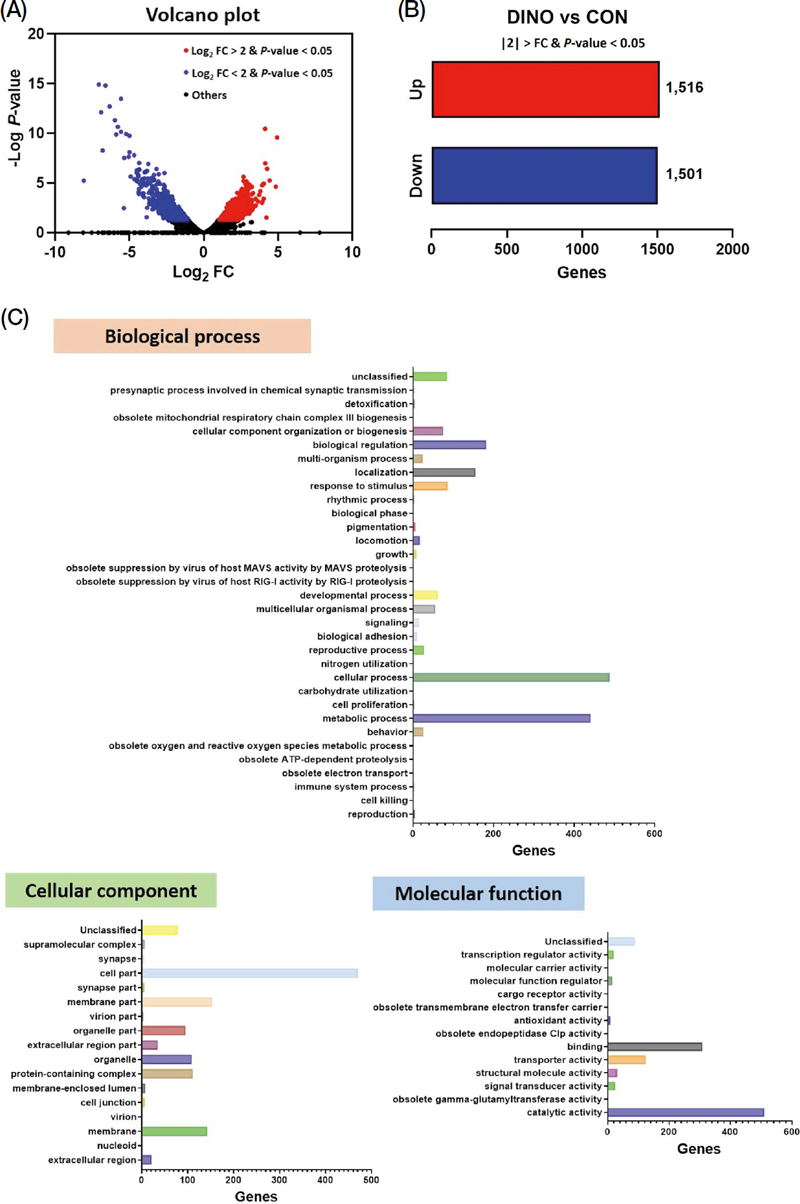
Analysis of differentially expressed genes (DEGs) in Apis mellifera after indirect exposure to dinotefuran. (A) Volcano plot comparing dinotefuran non-exposed (‘CON’) and exposed (‘DINO’) honey bees. Fold-change (FC) values and -Log P-values were used to generate the volcano plot. (B) DEGs between CON and DINO, selected with threshold levels of >2 FC and P-value <0.05 in Fragments per Kilobase of transcript per Million mapped reads (FPKM) values. (C) Gene Ontology (GO) analysis of DEGs performed for cellular component, molecular process, and biological process.
DEG 분석에서 선발된 유전자 중, biological process에 관여하는 유전자 그룹에서 obsolete oxygen and reactive oxygen species metabolic process 그룹에서 3개, signaling 그룹에서 12개, detoxification 그룹에서 4개 유전자를 선별하여 heatmap 분석을 통해 발현량을 확인하였다 (Fig. 3A). Cellular components에 해당하는 유전자 그룹 중, presynaptic process involved in chemical synaptic transmission 그룹에서 6개의 유전자가 관여하였으며, synapse 그룹에서 5개에 해당하는 유전자들을 heatmap 분석을 통해 발현량을 확인하였다 (Fig. 3B, Table 3).
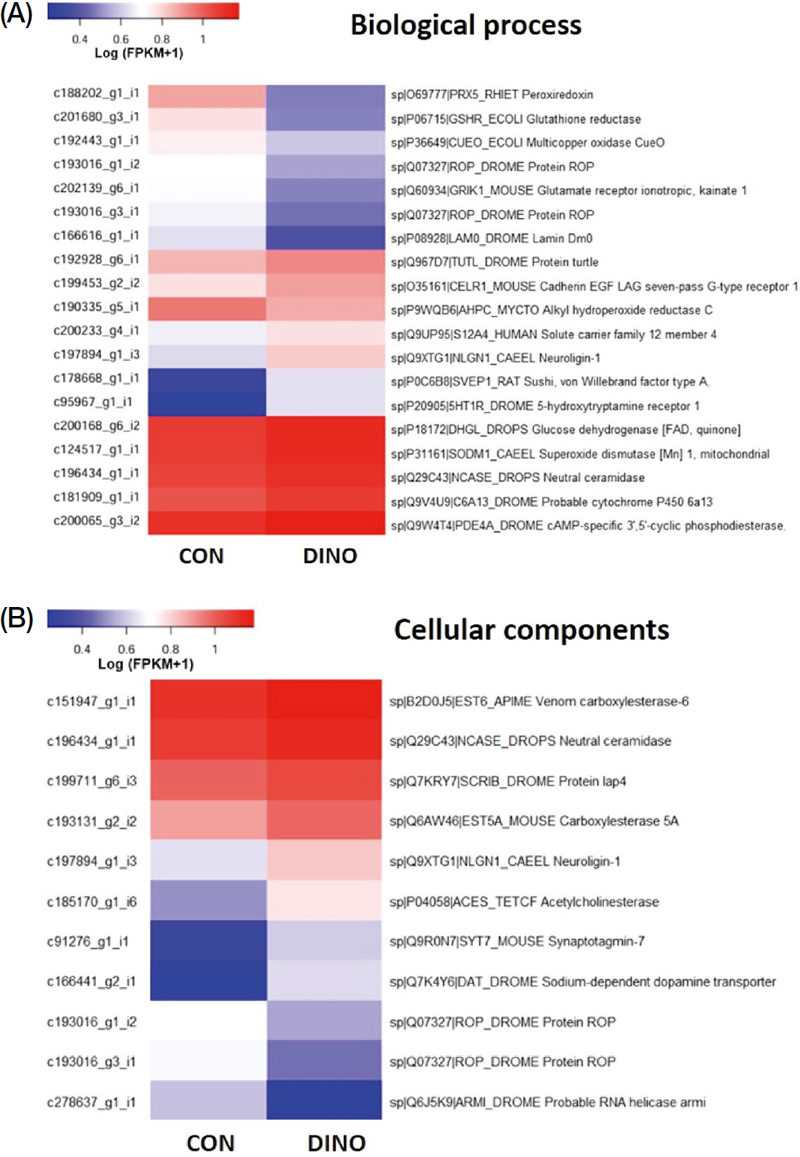
Heatmap analysis of pesticide resistance genes between dinotefuran non-exposed (‘CON’) and exposed (‘DINO’) honey bees from Gene Ontology (GO) analysis. (A) Biological process and (B) Cellular components heatmap. The heatmap was generated using log Fragmented per Kilobase of transcript per Million mapped reads (FPKM) values. The genes were selected from Table 2. The heatmap was created using the R program (version 4.2.1) from the heatmap library.
3. 살충제 저항성 관련 유전자 발현 분석
살충제 저항성과 관련하여 체내에 전달된 약물을 수용성으로 변환하는 변형단계 (phase I) 그룹의 유전자 가운데 cytochrome P450 (CYP450), carboxylesterase (CES) 및 hydrolase 유전자가 각각 6개, 2개 및 5개가 발견되었으며 모두 과발현되었다. Phase I에서 생성된 살충제 유도체를 수용성으로 변환하는 포합단계 (phase II) 그룹의 유전자는 sulfotransferase가 유일하게 발견되었으나, 발현이 억제되었다. 마지막으로 phase I과 phase II 단계에서 변형된 살충제를 세포 밖으로 배출하는 과정의 배출단계 (phase III) 그룹의 유전자는 ATP-binding cassette transporter가 발견되었으나, 발현은 억제된 것으로 DEG 분석 결과 확인되었다 (Table 4).
4. 꿀벌 체내의 살충제 대사 및 생리 관련 유전자 발현 분석
DEG 분석에서 발현량의 차이를 보이는 살충제 대사와 꿀벌 생리에 관여하는 5개 유전자가 관찰되었다 (Fig. 4). 디노테퓨란 처리에 따른 CON에 대비 DINO의 유전자 발현의 차이는 peroxiredoxin (Prx)의 경우 25배 감소, glutamate receptor inonotropic, kinate 1 (iGluR)의 경우 약 4배 정도 감소하였고, multicopper oxidase (CueO)의 경우 4배 감소하였다. 이와는 반대로 cytochrome P450의 경우 CYP6a13, CYP304a1, CYP4c1, CYP4g15, CYP315a1 및 CYP9e2 유전자가 각각 3배, 5배, 3배, 4배, 3배 및 8배 이상 증가하였고, acetylcholinesterase (AChE)의 경우 약 5배 정도의 유전자 발현이 증가하였다 (Table 4).
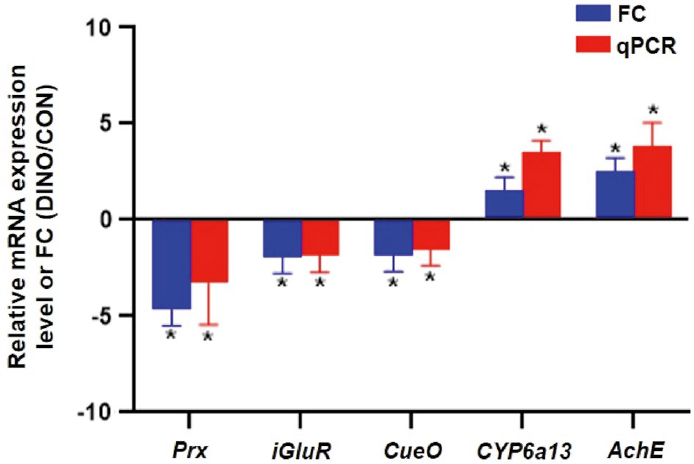
Validation of differentially expressed genes (DEGs) associated with pesticide resistance in Apis mellifera by qRT-PCR, comparing fold change (FC) data from RNA-Seq. Gene expression was compared between dinotefuran-exposed (‘DINO’) and non-exposed (‘CON’) honey bees. Relative expression level of each DEG is presented as FC and relative gene expression is shown as ratio of them per reference gene, RL32. Five genes were analyzed in this study: Peroxiredoxin (Prx), Glutamate receptor ionotropic, kainate 1 (iGlueR), Multicopper oxidase (CueO), Cytochrome P450 6a13 (CYP450), and Acetylcholinesterase (AChE). Each sample was analyzed in triplicate.
본 연구에서는 흥미롭게도 나비목 곤충의 인지질 생합성에 관여하는 phospholipase B1 유전자의 발현이 디노테퓨란에 노출된 꿀벌에서 30배 이상 발현이 증가하여 가장 높았으며, 초파리 수컷의 정자형성에 필요한 armadillo repeat-containing protein의 생합성에 관여하는 gudu 유전자의 발현은 100배 이상 감소하였다 (Table 5).
5. 디노테퓨란 in vivo 표면처리를 통한 전사체 분석 결과 검정
DEG 분석에서 FPKM 값을 기준으로 농약 저항성 관련 무처리구 대비 유전자의 발현량이 차이가 있었던 처리구의 Prx, iGluR, CueO, CYP450 및 AChE를 qRT-PCR을 이용하여 검정하였다. 그 결과 디노테퓨란 노출 후 발현량이 감소한 3개 유전자 (Prx, iGluR 및 CueO)와 증가한 2개 유전자 (CYP6a13 및 AChE)에서 DEG 분석에 이용한 FPKM 값과 qRT-PCR의 정량적 임계 반응 횟수 모두 P-value가 0.05 이하의 값에서 서로 동일하여 본 연구에 사용한 DEG 분석값을 신뢰할 수 있었다 (Fig. 4).
고 찰
디노테퓨란 살충제는 곤충의 뇌에서 니코틴성 아세틸콜린 수용체 (nAChRs)에 강하게 결합하여 신경독성을 나타내는 3세대 네오니코티노이드 살충제로 광범위한 해충에 대해 살충 활성을 가지고 있다. 디노테퓨란의 꿀벌에 대한 영향은 갓 우화한 성충일벌을 대상으로 0.773 mg/L (lethal concentration 50, LC50)와 0.465 mg/L (LC10)로 조사되어 급성독성을 보여주어, 비표적 곤충인 꿀벌에 대한 독성효과가 확인되었다 (Huang et al., 2021). 특히, RNA-Seq 분석을 통해 CON과 DINO를 대상으로 총 250,928개의 유전자 발굴과 DEG 분석을 통해 발현량에 변화가 있는 유전자 3,017개 (과발현 1,516개, 억제발현 1,501개)를 발견하였다 (Figs. 1, 2B).
본 연구에 이용한 DEG 분석을 통해 살충제 대사 및 생리 관련 유전자 5개 (Prx, giGluR, CueO, CYP6a13 및 AChE)를 대상으로 디노테퓨란 간접 노출에 따른 유의미한 차이를 발견할 수 있었다. 특히, 디노테퓨란 노출 후 CYP6a13와 AChE 유전자의 발현량은 크게 증가한 반면, Prx, iGluR 및 CueO 유전자의 발현량이 감소한 것을 확인하였다 (Fig. 4).
꿀벌이 네오니코티노이드계 농약에 대해서 저항성이 높은 이유는 CYP450과 같은 해독효소가 다양한 기질을 대상으로 산화반응을 일으켜 농약을 분해할 수 있기 때문이다 (Mclntosh et al., 2014; Tsvetkov et al., 2023). 본 연구의 전사체 분석 결과에서 디노테퓨란에 노출된 꿀벌에서 무처리구와 비교하여 CYP6a13, CYP4c1 및 CYP4g15 유전자의 발현량이 많이 증가한 것을 확인할 수 있었으며 (Fig. 4, Table 4), 이는 꿀벌 체내에서 네오니코티노이드계 살충제를 분해하기 위해 유전자 수준에서 과발현된 것으로 판단된다. 이와 같은 결과는 동일한 네오니코티노이드 계열의 클로티아니딘 (clothianidin) 농약에 노출된 딱정벌레목의 콜로라도잎벌레 (Leptinotarsa decemlineata)에서도 CYP6a13 및 CYP4c1, CYP4g15 등의 유전자 발현량의 차이가 보고되어 본 연구 결과와 동일하였다 (Bouafoura et al., 2022). 특히, CYP450 유전자는 곤충의 농약 저항성 (Claudianos et al., 2006; Zhao et al., 2020; Vlogiannitis et al., 2021)뿐만 아니라 곤충의 면역반응 (Vatanparast et al., 2020)과 큐티클 형성 (Wu et al., 2020)에도 관여하여 생리적으로 매우 중요한 유전자이다.
네오니코티노이드계 농약은 nAChR에 직접적으로 작용하며 Glu 합성을 유도하여 흥분상태를 유지하게 해서 살충작용을 일으키는 것으로 알려져 있다 (Willard and Koochekpour, 2013). 본 연구에서 디노테퓨란에 간접 노출 후 AChE 유전자의 발현은 크게 증가했지만, iGluR 유전자의 발현은 반대로 감소하였다. 이는 높아진 Glu의 농도에 대하여 GluR의 발현량을 조절하여 자극의 전달을 완화하여 항상성을 유지하는 것으로 보인다. 이와 같은 현상은 꿀벌과 같은 벌목에 속하는 호박벌을 대상으로 네오니코티노이드계의 농약을 섭식 시, AChE의 발현량이 증가되어 살충제에 대한 저항성을 나타내는 것과 동일하였다 (Samson-Robert et al., 2015).
CueO의 경우에는 거짓쌀도독거저리 (Tribolium castaneum)에서 수명과 생식 등에 관여하는 유전자로, RNA 간섭 (RNA interference)을 통해 유전자 발현을 일시적으로 억제할 경우, 산란 수의 감소와 수명이 줄어든다 (Peng et al., 2014). 따라서, 본 연구 결과에서 확인된 CueO 유전자의 발현량 감소는 디노테퓨란에 장기적으로 노출된 꿀벌집단의 수명 감소와 일벌들이 수집한 화분이 여왕벌에게 전달될 경우 이를 섭식한 여왕벌의 산란율 저하에 따른 잠재적인 꿀벌집단의 붕괴에도 관여할 수 있을 것으로 판단된다.
Prx 유전자는 활성산소종의 증가에 반응하는 산화스트레스 관련 유전자로 알려져 있다. 활성산소 축적에 의한 피해는 특정 질병이나 노화와 관련되어 있다고 연구되어 있다 (Gertz et al., 2009). 그러나 Ramirez et al. (2017)에 따르면 25℃ 이하의 온도에 단기적으로 노출된 꿀벌 유충의 경우 사망률이 40% 이상 증가하며, 이에 대응하기 위해 산화 및 열충격 반응과 관련한 유전자의 발현이 유도되어 산화스트레스와의 연관성을 보고하고 있다. 또한, 파리목의 초파리 (Drosophila melanogaster)의 경우 낮은 온도에서 성충이 생활할 때 산화 손상을 방지하거나 수리하기 위한 과정에서 Prx와 같은 관련한 대사 경로들이 전사체와 대사체 분석을 통해 확인되어 온도와 산화스트레스간 상관관계가 보고되었다 (MacMillan et al., 2016). 꿀벌 유충의 경우 저온 스트레스에 노출 시 Prx1과 Prx4 유전자의 발현이 높아져 저온에서의 생존을 유리하게 한다 (Mucci et al., 2021). 본 연구에서는 Prx 유전자의 발현이 디노테퓨란의 간접 노출 후 성충에서 억제되는 것을 확인할 수 있었다. 따라서, 디노테퓨란에 장기간 노출된 꿀벌은 유충에서 성충에 이르기까지 전체적으로 저온 스트레스에 대한 저항성을 낮춰 월동과 같은 저온기에 생존율에 영향을 줄 수 있을 것으로 사료된다.
본 연구에서는 전사체 발현 수준에서 디노테퓨란 살충제에 간접 노출된 꿀벌의 경우 유전자 발현량에 있어서 영향을 받는다는 것을 확인하였다. 네오니코티노이드계 디노테퓨란 살충제에 의한 꿀벌의 간접 노출 영향을 전사체와 DEG를 바탕으로 분석한 결과, 노출된 꿀벌은 살충제 대사와 관련한 일부 유전자의 과발현을 나타내어 전형적인 살충제 노출 시 나타나는 현상과 동일하였다. 이번 연구 결과에서 흥미로운 점은 저온 노출로 인한 활성산소종의 분해를 위한 유전자인 Prx의 발현을 억제하여 저온 생리를 교란할 수 있는 점이다. Prx 유전자의 구체적인 저온 관련 기능을 구명하기 위해서는 디노테퓨란 노출 기간에 따른 발육시기별 유전자의 발현량 조사, 저온 생존율과 기억 능력 및 이에 따른 귀소 능력의 행동학적인 변화에 관한 추가 연구가 향후 필요하다. 이를 통해 디노테퓨란 살충제에 대한 꿀벌의 영향과 최근 문제가 되는 CCD 현상의 원인을 유전자 수준에서 규명할 수 있을 것이다.
적 요
네오니코티노이드계 디노테퓨란 살충제에 대한 꿀벌의 영향을 분석하기 위해 이 약제가 살포된 고추밭에서 꿀벌집단을 수집하여 무처리 집단과 유전자 발현 차이를 RNA-seq로 분석하였다. 분석된 250,928개의 유전자 중 3,017개의 유전자가 차등 발현 유전자로 확인되었고, 이 중 1,516개는 발현이 증가하고 1,501개는 발현이 감소하였다. 이러한 차등 발현 유전자 가운데 꿀벌의 살충제 저항성에 관여하는 유전자 (glutamate receptor, peroxiredoxin, multicopper oxidase, cytochrome P450 및 acetylcholinesterase)가 확인되었다. 본 연구의 결과는 디노테퓨란에 간접적으로 노출된 양봉꿀벌의 변화를 유전자 수준에서 관찰할 수 있어 네오니코티노이드계 살충제의 꿀벌 위해성 평가를 위한 기초 생물 정보로 활용할 수 있을 것으로 사료된다.
Acknowledgments
본 연구수행에 필요한 꿀벌 시료를 확보하는 데 도움을 준 국립안동대학교 정철의 교수님과 오현하 대학원생에게 감사드린다. 본 연구는 2023학년도 국립안동대학교 학술 연구조성비에 의하여 수행되었다.
References
-
Boncristiani, H., R. Underwood, R. Schwarz, J. D. Evans, J. Pettis and D. vanEngelsdorp. 2012. Direct effect of acaricides on pathogen loads and gene expression levels in honey bees Apis mellifera. J. Insect Physiol. 58: 613-620.
[https://doi.org/10.1016/j.jinsphys.2011.12.011]

-
Bouafoura, R., P. Bastarache, B. C. Ouédraogo, P. Dumas, C. E. Moffat, J. L. Vickruck and P. Jr. Morin. 2022. Characterization of insecticide response-associated transcripts in the Colorado potato beetle: Relevance of selected cytochrome P450s and clothianidin. Insects 13: 505.
[https://doi.org/10.3390/insects13060505]

-
Claudianos, C., H. Ranson, R. M. Johnson, S. Biswas, M. A. Schuler, M. R. Berenbaum, R. Feyereisen and J. G. Oakeshott. 2006. A deficit of detoxification enzymes: Pesticide sensitivity and environmental response in the honeybee. Insect Mol. Biol. 15: 615-636.
[https://doi.org/10.1111/j.1365-2583.2006.00672.x]

-
Dulin, F., C. Zatylny-Gaudin, C. Ballandonne, B. Guillet, R. Bonafos, R. Bureau and M. P. Halm. 2014. Protecting honey bees: identification of a new varroacide by in silico, in vitro, and in vivo studies. Parasitol. Res. 113: 4601-4610.
[https://doi.org/10.1007/s00436-014-4150-z]

-
Gallai, N., J. M. Salles, J. Settele and B. E. Vaissière. 2009. Economic valuation of the vulnerability of world agriculture confronted with pollinator decline. Ecol. Econ. 68: 810-821.
[https://doi.org/10.1016/j.ecolecon.2008.06.014]

-
Gertz, M., F. Fischer, M. Leipelt, D. Wolters and C. Steegborn. 2009. Identification of Peroxiredoxin 1 as a novel interaction partner for the lifespan regulator protein p66Shc. Aging 1: 254-265.
[https://doi.org/10.18632/aging.100017]

-
Giannini, T. C., G. D. Cordeiro, B. M. Freitas, A. M. Saraiva and V. L. Imperatriz-Fonseca. 2015. The dependence of crops for pollinators and the economic value of pollination in Brazil. J. Econ. Entomol. 108: 849-857.
[https://doi.org/10.1093/jee/tov093]

-
Glavan, G., S. Novak, J. Bozic and A. Jemec Kokalj. 2020. Comparison of sublethal effects of natural acaricides carvacrol and thymol on honeybees. Pestic. Biochem. Physiol. 166: 104567.
[https://doi.org/10.1016/j.pestbp.2020.104567]

-
Goulson, D. 2013. REVIEW: An overview of the environmental risks posed by neonicotinoid insecticides. J. Appl. Ecol. 50: 977-987.
[https://doi.org/10.1111/1365-2664.12111]

-
Huang, M., J. Dong, H. Guo and D. Wang. 2021. Effects of dinotefuran on brain miRNA expression profiles in young adult honey bees (Hymenoptera: Apidae). J. Insect Sci. 21: 1-8.
[https://doi.org/10.1093/jisesa/ieaa131]

-
Hwang, L. H., H. R. Yang, J. K. Lee, C. K. Kim and M. J. Kim. 2020. Determination of neonicotinoid pesticides in commercial agricultural products by LC-MS/MS. J. Food Hyg. Saf. 35: 312-318.
[https://doi.org/10.13103/JFHS.2020.35.4.312]

-
Ihara, M. and K. Matsuda. 2018. Neonicotinoids: molecular mechanisms of action, insights into resistance and impact pollinators. Curr. Opin. Insect Sci. 30: 86-92.
[https://doi.org/10.1016/j.cois.2018.09.009]

-
Jin, G., H. Hrithik, E. Mandal, E. J. Kil, C. Jung and Y. Kim. 2024. Phospholipase A2 activity is required for immune defense of European (Apis mellifera) and Asian (Apis cerana) honeybees against American fourbrood pathogen, Paenibacillus larvae. PLoS One 19: e0290929.
[https://doi.org/10.1371/journal.pone.0290929]

-
Kim, E. H., J. H. Lee, J. H. Sung, J. H. Lee, Y. H. Shin and J. H. Kim. 2014. Exposure and risk assessment for operator exposure to insecticide acetamiprid during water melon cultivation in greenhouse using whole body dosimetry, Korean J. Pestic. Sci. 18: 247-257.
[https://doi.org/10.7585/kjps.2014.18.4.247]

-
Kim, J. W., J. M. Lee, D. S. Lee, S. T. Kang, D. W. Kim, D. S. Lee, K. Z. Riu and K. H. Boo. 2015. Residual chracteristics of insecticide acetamiprid in asparagus under greenhouse condition. Korean J. Pestic. Sci. 19: 204-209.
[https://doi.org/10.7585/kjps.2015.19.3.204]

-
Klein, A. M., B. E. Vaissière, J. Cane, I. Steffan-Dewenter, S. A. Cunningham, C. Kremen and T. Tscharntke. 2007. Importance of pollinators in changing landscapes for world crops. Proc. Biol. Sci. 274: 303-313.
[https://doi.org/10.1098/rspb.2006.3721]

-
Krupke, C. H., G. J. Hunt, B. D. Eitzer, G. Andino and K. Given. 2012. Multiple routes of pesticide exposure for honey bees living near agricultural fields. PLoS One 7: e29268.
[https://doi.org/10.1371/journal.pone.0029268]

-
Li, H. L., J. Tan, X. M. Song, F. Wu, M. Z. Tang, Q. Y. Hua, H. Zheng and F. Hu. 2017. Sublethal doses of neonicotinoid imidacloprid can interact with honey bee chemosensory protein 1 (CSP1) and inhibit its function. Biochem. Biophys. Res. Commun. 486: 391-397.
[https://doi.org/10.1016/j.bbrc.2017.03.051]

-
Livak, K. J. and T. D. Schmittgen. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods 25: 402-408.
[https://doi.org/10.1006/meth.2001.1262]

-
Lundin, O., M. Rundlof, H. G. Smith, I. Fries and R. Bommarco. 2015. Neonicotinoid insecticides and their impacts on bees: a systematic review of research approaches and identification of knowledge gaps. PLoS One 10: e0136928.
[https://doi.org/10.1371/journal.pone.0136928]

-
MacMillan, H. A., J. M. Knee, A. B. Dennis, H. Udaka, K. E. Marshall, T. J. S. Merritt and B. J. Sinclair. 2016. Cold acclimation wholly reorganizes the Drosophila melanogaster transcriptome and metabolome. Sci. Rep. 6: 1-14.
[https://doi.org/10.1038/srep28999]

- Matsumoto, T. 2013. Reduction in homing flights in the honey bee, Apis mellifera, after a sublethal dose of neonicotinoid insectcides. Bull. Insectol. 66: 1-9.
-
Mclntosh, J. A., C. C. Farwell and F. H. Arnold. 2014. Expanding P450 catalytic reaction space through evolution and engineering. Curr. Opin. Chem. Biol. 19: 126-134.
[https://doi.org/10.1016/j.cbpa.2014.02.001]

-
Mucci, C. A., L. Ramirez, R. S. Giffoni and L. Lamattina. 2021. Cold stress induces specific antioxitant responses in honey bee brood. Apodologie 52: 596-607.
[https://doi.org/10.1007/s13592-021-00846-w]

-
Mullin, C. A., M. Frazier, J. L. Frazier, S. Ashcraft, R. Simonds, D. vanEngelsdorp and J. S. Pettis. 2010. High levels of miticides and agrochemicals in North American apiaries: Implications for honey bee health. PLoS One 5: e9754.
[https://doi.org/10.1371/journal.pone.0009754]

-
Ollerton, J., R. Winfree and S. Tarrant. 2011. How many flowering plants are pollinated by animals. Oikos 120: 321-326.
[https://doi.org/10.1111/j.1600-0706.2010.18644.x]

-
Peng, Y. C. and E. C. Yang. 2016. Sublethal dosage of imidacloprid reduces the microglomerular density of honey bee mushroom bodies. Sci. Rep. 6: 19298.
[https://doi.org/10.1038/srep19298]

-
Peng, Z., P. G. Green, Y. Arakane, M. R. Kanost and M. J. Gorman. 2014. A multicopper oxidase-related protein is essential for insect viability, longevity and ovary development. PLoS One 9: e111344.
[https://doi.org/10.1371/journal.pone.0111344]

-
Potts, S. G., V. Imperatriz-Fonseca, H. T. Ngo, M. A. Aizen, J. C. Biesmeijer, T. D. Breeze, L. V. Dicks, L. A. Garibaldi, H. Rosemary, J. Settele and A. J. Vanbergen. 2016. Safeguarding pollinators and their values to human wellbeing. Nature 540: 220-229.
[https://doi.org/10.1038/nature20588]

-
Ramirez, L., P. Negri, L. Sturla, L. Guida, T. Vigliarolo, M. Maggi, M. Eguaras, E. Zocchi and L. Lamattina. 2017. Abscisic acid enhances cold tolerance in honeybee larvae. Proc. R. Soc. B Biol. Sci. 284: 20162140.
[https://doi.org/10.1098/rspb.2016.2140]

-
Saleem, M. S., Z. Y. Huang and M. O. Milbrath. 2020. Neonicotinoid pesticides are more toxic to honey bees at lower temperatures: Implications for overwintering bees. Front. Ecol. Evol. 8: 556856.
[https://doi.org/10.3389/fevo.2020.556856]

-
Samson-Robert, O., L. Geneviève, M. Pierre-Luc, C. Madeleine, D. Nicolas and F. Valérie. 2015. Increased acetylcho-linesterase expression in bumble bees during neonicotinoid-coated corn sowing. Sci. Rep. 5: 12636.
[https://doi.org/10.1038/srep12636]

- SAS Institute Inc. 1989. SAS/STAT User’s guide. Cary, NC.
-
Shi, T., S. Burton, Y. Zhu, Y. Wang, S. Xu and L. Yu. 2018. Effect of field-realistic concentrations of carbandazim on survival and physiology in forager honey bees (Hymenoptera: Apidae). J. Insect Sci. 18: 1-5.
[https://doi.org/10.1093/jisesa/iey069]

-
Simondelso, N., V. Amaralrogers, L. P. Belzunces, J. Bonmatin, M. Chagnon, C. A. Downs, L. Furlan, D. W. Gibbons, C. Giorio and V. Girolami. 2015. Systemic insecticides (neonicotinoids and fipronil): trends, uses, mode of action and metabolites. Environ. Sci. Pollut. Res. 22: 5-34.
[https://doi.org/10.1007/s11356-014-3470-y]

-
Sung, K. M., W. J. Kim and J. S. Yoon. 2023. Past and present of double-stranded RNA as a Sacbrood virus inhibitor for Asian honeybee, Apis cerana. J. Apic. 38: 93-101.
[https://doi.org/10.17519/apiculture.2023.04.38.1.93]

- Tirado, R., G. Simon and P. Johnston. 2013. A review of factors that put pollinators and agricultures in Europe at risk. Greenpeace Research Laboratories Technical Report. 1st ed., 48p. Greenpeace International, Amsterdam.
-
Tsvetkov, N., S. Bahia, B. Calla, M. R. Berenbaum and A. Zayed. 2023. Genetics of tolerance in honeybees to the neonicotinoid clothianidin. iScience 26: 106084.
[https://doi.org/10.1016/j.isci.2023.106084]

- vanEngelsdorp, D., R. Underwood, D. Caron and J. Hayes. 2007. An estimate of managed colony losses in the winter of 2006-2007: A report commissioned by the apiary inspectors of America. Am. Bee J. 147: 599-603.
-
Vatanparast, M., S. Ahmed, D. H. Lee, S. H. Hwang, B. Hammock and Y. Kim. 2020. EpOMEs act as immune suppressors in a lepidopteran insect, Spodoptera exigua. Sci. Rep. 19: 20183.
[https://doi.org/10.1038/s41598-020-77325-2]

-
Vlogiannitis, S., K. Mavridis, W. Dermauw, S. Snoeck, E. Katsavou, E. Morou, P. Harizanis, L. Swevers, J. Hemingway, R. Feyereisen, T. H. Leeuwen and J. Vontash. 2021. Reduced proinsecticide activation by cytochrome P450 confers coumaphos resistance in the major bee parasite Varroa destructor. Proc. Natl. Acad. Sci. USA 118: e2020380118.
[https://doi.org/10.1073/pnas.2020380118]

-
Willard, S. S. and S. Koochekpour. 2013. Glutamate, glutamate receptors, and downstream signaling pathways. Int. J. Biol. Sci. 9: 948-959.
[https://doi.org/10.7150/ijbs.6426]

-
Wu, L., Z. Yu, Q. Jia, X. Zhang, E. Ma, S. Li, K. Y. Zhu, R. Feyereisen and J. Zhang, 2020. Knockdown of LmCYP 303A1 alters cuticular hydrocarbon profiles and increases the susceptibility to desiccation and insecticides in Locusta migratoria. Pestic. Biochem. Phys. 168: 104637.
[https://doi.org/10.1016/j.pestbp.2020.104637]

-
Zhao, Y., Z. Wang, J. Zhu and N. Liu. 2020. Identification and characterization of detoxification genes in two cerambycid beetles, Rhaphuma horsfieldi and Xylotrechus quadripes (Coleoptera: Cerambycidae: Clytini). Comp. Biochem. Physiol. Part B 243: 110431.
[https://doi.org/10.1016/j.cbpb.2020.110431]