메타바코딩을 통한 아까시꿀에서 미생물 프로파일링
Abstract
The origin of food product affects its value assessment. This trend has led to various methods for tracing provenance, one of which is based on DNA. DNA-based analysis utilizing residual tissues of organisms potentially present in honey can be employed to determine its identity. Two-types of DNA, cell-free DNA and pollen DNA, were isolated from five samples of honey. A total of ten DNA samples were analyzed using the 16S marker for metagenomic barcoding analysis, identifying thirty species of bacteria. The quantitative distribution of these bacterial species exhibited variation according to their geographical origin, and DNA purifying methods. Thus, DNA-based metagenomic barcoding analysis enabled for the detection of diverse micoorganisms within the honey and provided differences between production regions, suggesting the possibility of distinguishing the origin.
Keywords:
Metabarcoding, Acacia honey서 론
꿀은 식물의 꽃꿀을 이용하여 꿀벌이 생산하는 천연감미물질로 탄수화물 특히 포도당과 과당으로 구성되어 있으며 그 외 단백질, 효소, 아미노산, 유기산, 미네랄, 비타민, 페놀성 화합물, 항균화합물과 같은 생리 활성을 갖는 화합물들을 포함하고 있다. 또한 꿀은 꿀샘 식물이 꽃을 피우는 시기, 꿀샘 식물의 분포지역 등에 따라 향, 맛의 차이를 보이며 독특한 생리활성과 치료제로서의 잠재력을 갖는다 (Brudzynski, 2021; Pathiraja et al., 2023). 꿀은 항염증, 항산화, 항균, 몇몇 암에 대한 항암작용부터 변비, 식욕부진, 위장 문제 개선에 영향을 줄 수 있음이 보고된 바 있다 (Kek et al., 2017). 전 세계적으로 가장 일반적인 식품 중 하나인 꿀은 미국 약전의 식품사기 데이터베이스 (Food Fraud Database)에 따르면 꿀의 근원에 대한 위조 및 혼합과 같은 조작이 세 번째로 흔한 식품으로 나타났다. 문제의 꿀은 저품질의 꿀이나 설탕용액을 혼합하거나 출처를 식별하기 어렵게 하기 위해 화분을 제거하거나 특정 화분을 첨가하거나 원산지를 숨기기 위한 우회 수입하는 방법으로 이루어진다 (Chavan et al., 2022). 이러한 문제를 해결하기 위해 가장 오래된 꿀 식별방법으로 현미경을 이용한 화분검사와 유도결합플라즈마질량분석법 (ICP-MS), 핵자기공명분광법 (NMR), 근적외선분광법 (NIR), 그리고 가스크로마토그래피 (GC) 등의 분석 기기를 이용한 검사법이 사용되어 왔다. 그러나 현미경을 이용한 화분 검사는 화분 함량이 낮거나 필터링된 꿀 시료를 식별하는 데 어려움이 있으며 유도결합플라즈마질량분석법 (ICP-MS), 핵자기공명분광법 (NMR), 근적외선분광법 (NIR), 그리고 가스크로마토그래피 (GC) 등의 분석기술은 그 결과에서 특이성이 한정적인 점과 고도의 장비에 의존하게 된다. 2000년대에 들어서 차세대 염기서열 분석을 이용하는 DNA barcoding 분석법은 DNA를 기반으로 식물의 진화, 종 보존 관련과 같은 학술적 연구, 범죄현장에서 생물학적 표본을 통한 증거확보뿐 아니라 식품 인증 분야에서 급속도로 확산되어 왔다 (Heber, et al., 2003; Miller, 2007; Dawan and Ahn, 2022; Gorini et al., 2023; Letsiou et al., 2024). 이 방법은 생물종을 식별하고 분류하기 위해 특정한 DNA 서열을 사용하는 기술로 화학적 분석법과 현미경법보다 시료에 대해 많은 정보를 제공하며 꿀의 원산지를 식별하는 데 잠재력이 있음이 여러 연구들을 통해 입증되었다 (Bruni et al., 2015; Laha et al., 2017; Saravanan et al., 2019; Milla et al., 2023; Namin et al., 2022; Soares et al., 2023). 최근 꿀에서 작은 RNA 분자들의 구성 및 특성에 대한 분석 정보들이 분자 기반 꿀 인증 도구로 제안되기도 하였다 (Gismondi et al., 2017; Smith et al., 2021). 이 연구들은 꿀의 높은 당함량이 핵산을 안정적인 형태로 존재할 수 있게 함을 보여주었다. 꿀에 존재할 수 있는 eDNA (environmental DNA)는 바이러스, 박테리아, 곰팡이, 식물, 꿀벌 및 기타곤충에서 기인하며 이러한 eDNA는 바코드화된 특정 유전자를 증폭하여 염기서열 분석을 통해 분석한다 (Bovo et al., 2018, 2020; Utzeri et al., 2018; Wirta et al., 2021). 연구를 통해 알려진 바코드 유전자로서는 박테리아의 경우 16S rRNA 유전자, 식물체는 엽록체 trnL, rbcL, matK. trnH-psbA, 리보좀에 ITS, ITS2, 그리고 꿀벌 및 기타 곤충의 경우 cytochrome c oxidase subunit (COI)를 대상으로 하여 꿀에서 박테리아, 식물 및 곤충 DNA의 분류학적 구성을 예측하거나 보완할 수 있는 결과가 보고된 바 있다 (Bruni et al., 2015; Laha et al., 2017; Saravanan et al., 2019; Chavan et al., 2022; Letsiou et al., 2024). 이에 이 연구에서는 아까시꿀에서 16S RNA 유전자를 대상으로 차세대 염기서열 분석하여 얻은 미생물종의 프로파일을 바탕으로 데이터베이스를 구축하여 꿀의 생산지 인증 및 품질 관리도구로 활용 가능성을 알아보고자 한다.
재료 및 방법
1. 시료
국내 3개 지역 (D1, D2, D3)과 국외 2개 지역 (AX, AY)의 아까시꿀을 대상으로 꿀에 잔류하는 DNA와 화분 DNA를 나누어 추출하여 DNA barcoding 분석 실험에 사용하였다.
2. 꿀에서 DNA 추출법
꿀에 잔류하는 DNA는 꿀 5 g과 10 g을 각각 담아 Kim et al. (2021)의 방법으로 추출하였으며 꿀에 있는 화분 DNA는 꿀 10 g을 각각 tube에 담아 5배의 증류수를 넣고 잘 섞은 후 15,000 rpm에서 15분간 원심분리한 후 상층액은 버리고 모인 화분에 Genomic DNA from Tissue Kit (Macherey-Nagel, Düren, Germany)를 사용하여 사용자 매뉴얼에 따라 DNA를 추출하였다.
3. DNA 증폭체 라이브러리 준비 및 염기서열 분석
시퀀싱 라이브러리는 v3/v4를 증폭하기 위해 Illumina 16S 메타유전체 시퀸싱 라이브러리 제작방법에 따라 준비하였다. 사용한 5 ng 혹은 10 ng 꿀 시료 DNA는 5× 반응 버퍼, 1 mM dNTP 혼합물, 16S Forward 및 Reverse 프라이머, 500 nM Herculase II fusion DNA 중합효소 (Agilent Technologies, Santa Clara, CA)와 함께 PCR 증폭되었다. 1차 PCR 반응 조건은 열 활성화 단계로 95℃에서 30초, 55℃에서 30초, 72℃에서 30초로 25사이클, 그리고 72℃에서 5분간 반응시켰다. 이 반응은 Illumina 어댑터 오버행 시퀸스가 포함된 16S V3-V4 범용 프라이머쌍을 사용하였다 (Forward 5ʹ-TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGCCTACGGGNGGCWGCAG-3ʹ, Reverse 5ʹ-GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGGACTACHVGGGTATCTAATCC-3ʹ). 1차 PCR 산물은 AMPure 비드 (Agencourt Biosciences, Beverly, MA)로 정제하였다. 정제 후 1차 PCR 산물 10 μL를 NexteraXT 인덱스 프라이머를 사용하여 최종 라이브러리를 만들기 위해 PCR을 수행하였다. 2차 PCR 조건은 98℃에서 1분, 98℃ 15초, 55℃에서 15초, 72℃에서 30초 반응을 10회전한 후 72℃에서 3분간 반응하였다. PCR 산물은 AMPure 비드로 정제하였고 최종 정제된 산물은 KAPA Library Quantification Kit (for Illumina sequencing platforms)를 사용하여 정량화하였고 TapeStation D1000 Screen Tape (Agilent Technologies, Waldbronn, Germany)를 사용하여 정성 분석을 한 후 MiSeq 플랫폼 (Illumina, San Diego, USA)을 사용하여 시퀀싱을 진행하였다.
4. 데이터 분석
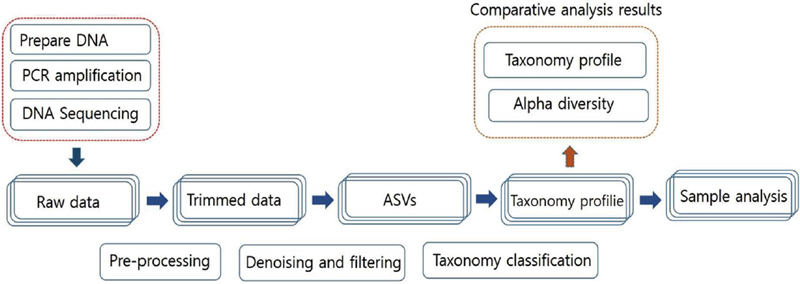
전체 분석 과정은 Fig. 1과 같으며 분석된 염기서열 대상은 DADA2, Uchime, Filtering하여 불필요한 서열들을 제거하여 얻은 ASV를 NCBI Refseq 16S (Ver. 2024-05-21) 데이터베이스와 BLAST 분석하였다. 각 시료에 존재하는 염기서열의 다양성을 나타내는 알파다양성은 Observed ASV (Amplicon Sequence Variants), Shannon, Simpson에 기초하여 평가하였고 베타다양성은 unweighted UniFrac 거리를 이용하여 분석하여 각각 어떤 분류군의 정보를 갖는지 시료별 어떤 분류군이 있는지 시각화를 통해 확인하였다.
결 과
1. 아까시꿀에서 미생물군의 동정을 위한 꿀 DNA 바코딩
박테리아의 16S rRNA 증폭체의 염기서열 분석은 국내 3개 지역 (D1, D2, D3)과 외국 2개 지역 (AX, AY)의 동일 아까시꿀 시료를 이용해 추출한 꿀 잔류 DNA와 꿀 내 화분 DNA에서 이루어졌다. 이들의 염기서열 분석은 35,572,180에서 72,775,178까지 길이를 분석할 수 있었고 trimmomatic 프로그램으로 보정한 후 27,866,872에서 61,294,365 염기서열 조각들이 생성되었고 (Table 1) 분석에 사용하기 위해 병합된 Amplicon Sequence Variants (ASVs)의 평균 길이는 452 bp로 나타났다.
2. 아까시꿀에서 미생물종 동정을 위한 DNA barcording
생성된 각 시료의 증폭체 (ASVs)을 대상으로 DADA2, Uchime, Filtering하여 불필요한 서열들을 제거하여 얻은 observed ASVs 수는 2157부터 6729로 시료별 차이를 보였으나 Shannon entropy와 Simpson 값을 통해 꿀에서 추출한 잔류 DNA와 화분에서 유래한 화분 DNA 시료 내 종의 분포를 고려하였을 때 그 다양성을 추정한 값이나 발견되는 종의 집중도에는 큰 차이를 보이지 않았다 (Table 2).
3. 아까시꿀에서 미생물 DNA의 분류종 조성
5종의 지역적 차이를 갖는 아까시꿀 DNA 시료에서 30개의 박테리아종들이 분석되었다. 알파다양성을 통해 시료 내에서 미생물의 다양성을 관찰한 결과 이들은 Bacillus속에서 4종, Clostridium속에서 2종, Tetragenococcus속에서 2종, Solirubrobacter속에서 2종이 동정되었으며 30개의 종은 화분 DNA와 꿀 잔류 DNA 간에 구성적 차이를 나타내며 각 시료 내 미생물의 다양성을 볼 수 있었다. 모든 시료에서 4종을 포함하는 Bacillus속이 가장 풍부하게 나타났다. D3 시료 잔류 DNA에서만 Pseudocyanosarcina phycocyania가 존재하였다. 반면 D3 화분 DNA에서만 Staphlococcus caprae의 상대적 풍부도가 높게 나타났다 (Fig. 2).
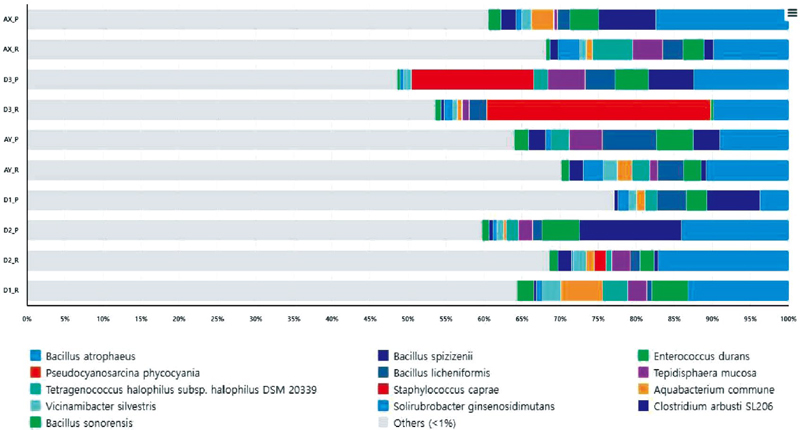
Bar plot showing the relative abundance of top 30 bacteria species obtained from NGS of pollen DNA and residual DNA from acacia honey. Sample D1, D2, and D3 represent domestic regions, while samples AX and AY are from foreign regions. P indicates pollen DNA, and R represents residual DNA.
상관풍부도에 따라 시료에 따라 1% 미만의 종으로 Kocuria marina, Skemanella aerolata, Capilimicrobium parvum, Luteitalea pratensis, Gaiella occulta, Paludibaculum fermentanas, Sphingomonas limnosedimincola, B. subtilis subsp. subtilis, Spriosoma areae, Arthrobacter pascens, C. disporicum, Sphaerobacter thermophiles, Solirubrobacter phytolaccae, Kroppensteditia sanguinis, Gemmatimonas phototrophica, Lawsonella clevelandensis이 있음을 볼 수 있었다 (Fig. 3).
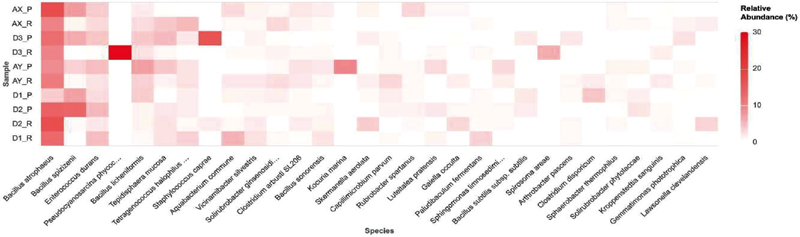
Major bacterial composition in acacia honey samples at species level. The relative abundance of the most abundant species in honey samples is shown. The top 30 bacteria species they belong to are shown here. Sample D1, D2, and D3 represent domestic regions, while samples AX and AY are from foreign regions. P indicates pollen DNA, and R represents residual DNA.
또한 시료 간 미생물 군집 구조의 차를 비교하는 베타다양성을 unweighted UniFrac distance를 가지고 분석하였다. 그 결과 5개의 아까시꿀 시료는 잔류 DNA와 화분 DNA 간 미생물종 군집 구조의 차이를 보였다. 또한 이들은 각 DNA에 따라 D1, D2, D3의 지역 간 차이를 보였다 (Fig. 4).
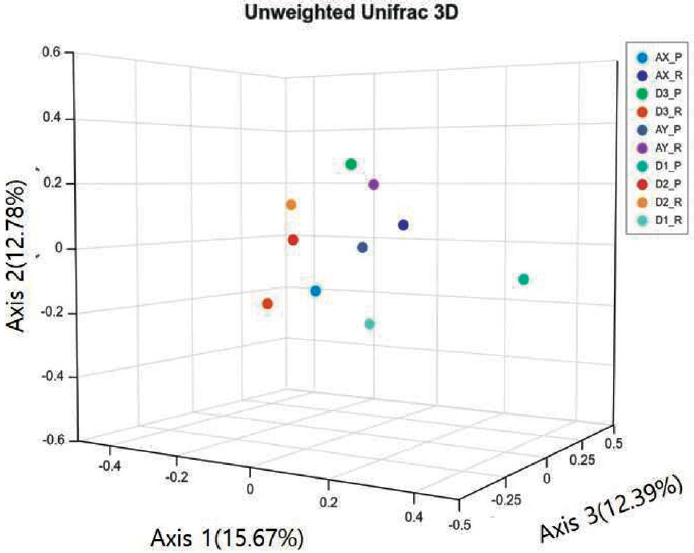
Comparison of bacterial composition among the honey samples from different DNA purifying methods and with different regions. Sample D1, D2, and D3 represent domestic regions, while samples AX and AY are from foreign regions. P indicates pollen DNA, and R represents residual DNA. Ordination analysis was performed using the unweighted UniFrac distance.
고 찰
꿀을 인증하는 방법은 화분의 형태적 분석과 꿀이 가지고 있는 화학적 특성을 분석하는 방법이 전통적으로 사용되어 왔다. 그러나 이들은 많은 식물 종이나 변종을 구별하기 어렵다. 이로 인해 꿀의 정확한 꽃 기원을 특정하기 어려울 수 있다. 또한 특정 유형의 화분이 존재한다고 해서 그 꿀이 특정 지역에서 생산되었다는 것을 의미하지는 않는다. 화분은 바람이나 벌에 의해 먼 거리까지 운반될 수 있어 지리적 출처를 정확하게 판단할 수 없게 된다. 또한 화분 형태 분석은 숙련된 분석가가 현미경을 사용하여 화분 입자를 식별해야 하는데 이는 시간이 많이 걸리고 분석가의 해석에 따라 결과가 달라질 수 있는 주관적인 과정이다 (Bruni et al., 2015; Hawkins et al., 2015). 꿀은 매우 다양한 화합물을 포함하고 있어 이들의 농도, 환경조건, 저장 및 가공방법 등에 따라 크게 달라질 수 있다. 이로 인해 꿀의 화학적 성분을 토대로 명확하고 일관된 인증 마커를 설정하기가 어렵다. 꿀의 화학적 프로필은 지역의 기후, 토양 유형, 그리고 꿀의 가공 및 저장방법에 의해 영향을 받을 수 있다 (El Sohaimy et al., 2015; da Silva et al., 2020). 이러한 변동은 꿀의 화학적 조성과 꽃 기원 또는 지리적 기원 간의 관계를 모호하게 만들 수 있다. 화합물 분석은 때때로 혼입물을 감지하지 못할 수 있다. 특히 혼입물이 꿀에 자연적으로 존재하는 화합물을 모방할 경우 이를 구별하기 위해서는 정교한 분석방법이 필요하다. 이러한 한계로 꿀의 진위 및 출처를 정확하고 신뢰성 있게 판단하기 위해 DNA 메타바코딩과 같은 보완적 기술이 탐구되고 있다 (Laha et al., 2017; Nehal et al., 2021). 그 응용분야로 다양한 환경에서 채취한 샘플이 어떤 종인지 빠르고 정확하게 식별하는 데 기존에 알려지지 않은 새로운 종을 발견하는 데 물, 토양, 공기 등에서 미생물 또는 작은 생물의 다양성을 평가하는 데 야생동물 및 식물의 불법 거래를 감시하는 데 적용하고 있다. 이처럼 DNA barcoding 분석은 분류학, 생태학, 환경과학, 보존 생물학 등 다양한 생물학적 연구 및 실용분야에서 중요한 도구로 자리잡고 있다 (Hebert et al., 2003; Miller, 2007; Dawan and Ahn, 2022; Gorimi et al., 2023; Lanubile et al., 2024; Letsiou et al., 2024). 최근에는 꿀에도 이 방법을 적용하여 성과를 보고하고 있다 (Kek et al., 2017; Laha et al., 2017; Milla et al., 2023). DNA를 이용한 분석은 DNA 유형에 따라 결과가 차이를 보인다 (Chavan et al., 2022). 우리의 실험 결과에서도 DNA 유래에 따라 차이를 보였다 (Figs. 2, 3). 화분 DNA와 잔류 DNA에서의 미생물종의 분포가 다른 이유로는 화분 DNA는 주로 꿀벌이 수집한 식물의 화분에서 유래하며 이에 따라 식물 표면에 존재하는 미생물일 가능성이 높은 반면 잔류 DNA는 꿀 속에서 죽은 세포나 분해된 세포에서 유래하여 벌집 내부나 꿀벌의 장내 미생물 등 다양한 환경에서 유래한 미생물의 잔재일 수 있다 (Wirta et al., 2021; Wu et al., 2021; Xiong et al., 2023; Anderson and Copeland, 2024). 꿀의 원산지나 인증에 적합한 DNA 유형에 대한 연구는 DNA 출처에 따라 주로 화분 DNA를 중심으로 이루어졌다 (Bruni et al., 2015; Hawkins et al., 2015; Laha et al., 2017; Milla et al., 2023). 그러나 꿀에서 화분 DNA를 추출하여 메타바코딩 분석을 하기 위해 일정량의 꿀 내에 존재하는 화분을 얻기 위해서는 많은 양의 꿀이 필요하고 또한 화분 세포벽은 견고하여 DNA를 충분히 얻는 데 시간적 노력이 필요하다. 반면 잔류 DNA는 많은 양의 꿀 소비와 화분을 모으는 전처리 과정을 거치지 않고 얻을 수 있어 시간적으로 분석이 좀 더 용이해질 수 있다. 우리의 실험 결과는 화분 유래 DNA 시료나 잔류 DNA 시료 모두 지역적 미생물 군집의 구조적 차이를 나타냄을 알 수 있었다. 종의 다양성이 확보된 잔류 DNA 시료에서는 지역적 특성을 반영할 수 있을 것으로 보인다. 그러나 추가적인 실험을 통해 다양한 데이터의 축적과 이에 따른 표준화가 필요할 것으로 판단된다. 꿀은 높은 삼투압, 산도 및 향, 미생물 특성을 가지고 있어 매우 낮은 미생물 부하를 가지며 이러한 조건에서 생존할 수 있는 미생물은 거의 없지만 꿀 속 미생물은 주로 2가지 출처에서 기인한다고 보고하고 있다. i) 꿀벌 장내, 꿀벌 병원균, 꽃가루, 꽃 및 먼지와 같은 자연적 출처와 ii) 사람 및 처리 기구로 인한 수확 후 오염으로 추정하고 있다. 따라서 이는 수확 후 처리, 위생, 상업적 품질에 대한 적절한 기술의 지표로 사용되고 있다 (Pathiraja et al., 2023). 꿀내 존재하는 미생물 간의 복잡한 상호작용이 꿀의 항균 특성에 중요한 역할을 한다. 미생물 간의 길항적 상호작용은 다양한 항균펩타이드, 계면활성제, 그리고 다른 생리활성물질의 생산을 촉진하며 이로 인해 생성된 이차대사산물은 꿀의 광범위한 항균성질에 크게 기여한다. 꿀에 존재하는 주된 미생물로는 20개 속의 분류군과 꿀벌 장내 미생물 10개 속이 존재한다. 최근 이와 같은 미생물군들이 꽃꿀과 벌의 상호작용으로 생성한 항균화합물의 저장고로서의 꿀의 역할에 주목하였다 (Brudzynski, 2021). 앞으로 꿀에 존재하는 미생물군집에 대한 연구는 꿀의 기능성 연구에 활용될 수 있을 것으로 판단된다. 이번 연구에 사용한 아까시꿀 시료에서는 병원성 미생물은 동정되지 않았으며 발견된 미생물은 Bacillus속에서 4종, Clostridium속에서 2종, Tetragenococcus속에서 2종, Solirubrobacter속에서 2종이 동정되었고 그 외 단일 종들이 존재하였다. Pathiriaka et al. (2023)의 연구에 따르면 한국, 미국, 베트남, 인도의 꿀에서 미생물 군집 분석 결과 우리나라 아까시꿀에서 13개 Firmicutes문에 속하는 미생물군집이 동정되었음을 보고한 바 있다. 그들의 결과와 비교하였을 때 Bacillus, Clostridium, Spingomonas속의 존재는 우리의 결과와 부합됨을 확인하였다. 이 결과에서 얻은 미생물 종들의 군집 구조에 대한 지속적인 연구는 꿀의 이력 및 원산지 구별에 화분학 및 화학적 성분 분석과 함께 활용할 수 있을 것으로 생각되며 미생물의 지속적 연구는 품질관리에도 활용될 수 있을 것으로 판단된다.
Acknowledgments
본 연구는 국립농업과학원 농업과학기반기술연구 (과제번호: PJ01745703)에 의하여 수행되었습니다.
References
-
Anderson, K. E. and D. C. Copeland. 2024. The honey bee “hive” microbiota: meta-analysis reveals a native and aerobic microbiota prevalent throughout the social resource niche. Front. Bee Sci. 2: 1410331.
[https://doi.org/10.3389/fr2021.1410331]

-
Bovo, S., A. Ribani, V. J. Utzeri, G. Schiavo, F. Bertolini and L. Fontanesi. 2018. Shotgun metagenomics of honey DNA: Evalutation of a methodological approach to describe a multi-kingdom honey bee derived environmental DNA signature. PLoS One 13: e0205575.
[https://doi.org/10.1371/journal.pone.0205575]

-
Bovo, S., V. J. Utzeri, A. Ribani, R. Cabbri and L. Fontanesi. 2020. Shotgun sequencing of honey DNA can describe honey bee derived environmental signatures and the honey bee hologenome complexity. Sci. Rep. 10: 9279.
[https://doi.org/10.1038/s41598-020-66127-1]

-
Brudzynski, K. 2021. Honey as an ecological reservoir of anti-bacterial compounds produced by antagonistic microbial interactions in plant nectars, honey and honey bee. Antibiotics 10: 551.
[https://doi.org/10.3390/antibiotics10050551]

-
Bruni, I., A. Galiberti, L. Caridi, D. Scaccabarozzi, F. De Mattia, M. Casiraghi and M. Labra. 2015. A DNA barcoding approach to identify plant species in multiflower honey. Food Chem. 170: 308-315.
[https://doi.org/10.1016/j.foodchem.2014.08.060]

-
Chavan, D., J. R. T. Adolacion, M. Crum, S. Nandy, K. H. Lee, B. Vu, K. Kourentzi, A. Sabo and R. C. Willson. 2022. Isolation and barcoding of trace pollen-free DNA for authentication of honey. J. Agric. Food Chem. 70: 14084-14095.
[https://doi.org/10.1021/acs.jafc.2c04309]

-
da Silva, P. M., L. V. Gonzaga, M. S. de Azevedo, F. C. Biluca, M. Schulz, A. C. O. Costa and R. Fett. 2020. Stability of volatile compounds of honey during prolonged storage. J. Food Sci. Technol. 37: 1167-1182.
[https://doi.org/10.1007/s13197-019-04163-0]

-
Dawan, J. and J. Ahn. 2022. Application of DNA barcoding for ensuring food safety and quality. Food Sci. Biotechnol. 31: 1355-1364.
[https://doi.org/10.1007/s10068-022-01143-7]

-
El Sohaimy, S. A., S. H. D. Masry and M. G. Shehata. 2015. Physicochemical characteristics of honey from different origins. Ann. Agric. Sci. 60: 279-287.
[https://doi.org/10.1016/j.aoas.2015.10.015]

-
Gismondi, A., G. D. Marco and A. Canini. 2017. Detection of plant microRNAs in honey. PLoS One 12: e0172981.
[https://doi.org/10.31371/journal.pone.0172981]

-
Gorini, T., V. Mezzasalma, M. Deligia, F. D. Mattia, L. Campone, M. Labra and J. Frigerio. 2023. Check your shopping cart: DNA barcoding and mino-barcoding for food authentication. Foods 12: 2392.
[https://doi.org/10.3390/foods12122392]

-
Hebert, P. D. N., A. Cywinska, S. L. Ball and J. R. deWaard. 2003. Biological identifications through DNA barcodes. Proc. Biol. Sci. 270: 313-321.
[https://doi.org/10.1098/rspb.2002.2218]

-
Kek, S. P., N. L. Chin, S. W. Tan, Y. A. Yusof and L. S. Chua. 2017. Molecular identification of honey entomological origin based on bee mitochondrial 16S rRNA and COI gene sequences. Food Control 78: 150-159.
[https://doi.org/10.1016/j.foodcont.2017.02.025]

-
Kim, S., S. G. Kim, S. M. Han and B. Yoon. 2021. Optimization of practical protocol for residual DNA from honey. J. Apic. 36: 195-206.
[https://doi.org/10.17519/apiculture.2021.09.36.3.195]

-
Laha, R. C., S. D. Mandal, L. Ralte, L. Ralte, N. S. Kumar, G. Gurusubramanian, R. Satisshkumar, R. Mugasimangalam and N. A. Kuravadi. 2017. Meta-barcoding in combination with palynological inference is a potent diagnostic marker for honey floral composition. AMB Expr. 7: 132.
[https://doi.org/10.1186/s13568-017-0429-7]

-
Lanubile, A., L. Stagnati, A. Marocco and M. Busconi. 2024. DNA-based techniques to check quality and authenticity of food, feed and medicinal products of plant origin: A review. Trends Food Sci. Technol. 149: 104568.
[https://doi.org/10.1016/j.tifs.2024.104568]

-
Letsiou, S., P. Madesis, E. Vasdekis, C. Montemurro, M. E. Grigoriou, G. Skavdis, V. Moussis, A. E. Koutelidakis and A. G. Tzakos. 2024. DNA barcoing as a plant identification method. Appl. Sci. 14: 1415.
[https://doi.org/10.3390/app14041415]

-
Milla, L., A. Schmidt-Lebuhn, J. Bovill and F. Encinas-Viso. 2023. Monitoring of honey bee floral resources with pollen DNA metabarcoding as a complementary tool to vegetation surveys. Ecol. Solut. Evid. 3: e12120.
[https://doi.org/10.1002/2688-8319.12120]

-
Miller, S. 2007. DNA barcoding and the renaissance of taxonomy. Proc. Natl. Acad. Sci. U.S.A. 140: 4775-4776.
[https://doi.org/10.1073/pnas.0700466104]

-
Namin, S. M., M. J. Kim, M. Son and C. Jung. 2022. Honey DNA metabarcoding revealed foraging resource partitioning between Korean native and introduced honey bees (Hymenoptera: Apidae). Sci. Rep. 12: 14394.
[https://doi.org/10.1038/s41598-022-18465-5]

-
Nehal, N., B. Choudhary, A. Nagpure and R. K. Gupta. 2021. DNA barcoding: a modern age tool for detection of adulteration in food. Crit. Rev. Biotechnol. 41: 767-791.
[https://doi.org/10.1080/07388551.2021.1874279]

-
Pathiraja, D., J. Cho, J. Kim and I. G. Choi. 2023. Metabarcoding of eDNA for tracking the floral and geographical origins of bee honey. Food Res. Int. 164: 112413.
[https://doi.org/10.1016/j.foodres.2022.112413]

-
Saravanan, M., G. Mohanapriya, R. Laha and R. Sathishkumar. 2019. DNA barcoding detects floral origin of Indian honey samples. Genome 62: 341-348.
[https://doi.org/10.1139/gen-2018-0058]

-
Smith, C., N. Cokcetin, T. Truong, E. Harry, G. Hutvagner and S. Bajan. 2021. Cataloguing the small RNA contnt of honey using next generation sequencing. Food Chem. Mol. Sci. 2: 100014.
[https://doi.org/10.1016/j.fochms.2021.100014]

-
Soares, S., F. Rodrigues and C. Delerue-Matos. 2023. Towards DNA-based methods analysis for honey: An update. Molecules 28: 2106.
[https://doi.org/10.3390/molecules28052106]

-
Utzeri, V. J., A. Ribani, G. Schiavo and F. Bertolini. 2018. Application of next generation semiconductor based sequencing to detect the botanical composition of monofloral, polyfloral and honeydew honey. Food Control 86: 342-349.
[https://doi.org/10.1016/j.foodcont.2017.11.033]

-
Wirta, H., N. Abrego, K. Miller, T. Roslin and E. Vesterinen. 2021. DNA traces the origin of honey by identifying plants, bacteria and fungi. Sci. Rep. 11: 4798.
[https://doi.org/10.1038/s41598-021-84174-0]

-
Wu, J., H. Lang, X. Mu, Z. Zhang, Q. Su, X. Hu and H. Zheng. 2021. Honey bee genetics shape the strain-level structure of gut microbiota in social transmission. Microbiome 9: 225.
[https://doi.org/10.1186/s40168-021-01174-y]

-
Xiong, Z. R., J. H. Sogin and R. W. Worobo. 2023. Microbiome analysis of raw honey reveals important factors influencing the bacterial and fungal communities. Front. Microbiol. 13: 1099522.
[https://doi.org/10.3389/fmicb.2022.1099522]