시맥 형질과 분자 마커에 의한 국내 서양종 꿀벌(Apis mellifera L.)의 계통 구분
Abstract
It was difficult to discriminate each inbred line of the European honeybees, Apis mellifea L., being kept for breeding program in Korea. The hybrid honeybees supplied by the governmental recommendation also need to be differentiated from other honeybee populations. Nineteen cross-points of worker bee’s wing vein were observed and measured their main characters by computer-assisted image analyzer. The six inbred lines and one triple hybrid were compared on their clouds resulted from principle component analysis by DrawWing software (Tofilski, 2004). The cluster zones from wing vein characters could contribute as the first discriminating tool of honeybee lines in Korea. To apply feasible molecular markers for honeybee lines in Korea, we found out seven micro-satellite markers from which the allele frequencies in each honeybee population could be differentiated.
Keywords:
Honeybee, Apis mellifera L., Line, Wing vein, Molecular marker서 론
유럽과 아프리카가 원산지인 서양종 꿀벌(Apis mellifera L.)은 근대 양봉산업의 발전과 더불어 전 세계로 확산하여 각 지역의 환경에 적응하여 정착하였다(Rinderer, 1986; Ruttner, 1988; Page and Laidlaw, 1992). 원산지의 서양종 꿀벌 약 26개 아종(亞種; subspecies) 중에서 가장 경제적 가치가 높은 것으로 4개 아종이 알려져 있다. 그 중 이탈리안(A. m. ligustica), 카니올란(A. m. carnica), 코카시안(A. m. caucasica) 꿀벌이 양봉에 적합한 우수 형질을 갖춤으로써 전 세계에 널리 보급되었고, 선호도가 떨어지는 북구흑색종(A. m. mellifera)은 주로 유럽에서만 사육되는 편이다(Dietz, 1992).
1900년대 초에 우리나라에 처음으로 도입된 서양종 꿀벌은 오랜 세월 여왕벌 공중교미 습성에 의해 계통 간 반복 교잡이 일어나 순종을 찾아보기가 어려웠다(최, 1974). 서양 꿀벌이 우리나라에 도입된 이후 최근까지 간헐적으로 외국 여왕벌을 들여와 일부 양봉 농가에 보급된 적이 있었는데, 1990년대에는 호주, 뉴질랜드에서 우수한 꿀벌이 패키지 벌로 대량 수입되어 널리 보급된 적이 있었다. 그러나 우수 계통이라도 격리교배로 잘 관리하지 않으면, 여왕벌의 공중교미에 의해 쉽게 잡종으로 형질이 저하될 수 있다(Adam et al., 1977). 따라서 꿀벌의 교유한 형질을 유지하고 유전적인 순계로 관리하기 위한 노력과 기술이 필요하다(Mackensen, 1955; Woyke, 1976).
꿀벌의 아종과 계통들을 구분하는 형질로는 형태적 특성과 행동습성의 차이를 이용하여 왔고, 최근에는 다양한 분자생물학적 방법이 응용되고 있다(Whitfield et al., 2006). 형태적 형질을 계측하고 수리학적인 다변량 통계분석에 의해 계통을 분류하는 방법(DuPraw, 1964)이 제시되었고, Ruttner 등(1978)이 제시한 42개 외부 형질이 측정 기준이 되어 지리적 변이계통을 구분하는데 이용되어 왔다. 최근 컴퓨터를 이용하여 자동 측정이 가능하고, 짧은 시간에 정확한 형질정보를 얻을 수 있는 날개의 시맥을 수리학적으로 분석하는 방법이 활용되고 있다(Tofilski, 2007, 2008).
최근 국내 꿀벌 육종연구의 일환으로 국내에서 수집하여 순계로 유지하고 있는 꿀벌 계통들과 이들의 교배조합에서 선발한 우수 교배종(이 등, 2014)을 상호간에 정확하게 구분하기가 어려운 실정이다. 이 연구는 국내 유전자원으로 보존하고 있은 서양종 꿀벌 6계통과 우수 교배종 1종을 대상으로 시맥형질 분석과 초위성체(micro-satellite) 분자마커에 의해 계통을 구분하는 방법을 제시하고자 수행하였다.
재료 및 방법
시험재료
이 연구에 사용한 서양종꿀벌(Apis mellifera L.) 계통은 꿀벌 육종 재료로 사용하기 위하여 2005년부터 2008년까지 국내에서 수집하여 순계로 분리한 6계통과 1개 교배종(이 등, 2014)으로 그 목록은Table 1과같다.
시맥 영상 및 주요인 분석
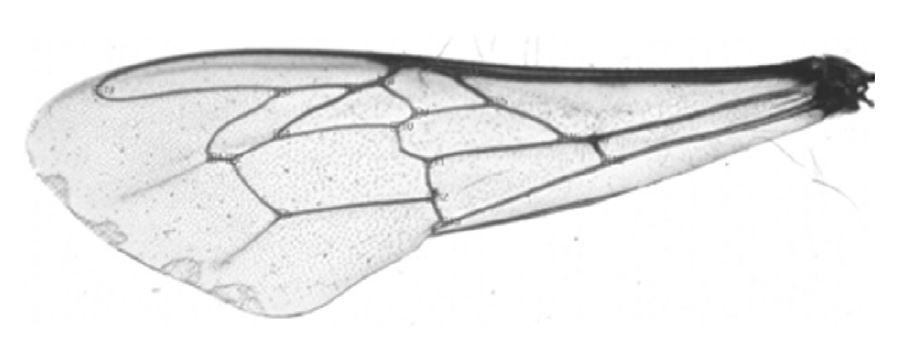
시맥 교차점을 측정하여 주요인 분석에 사용한 날개는 가능한 봉군 내에서 어린 일벌을 대상으로 채집하였다. 계통별 3개 봉군에서 봉군 당 30마리의 일벌을 채집하여 실험실에서 냉동 보관 후, 조사 시점에서 건조한 다음 핀셋으로 오른쪽 앞날개를 떼어내어 투명한 슬라이드 필름에 접착하였다. 날개가 고정된 슬라이드 필름을 전용스캐너(OpticFilm 8100, Plustek Co.)를 이용하여 이미지 파일을 생성하였고, 컴퓨터에서 명암을 보정한 후 DrawWing ver. 0.46(Tofilski, 2004; www.drawing.org)을 이용하여 앞날개 시맥의 19개 지점의 좌표 측정과 그 결과를 토대로 한 주요인 분석을 수행하였다(Fig. 1).
분자마커 분석
보유하고 있는 국내 서양종꿀벌의 계통을 판별하기 위하여 선발한 초위성체(micro-satellite) 분자마커는 차세대 DNA 염기분석에 의해 2~6개 염기단위의 단순반복 서열 모티브를 갖는 곳에서 발굴하였다(Kim et al., 2015). 자체 설계한 PCR primer에 의해 50개 초위성체 DNA를 대상으로 하여 재현성과 다형현상(polymorphism) 정도를 기준으로 9개 유전자를 최종 선발하였다. 이를 분자마커로 하여 대립유전자(allele) 길이와 유전자형 분석을 GeneMappeⓇ v.4.1 (Applied Biosystems, USA)을 통해 수행하였다. 각 계통별로 3개 봉군에서 봉군 당 일벌 3마리를 채집하여 DNA를 추출한 후, 7개 초위성체 마커에 대한 서양종꿀벌 6개 계통의 대립유전자 분포를 조사하였다.
결과 및 고찰
시맥 형질
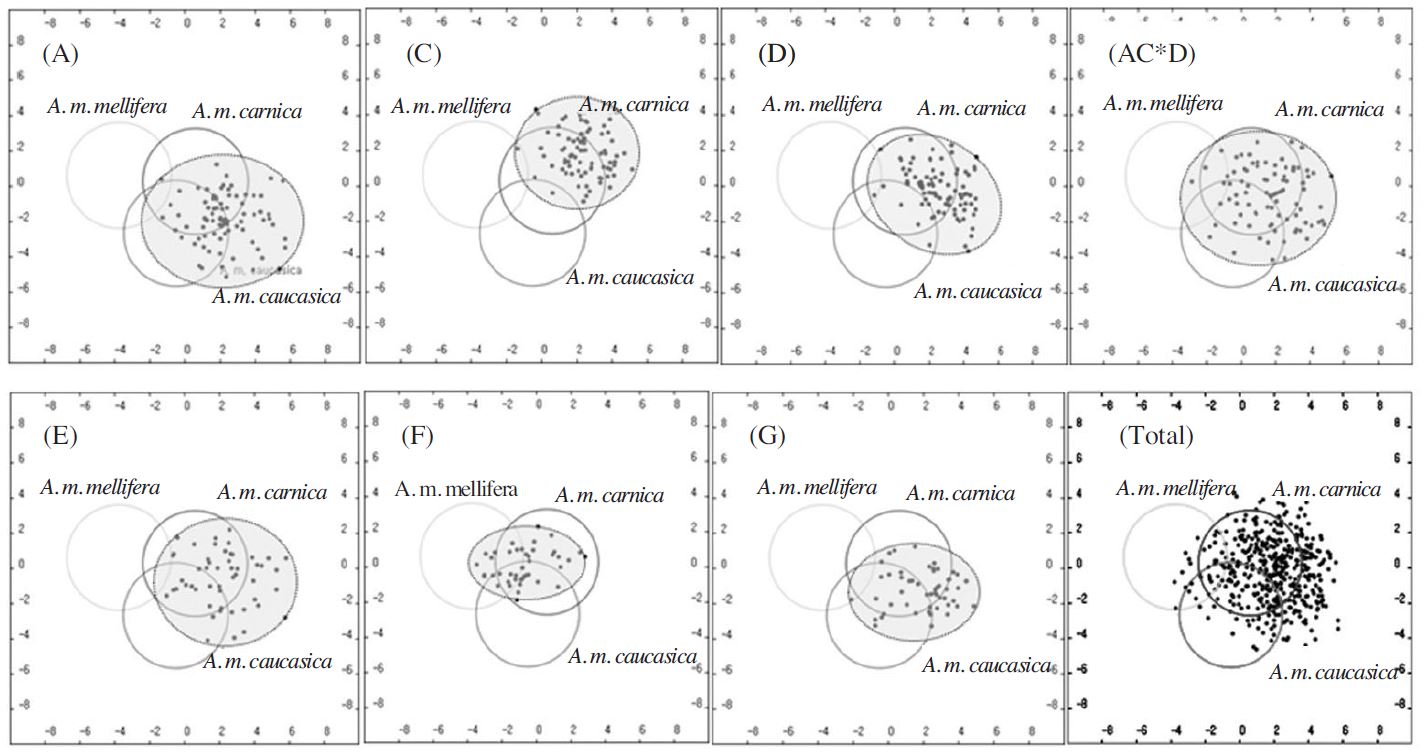
국내에서 수집한 서양종꿀벌 6계통(A, C, D, E, F, G)과 이들 교배조합에서 육종한 삼원교배종(ACxD)에서 수집한 일벌의 앞날개를 채취하여 시맥 형질(Fig. 1)에 대한 주요인 분석 결과는 Fig. 2와같다.
수집한 전체 집단에서의 형질 분포는 Gerula 등(2009)이 제시한 유럽의 북구흑색종(A. m. mellifera), 카니올란(A. m. carnica), 코카시안(A. m. caucasica) 등 3개 아종의 녹색, 청색, 적색의 분포영역에 걸쳐 넓게 분포하면서 주로 카니올란 영역(청색)과 그 외곽에 집중된 것으로 나타났다(Fig. 2-Total). Gerula 등(2009)이 이탈리안(A. m. ligustica) 아종은 이들 3개 영역 어디에도 분포할 수 있다고 강조한 점을 감안하면, 주로 이탈리안 아종 그룹으로 구성된 국내 서양종꿀벌을 카니올란 아종 그룹과 연관시키는 것은 큰 의미가 없다. 특히 이탈리안 계통에서 유래된 A, C, F, G 계통(Table 1)이 3개 아종 영역 내외부에 걸쳐 분포하는 점으로 보아 국내 이탈리안 계통에 대한 특정 영역을 여기에 설정하는 것은 불가능하고, 향후 국내 계통들을 기준으로 별도의 다변량 분석법을 시도할 필요가 있다.
루마니안 계통(Carpathian honeybee; A. m. carpatica)에서 유래된 D 계통의 경우에도 카니올란(A. m. carnica) 유래 E 계통과 유사하게 청색 영역으로 편중하는 경향을 보임으로써 Gerula 등(2009)이 제시한 분류 영역은 국내에 적용하기에는 무리가 따른다. 수밀력이 우수한 삼원교배종의 교배양식 (AxC)xD에서 1차 교배에 사용한 A 계통과 C 계통은 동일 이탈리안 아종임에도 불구하고 상이한 분포(Fig. 2A, 2C)를 보였고, 삼원교배종(ACxD)는 C 계통의 영역에서 다소 벗어나 주로 A와 D 계통의 분포 영역에서 나타나는 특징을 보였다.
이탈리안 아종에 속하는 F 계통은 유일하게 북구흑색종(A. m. mellifera)의 녹색 영역에 다수가 분포하는 특성을 보였던 점을 고려하여 국내 계통들에 대한 판별 능력을 개선하기 위한 차별화된 수리분석 방안을 마련할 필요가 있다. 또한 새로 보급을 시작한 우수삼원교배종의 시맥형질이 국내 양봉농가가 사육하고 있는 서양종꿀벌로부터 정확히 구분되어 판별될 수 있는지 여부를 광범위하게 조사하고, 이와 병행한 다변량 분석을 추가 적용해야 할 것이다.
초위성체 분자마커
국내 수집 6개 계통의 봉군에서 채집한 일벌을 대상으로 하여 Kim 등(2015)이 선발한 7개 초위성체 마커에 의한 대립인자의 분포를 조사한 결과는 Table 2와같다.
Table 2A는 2염기 반복서열인 P5 마커에 대한 계통별 14개 대립인자 분포를 보여주는 것으로 대립인자 173과 175는 모든 계통에 공통적으로 나타나고, 전체빈도가 가장 높은 대립인자 189는 C, E, G 계통에서 나타나고 있다. 대립인자 179는 F 계통, 185는 A 계통, 193과 195는 D 계통에 특이적으로 나타나고 있다.
3염기 반복서열인 P127 마커(Table 2B)에 대한 전체 98개 대립인자의 분포를 보면, 대립인자 207과 210가 모든 계통에 거의 공통적으로 나타나고 있으며, 198은 G 계통에, 213과 222는 F계통과 A 계통에 각각 특이성을 보이고 있다. 대립인자 210은 C 계통, 219는 F계통에 더욱 특이적인 것을 알 수 있다. P2716 마커는 계통 특이적인 대립인자를 다수 보유하고 있었는데, 대립인자 130은 F, 142는 G, 144는 A, 154는 E, 156은 G, 168은 F, 170은G에 특이적이다. 158을 갖고 있어 C 계통과 차별화된 D 계통을 포함하면, 전체 계통을 이 P2716 마커를 통해 구분이 가능할 것이다(Table 2C).
3염기 반복서열인 Table 2D의 P4461 마커는 대립인자가 4개로 제한되어 있고, 대립인자 195가 모든 계통에서 공통적으로 나타나지만 계통간 대립인자 빈도는 다소 차이를 보인다. 계통특이 대립인자를 찾아볼 수는 없으나 대립인자 255를 공유하는 C, E, F, G와 189를 공유하는 A, D 그리고, 192를 공유하는 A, F, G 등으로 그룹을 나누어 볼 수 있다. C와 E 계통은 195와 255를 갖는 점이 특징이다. P17394 마커는 14개의 대입인자를 갖고 있어 상대적으로 각 계통에 대한 판별이 용이하다(Table 2E). A 계통은 대립인자 248(22%), D 계통은 236(50%), E 계통은 228(28%), G 계통은 264(94%)만을 갖고 있어 상호 구분하기가 쉽다. 개별 특이 대립인자를 갖지 않는 C 계통과 F 계통은 256을 제외하고, 서로 상이한 대립인자를 보유하고 있기에 쉽게 구분할 수 있고, 대립인자 256도 그 빈도에 있어서는 C 계통 50%, F 계통 6%로 차이가 나타난다.
Table 2F의 P21438 마커에서는 대립인자 152(G 계통 제외)와 158가 전 계통에 공통되는 대립인자로 나타나지만, 그 빈도는 계통 간에 차이를 보인다. G 계통은 빈도는 다소 낮지만 대립인자 148(5.5%)과 162(11.0%)가 특징적으로 나타나고 있고, 나머지 5개 계통들은 152, 154, 156, 158, 160에서 상이한 분포와 빈도들 보인다. 2염기 반복서열인 P24351에서는 10개 대립인자가 나타나고 있는데, 그 중 224와 244는 전 계통에 공통적으로 나타난다. E, F, G 계통은 다소 낮은 빈도이지만, 각각 대립인자 262, 242, 214를 특징적으로 갖고 있다. 이 계통들을 포함한 모든 계통들이 각기 보유하고 있는 3~5개 대립인자에 대해 차별화된 분포 양상을 나타낸다(Table 2G).
이 조사는 계통별로 3개 봉군에서 봉군 당 3마리 일벌을 대상으로 18개 대립인자를 대상으로 불분명한 검출 건을 제외하고 조사한 것으로, 향후 조사 표본수를 늘려 보다 신뢰도 높은 판별 기준을 제시하는 것이 필요할 것 같다. 아울러 6개 계통뿐만이 아닌 교배계통과 일반 농가 사육계통, 그리고 원산지 꿀벌 표본에 대해서도 초위성체 마커별 대립인자와 그 빈도를 비교하는 작업이 수행되어야할 것이다.
적 요
우리나라에서 육종 연구를 위해 순계로 보존하고 있는 유럽 꿀벌 계통들과 정부에서 보급하고 있는 우수 교배종을 서로 구분하기가 어려운 실정이다. 일벌의 날개 시맥의 19개 교차점과 이를 토대로 한 주요 형질을 컴퓨터 영상 분석을 통해 측정하였다. 소프트웨어 DrawWing(Tofilski, 2004)를 이용하여 주요인 분석에 의한 2차원 분포도를 작성하여 6개 순계와 1개 교배종을 비교하였다. 시맥 형질의 분포 영역의 차이는 국내의 꿀벌 계통을 1차 판별하는 수단으로 활용될 수 있다. 또한 국내 꿀벌 계통을 판별하기에 적합한 분자 마커로서 7개 초위성체 마커를 선발하였고, 계통별로 분자마커에 대한 대립인자의 종류와 빈도조사를 통해 각 계통 집단을 구분할 수 있음을 확인하였다.
Acknowledgments
본 연구는 농촌진흥청 농업공동연구사업(과제번호: PJ008266)의 연구비 지원에 의하여 이루어졌습니다.
Reference
- 이명렬, 이만영, 심하식, 최용수, 김혜경, 변규호, 김인석, 권천락, (2014), 우수 삼원교배종 꿀벌(Apis mellifera L.)의 형질 특성: 수밀량, 월동, 청소행동, J. Apiculture, 29, p257-262.
- 최승윤, (1974), 신제 양봉학, p439, 집현사, 서울.
- Adam, J., E. D. Rothman, W. E. Kerr, and Z. L. Paulino, (1977), Estimation of the number of sex alleles and queen matings from diploid male frequencies in a population of Apis mellifera, Genetics, 86, p583-596.
- Dietz, A., (1992), Honey bees of the world. in The Hive and the Honeybee, p23-72, Dadant & Sons, Inc, Hamilton, Illinois.
-
DuPraw, E. J., (1964), Non linnean taxonomy, Nature, 202, p849-852.
[https://doi.org/10.1038/202849a0]

- Gerula, D., A. Tofilski, P. Wegrzynowicz, and W. Skowronec, (2009), Computer assisted discrimination of honeybee subspecies used for breeding in Poland, J. Apic. Sci, 53, p105-114.
-
Kim, H. K., I. S. Kim, M. L. Lee, Y. S. Choi, and B. R. Jin, (2015), Microsatellite markers developed by next generation sequencing differentiate inbred lines of Apis mellifera, J. Asia Pacific Ent, (in press).
[https://doi.org/10.1016/j.aspen.2015.10.003]

- Mackensen, O., (1955), Further studies on a lethal series in the honey bee, J. Hered, 46, p72-74.
- Page, R. E., and H. H. Laidlaw, (1992), Honey bee genetics and breeding. in The Hive and the Honeybee, p235-267, Dadant & Sons, Inc, Hamilton, Illinois.
- Rinderer, T. E., (1986), Bee genetics and breeding, p426, Academic Press, Orlando.
-
Ruttner, F., (1988), Biogeography and taxonomy of honeybees, p284, Springr Verlag, New York.
[https://doi.org/10.1007/978-3-642-72649-1]

-
Ruttner, F., L. Tassencourt, and J. Louveaux, (1978), Biometrical statistical analysis of the geographic variability of Apis mellifera L., Apidologie, 9, p363-381.
[https://doi.org/10.1051/apido:19780408]

-
Tofilski, A., (2004), DrawWing, a program for numerical description of insect wings, J. Insect Sci, 4, p1-5.
[https://doi.org/10.1673/031.004.1701]

-
Tofilski, A., (2007), Automatic measurements of honeybee wings, p289-298, in MacLeod, N. Ed, Automated object identification in systematics: theory, approaches, and applications, CRC Press, Boca Raton, Florida.
[https://doi.org/10.1201/9781420008074.ch17]

-
Tofilski, A., (2008), Using geometric morphometrics and standard morphometry to discriminate three honeybee subspecies, Apidologie, 39, p558-563.
[https://doi.org/10.1051/apido:2008037]

-
Whitfield, C. W., S. K. Behura, S. H. Berlocher, A. G. Clark, J. S. Johnston, W. S. Sheppard, D. R. Smith, A. V. Suarez, D. Weaver, and N. D. Tsutsui, (2006), Thrice out of Africa: Ancient and recent expansions of the honey bee, Apis mellifera, Science, 314, p642-645.
[https://doi.org/10.1126/science.1132772]

- Woyke, J., (1976), Population genetics studies on sex alleles in the honeybee using the example of the Kangaroo Island Bee Sanctuary, J. Apic. Res, 15, p105-123.