
꿀벌의 석고병, 백묵병 현장 진단을 위한 신속 Ascosphaera apis 및 Aspergillus flavus 검출법 개발
Abstract
Rapid detection methods against Ascosphaera apis and Aspergillus flavus were developed for Chalkbrood and Stonebrood in honeybee. Specific primer sets were selected which detect A. flavus or A. apis based on 18s ribosomal RNA independently and were evaluated using real-time PCR and Ultra-fast PCR (UF-PCR). As the result, Minimal detection time was reduced until 8 min 38 seconds for A. flavus and 12 min 57 seconds for A. apis under 30 cycles UF-PCR. The detections of these methods show that even 101 (100 atto gram) molecules of DNA template could be detected for Aspergillus flavus and that 103 (10 femto gram) molecules of DNA template could be detected for Ascosphaera apis. Specific UF-PCRs are not only useful to confirm pathogen-detection with naked eye, but be easy to handle a mobile PCR instrument in field. To monitoring of Chalkbrood or Stonebrood in apiary field directly, applications of on-site pathogen detection would be expected.
Keywords:
Ascospheara apis, Aspergillus flavus, Ultra-Fast PCR, Chalkbrood, Stonebrood서 론
백묵병은 Ascosphaera apis에 의하여 감염되는 진균성 질병으로 A. apis는 Maassen에 의하여 1913년 처음 발견되었다. Ascosphaera apis는 주로 유충에 주로 감염되며, 유충의 몸체를 건조시켜 작은 백묵과 같은 모양으로 죽음에 이르게 한다(Bayley, 1967; Flores et al., 1996). 또한 노제마, 바이러스, 부저병과 함께 동시감염을 일으킬 수 있는 위험요인이 많은 병원체로 거론되었다(이등, 2005).
석고병은 Aspergillus속의 진균에 의하여 발병된다. 발병을 일으키는 많은 병원체 중 가장 많은 감염을 일으키는 병원체는 Aspergillus flavus이며, 이도 주로 유충에게 감염되고 병원성 포자가 유충의 중장관에 유입하여 자라는 등 백묵병과 주요 증상과 감염 경로가 유사하다. 뿐만 아니라 딱딱하게 굳는 사체의 모습도 유사하여 육안으로 병원체의 구별이 어렵다(Bayley, 1967; Puerta et al., 1994; Deans, 1940; De Jong, 1977; Bailey, 1981). 이러한 특징 때문에, 국내외에서 석고병과 백묵병을 혼동하여 사용하였으나, 점차 병원체에 따라 석고병과 백묵병을 구분하여 사용하고 있다(이등, 2004).
국내의 서양종 꿀벌(Apis mellifera)와 동양종 꿀벌(Apis ceranae)을 대상으로, 12가지 꿀벌 질병의 발병 현황 조사는, 백묵병이 조사된 질병체들 중 4.51%를 차지하였으며, 석고병은 37.05%를 차지함을 보여주었다. 이는 국내에는 Aspergillus flavus에 의한 석고병이 보다 창궐하고 있음을 보여주는 것으로, 진균성 질병에서도 유행이 있는 것이 아닌가 의문을 들게 한다. 한편 동시감염 결과를 포함하여 석고병은 12가지의 질병 중 BQCV 다음으로 가장 높은 발병률을 보였고, 여름을 제외한 봄, 가을, 겨울 모두 높은 발병률을 보이고 있음을 보여주었다(유등, 2014).
본 연구는 석고병과 백묵병의 병원균을 대상으로 새로운 진단법을 개발하고자 하는 것으로, 기존의 실험실적 검사방법과는 달리, 현장진단의 중요성을 인식하여 Ultra-fast PCR system (UF-PCR)을 기반으로 하고자 하였다. UF-PCR은 SyBr Green과 같은 intercalating 형광 염료가 DNA 사이에 삽입되어야 형광 강도가 증가되는 것을 기본 원리로 하여, 이로써 증폭된 DNA의 양과 형광 강도는 정량적으로 비례하기에, PCR 산물의 증폭 유무를 형광의 강도로 판정하고, 이 판정이 바로 육안으로도 가능하다는 장점이 있다. 또한 일반적으로 사용하는 100μl PCR-tube가 아닌 8×10μl PCR-chip을 사용하기에 PCR의 thermocycling에서 보다 빠른 열전도가 가능하여, 보다 신속한 PCR이 가능하며 또한 8개의 다중검사를 동시에 수행할 수 있는 장점도 있다(임, 2013).
Ascosphaera apis의 검출법의 경우, real-time PCR 진단법(Lee et al., 2006)과 LAMP 진단법(Lee et al., 2011) 등이 보고되었으며, 시료의 DNA로 부터 real-time PCR 진단법에 의한 판정은 17분이 요구되었고, 현장 실험을 위한 LAMP 진단법의 경우 1시간의 검사시간이 요구되었다. real-time PCR과 LAMP진단법은 민감성에서 우수한 유전자 기반의 검출법으로 알려져 왔으나, Ascosphaera apis의 real-time PCR 검출법의 경우 102개 수준의 Ascosphaera apis 특이 DNA 분자까지 정량적 검출이 가능하였고, LAMP 진단법의 경우 Ascosphaera apis 특이 103개 수준의 DNA 분자까지 진단 가능하였다.
Aspergillus flavus 검출법은 LAMP 진단법(Lee et al., 2013)과 Real-time PCR 진단법(Bu, 2005) 등이 보고되었으며, LAMP 진단법은 1 시간, real-time PCR 진단법은약1시간30분정도가소요되는것으로알려져있다.
기 보고된 백묵병 및 석고병의 검출법들은 모두 Real-Time PCR 기반이나LAMP기반의 진단법들이다. 이들은 모두 실험실에서 사용하기에 충분히 민감한 유전자 검사법이나, 양봉현장에서 사용하기에는 검사에 소요되는 시간, 기기의 이동성 등에서 반드시 적합하다고 하기는 어려우며, 백묵병 및 석고병의 병징이 유사한 이유로 양자의 꿀벌 진균성 질병들을 동시에 판별 검출하는 방법은 요긴할 것이다.
따라서, 본 연구는 진균인 Ascosphaera apis와 Aspergillus flavus를 동시에 특이 유전자 검사를 통하여 검출할 수 있으며, 양봉현장에 보다 쉽게 적용시키기 위한 현장진단법으로써 시험 및 판정을 용이하게 하고, 또한 기존 PCR 기반의 검출법보다 빠르고 민감한 검사법을 새로이 개발하고자 하였다.
재료 및 방법
시료 수집과 DNA 확보
2015년 12월 제주대 양봉장에서 수집된 석고병 또는 백묵병 감염된 유충을 기본 질병 시료로 하였다. 해당 유충을 MagNA lyser (Roche, Korea)로 분쇄한 후, Accu prep Genomic Extraction kit (Bioneer, Korea)를 사용하여 염색체DNA를 순수분리 하였으며, 이는 분주 후, -70°C에서 보관하며 실험에 사용하였다. PCR실험에서 주형으로 사용한 Ascosphaera apis와 Aspergillus flavus의 각 18s ribosomal RNA 유전자들의 재조합 DNA체들은 경기대 유전학 실험실에서 확보하였다(이등, 2004; 이등, 2006).
Ascosphaera apis-specific PCR과 Aspergillus flavus-specific PCR를 위한 primer의 선발
UF-PCR을 위한 primer들은 설계, 주문 제작하였고 (Bionics사, Korea), 이들 primer쌍들은 먼저 Real-Time PCR로 평가하여 선발하였다. Real-Time PCR은 ExicyclerTM 96 (Bineer, Korea)을 사용하였으며, 주형은 pBX-A. apis, 2.34×106 molecules 또는 pBX-A. flavus, 2.83×106 molecules를, 각 primer후보는 5 pmole을 각기 사용하였으며, 중합 온도 50~60°C gradient real-time PCR을 통하여 최고 형광값, Threshold Cycles (Ct value)의 강약을 기준으로 각 primer쌍 후보군들을 평가하여 최종 각 병원체의 검출 프라이머로 선정하였다(Table 1).
Aspergillus flavus-specific PCR과 Ascosphaera apis-specific PCR의 검출 한계
Real-Time PCR로 각 재조합 DNA 주형을 1 ng부터 10 ag (attogram)까지 serial dilution하여 초기 주형량에 따른 검출 한계를 확인하였다. UF PCR 기기는 GenecheckerTM (Gensystem, Korea)으로, 이 역시 1 ng부터 10 ag까지 serial dilution하여 같은 실험을 진행하며 비교하였다. 사용한 PCR mix는 SsoFastTM EvaGreen ⓇSupermix (Biorad, USA)이며, Aspergillus flavus 검출에서는 한 반응 당 2.5 pmole의 프라이머를 사용하였고, Ascosphaera apis는 5 pmole의 프라이머를 각 PCR에 사용하였다. 반응액은 총 10μl이었으며, UF PCR은 94°C pre-denaturation 100초 진행 후, 94°C denaturation 10초, 57°C annealing 10초, 72°C extension 10초씩, 총 30 cycle로 수행하였다.
꿀벌 DNA 존재 하에 Aspergillus flavus-, Ascosphaera apis-specific PCR의 검출 한계
재조합 DNA 주형을 1 ng부터 10 ag까지 serial dilution하여, 알려진 어떠한 병원체에도 검출되지 않은 꿀벌의 염색체 DNA 1 ng과 함께 DNA template로 사용하였다. PCR과 결과 분석은 초기 주형량에 따른 검출 한계 실험과 동일하게 진행하였다.
Ascosphaera apis 감염 꿀벌 검체를 사용한 Ascosphaera apis-specific PCR
Real-Time PCR을 통하여 Ascosphaera apis에 감염된 것으로 확인된 꿀벌의 염색체 DNA를 순수분리하여, 그 1 ng을 주형으로 Ascosphaera apis-specific PCR을 수행하였다. 대조군으로 Nosema ceranae에 감염된 성충의 염색체DNA도 사용하였다.
한편, Aspergillus flavus-specific PCR은 Ascosphaera apis에 감염된 꿀벌, Nosema ceranae에 감염된 꿀벌을 대상으로 비특이적 PCR 증폭이 없음만을 확인하였다. PCR 조성과 조건은 초기 주형량의 검출 한계 실험과 동일하게 수행하였다.
Aspergillus flavus-specific UF-PCR의 최소 검출시간
Aspergillus flavus의 18s rRNA 유전자를 포함한 재조합 DNA, pBX-A. flavus를 각 PCR당 2.83×105 분자를 주형으로 사용하였으며, Aspergillus flavus-specific UF-PCR의 조성은 2x SsoFastTM EvaGreen ⓇSupermix(Biorad, USA), 2.5 pmole specific primers (Table 1), PCR 반응액은 총 10μl로 하였다.
Aspergillus flavus-specific UF-PCR과 Ascosphaera apis-specific UF-PCR의 온도 조건은 양자를 동시에 진단하고자 동일하게 조정하였으며, 94°C pre-denaturation 30초 진행 후, 94°C denaturation 5초-1초, 57°C annealing 5초-1초, 72°C extension 5초-1초씩, 총 30 cycle을 수행하였다.
Ascosphaera apis-specific UF-PCR의 최소 검출시간
Ascosphaera apis-specific UF-PCR의 조성은 2x SsoFastTM EvaGreen ⓇSupermix (Biorad, USA), 5 pmole specific primers (Table 1), 반응 당 총 10μl로 하였다. Ascosphaera apis-specific UF-PCR의 온도 조건은 Aspergillus flavus-specific UF-PCR의 그것과 동일하게 조성하였으나, 각 단계(step)의 시간은 검출이 가능한 시간대로 각기 축소하였다.
결과 및 고찰
Ascosphaera apis-specific PCR과 Aspergillus flavus-specific PCR를 위한 primer의 선발
Ascosphaera apis와 Aspergillus flavus의 specific UF-PCR을 위하여, 설계 제작된 primer쌍들은 먼저 Real-Time PCR로 평가하여 선발하였다. 평가에서 주형은 Ascosphaera apis의 경우, pBX-A. apis를, Aspergillus flavus의경우, pBX-A. flavus를 사용하였다.
Aspergillus flavus UF-PCR에 적용시킬 후보 primer 쌍들은 Asp312 F/R, Asp163 F/R A. flavus-199 F/R, 또는 A. flavus-163 F/R쌍들이었으며, 모두 annealing 온도 50.0~60°C 범위에서 적합한 결과를 보여 주었으나, 최적 annealing 온도를 구하는 실험 중, annealing 온도 50.0°C에서 최종 산물의 생성량, 최저의 Ct (Threshold Cycles) 등을 고려하여 Asp312 F/R쌍을 Aspergillus flavus의 UF-PCR을 위한 primer쌍으로 최종 선발하였다(Fig. 1).
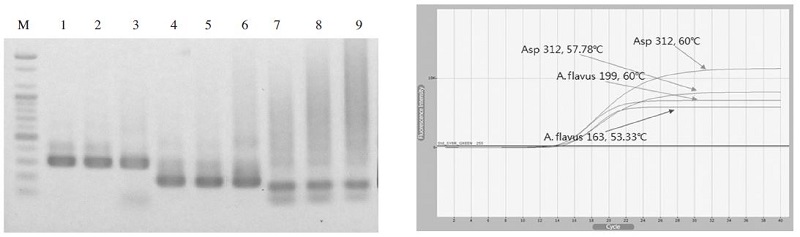
Primer selection for Aspergillus flavus-specific UF-PCR. (Left) Lane M is 100bp ladder marker (bioneer, Korea). Lane 1; PCR was performed with pBX-A. flavus using Asp 312 F/R pairs at 50°C, annealing temperature. Lane 2; same PCR at 55.56°C. Lane 3; same PCR at 60°C. Lane 4; PCR using A. flavus-199 F/R pairs at 50°C. Lane 5; same PCR at 55.56°C. Lane 6; same PCR at 60°C. Lane 7; PCR using A. flavus-163 F/R pairs at 50°C. Lane 8; same PCR at 55.56°C. Lane 9; same PCR at 60°C (Left). Fluorescence graphs of PCRs using various primer pairs (Right).
Ascosphaera apis의 UF-PCR에 적용시킬 primer 쌍들은 QP Asco F/R 또는 QP Asco F/A. apis B3쌍들이 적합한 결과를 보여 주었으나, 최적 annealing 온도를 구하는 실험 중, annealing 온도 50.0°C에서 최종 산물의 생성량, 최저의 Ct (Threshold Cycles) 등을 고려하여 QP Asco F/A. apis B3 primer쌍을 Ascosphaera apis의 UF-PCR을위한primer쌍으로 선발하였다(Fig. 2).
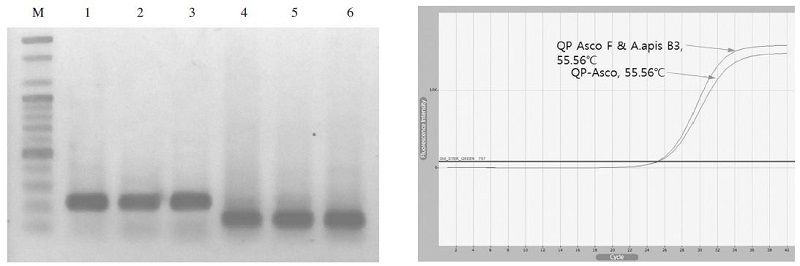
Primer selection for Ascosphaera apis-specific UF-PCR. Lane M is 100bp ladder marker (bioneer, Korea). Lane 1; PCR was performed with pBX-A.apis using QP Asco F/R pairs at 50°C, annealing temperature. Lane 2; same PCR at 55.56°C. Lane 3; same PCR at 60°C. Lane 4; PCR was performed using QP Asco F & A. apis B3 pairs at 50°C. Lane 5; same PCR at 55.56°C. Lane 6; same PCR at 60°C. PCRs using QP Asco F & A. apis B3 pairs were slightly better yields and Ct values than PCRs using QP Asco F/R (on right) in same conditions.
Aspergillus flavus-specific PCR과 Ascosphaera apis-specific PCR의 검출 한계
육안에 의한 형광의 유무 또는 강도로써, 병원체의 존재 유무 및 양을 판단해야 하는 UF-PCR에서, Aspergillus flavus 또는 Ascosphaera apis의 검출 한계를 알아보고자, 해당 주형 재조합DNA를 연속 희석하여 각각 특이 UF PCR을 각각 실시하였다.
pBX-A. flavus를 주형으로 사용한 Aspergillus flavus 특이 유전자의 검출은, 최소 2.83×101 분자(100 ag)까지도, Asp312 F/R primer쌍을 사용한 최적화된 Aspergillus flavus-specific PCR에 의해 증폭이 가능함을 보여주었다. 한편 2.83 분자(10 ag)에 대한 상기 PCR에서는 증폭이 확인되지 아니하였으며, 이는 agarose gel 전기영동과 UF-PCR의 형광으로, 쉽게 인지되었다(Fig. 3).
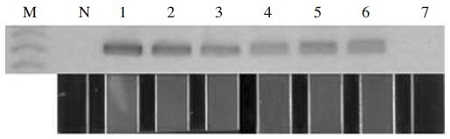
Detection limit of Aspergillus flavus-specific PCR. In agarose gel electrophoresis (upper) and panels in window (low), the existences of taget gene (pBX-A. flavus) were well recognized by Aspergillus flavus-specific UF PCR using Asp312 F/R primer pairs. Lane M, 100bp ladder marker (bioneer). Lane N, negative control without template. Lane 1, Aspergillus flavus-UF PCR with 2.83×106 molecules of target template. Lane 2, with 2.83×105, Lane 3, with 2.83×104, Lane 4, 2.83×103, Lane 5, 2.83×102, Lane 6, 2.83×101, lane 7, 2.83×101, respectively.
pBX-A. apis를 주형으로 사용한 Ascosphaera apis 특이 유전자의 검출은, 최소 2.34×101 분자(100 ag)까지도, Asco F/A. apis B3 primer쌍을 사용한 최적화된 Ascosphaera apis-specific PCR에 의해 증폭이 가능함을 보여주었다. 또한, 2.83 분자(10 ag)에 대한 상기 PCR에서 증폭은 확인되지 아니하였으며, 이 역시 전기영동과 UF-PCR의 창에서 육안으로 쉽게 인지되었다(Fig. 4).
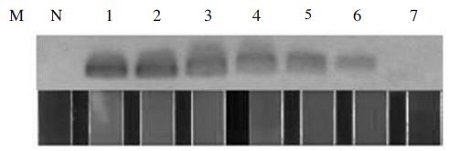
Detection limit of Ascosphaera apis-specific PCR. In agarose gel electrophoresis (upper) and panels in window (low), the existences of taget gene (pBX-A. apis) were well recognized by Ascosphaera apis-specific UF PCR using QP Asco F/A. apis B3 primer pairs. Lane M, 100bp ladder marker (bioneer). Lane N, negative control without template. Lane 1, Ascosphaera apis-UF PCR with 2.34×106 molecules of target template. Lane 2, with 2.34×105, Lane 3, with 2.34×104, Lane 4, 2.34×103, Lane 5, 2.34×102, Lane 6, 2.34×101, lane 7, 2.34×100. respectively.
꿀벌 DNA 존재 하에 Aspergillus flavus-, Ascosphaera apis-specific PCR의 검출 한계
꿀벌 병원체의 검색을 위하여 사용되는 검체는 주로 유충 또는 성충의 사체이며, 이 이유로 검체는 기본적으로 다량의 꿀벌 유래 유전체를 함유하고 있다. 이 꿀벌 유전체의 존재 하에 Aspergillus flavus-specific PCR과 Ascosphaera apis-specific PCR이 각각 특이적과 민감성을 유지하는지 검사하였다.
Asp312 F/R primer쌍을 사용한 최적화된 Aspergillus flavus-specific PCR에, 진균에 감염되지 않은 것으로 확인된 꿀벌의 1 ng chromosomal DNA과 정량화된 pBX-A. flavus를 혼합하여 각기 DNA 주형으로 사용하였다. pBX-A. flavus는 106 분자부터 100 분자까지 희석하여 각기 사용하였으며, 상기의 조건에서 Aspergillus flavus-specific PCR은, 꿀벌 DNA 혼합이 없이 실시한 같은 실험(Fig. 3)과 같은 수준으로, 최소 2.83×101 분자(100 ag)의 specific target를 특이적으로 증폭시켰다. 역시, 최소량인 2.83 분자(10 ag)의 특이 기질에 대한 상기 PCR에서 증폭은 확인되지 아니하였으며, 이는 전기영동과 UF-PCR의 창에서 육안으로 쉽게 인지되었다(Fig. 5)
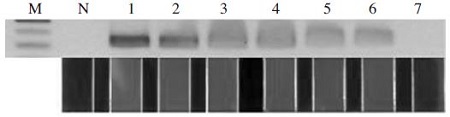
Detection limit of Aspergillus flavus-specific PCR with chromosomal DNA from honeybee. In agarose gel electrophoresis (upper) and panels in window (low), the existences of taget gene (pBX-A. flavus) were well recognized by Aspergillus flavus-specific UF PCR using Asp312 F/R primer pairs with chromosomal DNA from noninfected honeybee. Lane M, 100bp ladder marker (bioneer). Lane N, negative control without template. Lane 1, Aspergillus flavus-UF PCR with 2.83×106 molecules of pBX-A. flavus and 1 ng chromosomal DNA. Lane 2, with 2.83×105 and 1 ng chromosomal DNA, Lane 3, with 2.83×104 and 1 ng, Lane 4, 2.83×103 and 1 ng, Lane 5, 2.83×102 and 1 ng, Lane 6, 2.83×101 and 1 ng, lane 7, 2.83×100 and 1 ng, respectively.
또한, QP Asco F/A. apis B3 primer쌍을 사용한 최적화된 Ascosphaera apis-specific PCR의경우, 꿀벌의 1 ng chromosomal DNA과 정량화된 pBX-A. apis를 혼합하여 각기 DNA 주형으로 사용하였다. pBX-A. apis는 106분자부터 100 분자까지 희석하여 각기 사용하였으며, 상기의 조건에서 Ascosphaera apis-specific PCR은, 꿀벌 DNA 혼합이 없이 실시한 같은 실험(Fig. 4)과 는 달리, 최소 2.83×102 분자(1 fg)의 specific target까지만 특이적으로 증폭됨이 확인되었다. 특이 기질 2.83×101 분자(100 ag)이하는 agarose gel 전기영동에서도 증폭이 확인되지 아니하였으며, UF-PCR의 창에서는 2.83×103 분자(10 fg), 2.83×102 분자(1 fg)를 의양성으로 판정하여야 하였다(Fig. 6).
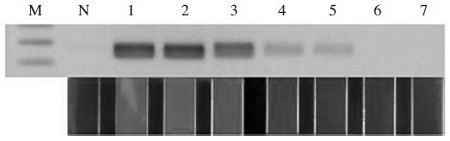
Detection limit of Ascosphaera apis-specific PCR with chromosomal DNA from honeybee. In agarose gel electrophoresis (upper) and panels in window (low), the existences of taget gene (pBX-A. apis) were well recognized by Ascosphaera apis-specific UF PCR using QP Asco F/A. apis B3 primer pairs with chromosomal DNA from noninfected honeybee. Lane M, 100bp ladder marker (bioneer). Lane N, negative control without template. Lane 1, Ascosphaera apis-UF PCR with 2.83×106 molecules of pBX-A. apis and 1 ng chromosomal DNA. Lane 2, with 2.83×105 and 1 ng chromosomal DNA, Lane 3, with 2.83×104 and 1 ng, Lane 4, 2.83×103 and 1 ng, Lane 5, 2.83×102 and 1 ng, Lane 6, 2.83×101 and 1 ng, lane 7, 2.83×100 and 1 ng, respectively.
선발된 primer쌍을 사용한 최적화된 Aspergillus flavus-specific PCR과 Ascosphaera apis-specific PCR은 30 회전의 UF PCR 조건에서 충분한 target 증폭능력을 보여주었으며, 각기 수십개 분자의 target gene으로부터 특이한 증폭이 가능함을 보여주었다. 한편, 꿀벌의 DNA가 포함된 같은 실험(Fig. 5)에서, Aspergillus flavus-specific PCR은 꿀벌의 DNA가 없는 환경에서의 실험(Fig. 3)과 거의 대등한 결과를 보여준 것에 비하여, Ascosphaera apis-specific PCR은 꿀벌의 DNA가 포함된 같은 실험(Fig. 6)에서, 꿀벌의 DNA가 없는 환경에서의 실험(Fig. 4)에 비하여 100배 수준의 낮은 증폭력을 보여주었다. 이는 제한된 target DNA의 수에 대하여 양자의 UF-PCR은 그 민감도가 다름을 보여주는 것으로, 동량의 target DNA를 동등하게 검출해 내기 위하여, 민감도가 열세인 Ascosphaera apis-specific PCR에서는, 우선 가급적 많은 양의 검체DNA를사용하고, 사용하는 각 primer의 양도 5 pmole로 상향 조정하도록 하였다,
또한, UF-PCR에서 형광강도에 의한 판정은, 같은 PCR산물의 전기영동에 의한 판독에 비하여, 극소량의 target DNA를 증폭시켰을 경우, 보다 민감하지 않음이 확인되었다. 이는 UF-PCR의 창에서 형광 유무/강도를 관찰하는 것이 agarose gel 전기영동에서 PCR 산물을 검색하는 것에 비하여 10배~100배 불리함을 보여 주는 것으로, 형광강도를 수리적으로 측정, 제시할 수 있는 보완적 방법, 또는 현장판정이후, 실험실에서 해당 PCR산물의 전기영동에 의한 확인 등도 필요한 것으로 판단되었다(Fig. 6).
Ascosphaera apis 감염 꿀벌 검체를 사용한 Ascosphaera apis-specific PCR
Ascosphaera apis에 감염된 것으로 확인된 꿀벌에서 Ascosphaera apis-specific PCR의 유용성을 확인하였다. 최적화된 Ascosphaera apis-specific PCR은 QP Asco F/A. apis B3 primer쌍을 사용한 것이며, DNA 주형으로 감염꿀벌에서 chromosomal DNA를 순수분리하여 사용하였고, 대조군으로 재조합 DNA pBX-A. apis 106 분자를 사용하였다. 또 다른 대조군으로 재조합 DNA pBX-A. flavus 106 분자를, 그리고, Nosema ceranae가 감염된 것으로 확인된 꿀벌의 chromosomal DNA를 사용하였다.
결과로써, 최적화된 Ascosphaera apis-specific PCR은 쉽게 Ascosphaera apis에 감염된 꿀벌시료에서 Ascosphaera apis의 존재를 인지하게 하였으며, Nosema ceranae 감염 꿀벌과 Aspergillus flavus-특이 염기서열 (pBX-A. flavus)에 대하여 확실한 음성을 보여주었다(Fig. 7).
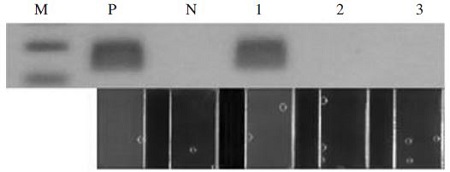
Specific detection of Ascosphaera apis-specific PCR with Ascosphaera apis-infected honeybee. In agarose gel electrophoresis (upper) and panels in window (low), the existences of Ascosphaera apis in honeybee and pBX-A.apis were well recognized by Ascosphaera apis-specific UF PCR using QP Asco F/A.apis B3 primer pairs. Lane M, 100bp ladder marker (Bioneer). Lane P, Ascosphaera apis-UF PCR with 2.34×106 molecules of pBX-A. apis. Lane N, negative control without template. Lane 1, with 1 ng chromosomal DNA from Ascosphaera apis-infected honeybee, Lane 2, with 1 ng chromosomal DNA from Nosena ceranae-infected honeybee, Lane 3, with 2.83×106 molecules of pBX-A. flavus, respectively.
한편, Aspergillus flavus-specific PCR의 유용성은 Nosema ceranae 감염 꿀벌의 chromosomal DNA와 재조합 DNA pBX-A. apis를 각기 음성 검출대상으로 하여, Asp312 F/R 쌍을 사용한 최적화된 Aspergillus flavus-specific PCR을 수행하였다. 결과는 모두 분명한 음성이었으며, 향후 Aspergillus flavus 감염 꿀벌의 검출법으로 준비를 완료하였다(자료 미제시).
Aspergillus flavus-specific UF-PCR 최소 검출시간
Aspergillus flavus의 특이 염기서열을 보유한 pBX-A. flavus 2.83×105개 분자를 사용하여 Aspergillus flavus-specific PCR 최소 검출시간을 측정하였다. Aspergillus flavus-specific UF-PCR은, pre-denaturation 30 sec 후, 각 회전 중 변성 단계 3초, 중합 단계 3초, 신장 단계 3초로 하였으며, 총 30회전의 UF-PCR을 수행하여, 완료 및 판정까지 총 10분 33초가 소요되었다. 이 실험 결과를 전기영동상과 UF-PCR의 창에서 형광으로 양성임을 확인하였으며(자료 미제시), 나아가 predenaturation 30 sec 후, 각 회전 중 변성 단계 2초, 중합 단계 1초, 신장 단계 2초로 하여, 총 30회전의 UF-PCR을 수행하였다. 완료 및 판정까지 소요된 시간은 총 8분 38초이 결과를 agarose gel 전기영동과 UF PCR의 형광에서 모두 양성임을 확인할 수 있었다(Fig. 8).
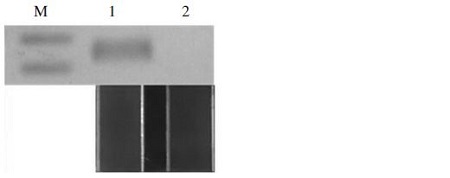
Minimum time of detection using Aspergillus flavus-specific UF-PCR. UF-PCR were performed using Asp312 F/R primer pairs, after 30 sec pre-denaturation, under conditions of 2 sec denaturation step, 1 sec annealing step, 2 sec extension step in each cycles. Total 30 cycles were completed on 8 min 38 sec. Lane M, 100bp ladder marker (bioneer). Lane 1, Aspergillus flavus-UF PCR with 2.83×105 molecules of pBX-A. flavus. Lane N, negative control without template. In agarose gel electrophoresis (upper) and panels in window (low), the amplifications using Aspergillus flavus-specific UF PCR were well recognized by naked eyes.
Ascosphaera apis-specific PCR 최소 검출시간
Ascosphaera apis의 특이 염기서열을 보유한 pBX-A. apis 2.34×105개 분자를 사용하여 Ascosphaera apis-specific PCR 최소 검출시간을 측정하였다. Ascosphaera apis-specific UF-PCR은, pre-denaturation 30 sec 후, 각 회전 중 변성 단계 5초, 중합 단계 5초, 신장 단계 5초로 하였으며, 총 30회전의 UF-PCR을 수행하여, 완료 및 판정까지 총 12분 57초가 소요되었다. 이 실험 결과를 전기영동상과 UF-PCR의 창에서 형광으로 모두 양성임을 확인할 수 있었다(Fig. 9).

Minimum time of detection using Ascosphaera apis-specific UF-PCR. UF-PCR were performed using QP Asco F/A. apis B3 primer pairs, after 30 sec pre-denaturation, under conditions of 5 sec denaturation step, 5 sec annealing step, 5 sec extension step in each cycles. Total 30 cycles were completed on 12 min 57 sec. Lane M, 100bp ladder marker (bioneer). Lane 1, Ascosphaera apis-UF PCR with 2.34×105 molecules of pBX-A. apis. Lane N, negative control without template. In agarose gel electrophoresis (upper) and panels in window (low), the amplifications using Ascosphaera apis-specific UF PCR were well recognized by naked eyes.
나아가 pre-denaturation 30 sec 후, 각 회전 중 변성 단계 3초, 중합 단계 3초, 신장 단계 3초로 하여, 총 30회전의 UF-PCR을 수행하였으며, 완료 및 판정까지 소요된 시간은 총 10분 33초이었다. 이 PCR 결과는 agarose gel 전기영동에서 확인되었으나 UF PCR의 형광창에서는 양성으로 판정할 수 없었다(자료 미제시).
병원체의 검출법 중 최소의 검출 시간은 현장 실험이 가능한가를 판단할 수 있는 중요한 기준이다. 본 연구에서 확인된, 초기 변성 30초를 포함한, 변성 단계 2초, 중합 단계 1초, 신장 단계 2초의 30 cycles PCR은 105개 분자의 target DNA에 대하여 총 8분 38초만에 Aspergillus flavus 특이 검출이 가능함을 보여주었다. 한편, Ascosphaera apis 검출의 경우, 초기 변성 30초를 포함하여, 5초 변성 단계, 5초 중합 단계, 5초 신장 단계, 총 30 cycles PCR을 총 12초 57분 만에 유효하게 완료하였다. 이 수준의 검사시간이 소요된다면, 이실험법은 현장 실험법으로 충분히 유용한 것이라 판단되며, 유전자 진단법이기에 어떤 다른 진단법보다 우월한 정확성 및 민감성을 또한 입증할 수 있을 것이다.
현장 진단은, 현장에서 병원체의 확인 시, 즉각적인 대응에 대한 판단이 가능할 것이며, 미동정 검체를 실험실로 우송하는 번거롭고 위험한 과정이 생략되기에, 질병의 전파를 최소화 할 수 있다. 백묵병과 석고병에 한정된 본 연구의 결과는, Ultra-fast PCR (UF-PCR)에 기반을 둔 것으로, 이 실험법이 유전자 진단법으로 많은 다른 병원체에 대한 현장 실험법 등으로 응용되기를 또한 기대한다.
적 요
꿀벌의 진균성 질병인 백묵병과 석고병 진단을 위한 Ascosphaera apis와 Aspergillus flavus의 신속한 검출법을 새로이 개발하였다. 이는 초고속 PCR (Ultra-fast PCR; UF-PCR)에 기반한 검출법으로써 A. flavus와 A. apis의 18s ribosomal RNA유전자를 특이적으로 검출하는 프라이머 세트들을 선정하고 real time PCR과 UF-PCR를 이용하여 개발된 것이다. 결과로써, Aspergillus flavus는 8분 38초만에 그 존재여부를 판정할 수 있었고, Ascosphaera apis는 12분 57초만에 판정할 수 있었다. 또한, 이 실험법으로 Aspergillus flavus의 초기 주형 101 DNA까지 존재를 확인할 수 있었으며, Ascosphaera apis는 초기 주형 103 DNA까지 존재가 확인되었다. 본 실험법은 전기영동의 과정 없이 육안으로 결과를 확인할 수 있다는 장점과 이동이 용이하다는 점에서 병원체 검출을 위한 현장 실험법으로 매우 유용하리라 판단된다. 이러한 실험법의 장점들은 석고병과 백묵병 뿐 아니라 다양한 병원체의 현장 검출법 개발에 널리 적용될 수 있을 것이라 기대된다.
Acknowledgments
본 연구는 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 수출전략기술개발사업(115067-02)과 첨단생산기술개발사업(115058-02, 115102-03) 그리고 농생명산업기술개발사업(312027-03)의 지원을 받아 수행되었으며, 저자 일동은 이에 감사하는 바입니다.
References
- 이보람, 유미선, Van PhuNguyen, 노지나, 윤병수, (2011), Loop-mediated Isothermal Amplification (LAMP) 법을 이용한 백묵병 원인균 Ascosphera apis의 검출법 개발, 한국양봉학회지, 26, p103-111.
- 이혜민, 하정순, 조용호, 남성희, 윤병수, (2004), 꿀벌 진균성 질병의 신속 확인을 위한 Ascosphera apis, Aspergillus flavus의 PCR 검출법, 한국양봉학회지, 19, p139-148.
- 이혜민, 이도부, 한상훈, 남성희, 임윤규, 윤병수, (2005), Real-Time PCR 을 이용한 백묵병 원인균 Ascosphera apis의 신속 검출, 한국양봉학회지, 20, p109-116.
- 이혜민, 유미선, 김을환, 이동우, 한상훈, 윤병수, (2006), 백묵병 원인균 Ascosphaera apis의 Quick Real-time PCR 검출법, 한국양봉학회지, 21, p107-112.
-
Bailey, L., (1981), Honey bee pathology, Ann. Rev. Entomol, 13, p191-212.
[https://doi.org/10.1146/annurev.en.13.010168.001203]

- Bailey, L., and B.V. Ball, (1991), Honey Bee Pathology, Academic Press, London.
- Bailey, L., (1967), The effect of temperature on the pathogenicity of the fungus Ascosphaera apis for larvae of the honeybee, Apis mellifera, Insect Pathology and Microbial Control, p162-167.
-
Bu, R., R.K. Sathiapalan, M.M. Ibrahim, I. Al-Mohsen, E. Almodavar, M.I. Gutierrez, and K. Bhatia, (2005), Monochrome Light Cycler PCR assay for detection and quantification of five common species of Candida and Aspergillus, J. medical microbiology, 54, p243-248.
[https://doi.org/10.1099/jmm.0.45856-0]

- Deans, A.S.C., (1940), Chalk brood, Bee World, 21, p46-51.
- De Jong, D., (1977), A study of chalk brood disease of honey bee, Cornell University, Ithaca, USA, M. Sc. Thersis.
-
Flores, J.M., et al , (1996), Effect of temperature and humidity of sealed brood on chalkbrood development under controlled conditions, Apidologie, 27, p185-192.
[https://doi.org/10.1051/apido:19960401]

-
Lee, J.S., S.J. Yong, H.Y. Lim, and B.S. Yoon, (2013), A Simple and Sensitive Gene-Based Diagnosis of Aspergillus flavus by Loop-Mediated Isothermal Amplification in Honeybee, J. Apiculture, 30, p53-59.
[https://doi.org/10.17519/apiculture.2015.04.30.1.53]

- Lim, H.Y., (2013), Development of Novel Rapid Detection Method for Deformed Wing Virus (DWV) using Ultra-Fast High-Performance PCR (UF-HP PCR), J. Apiculture, 28, p237-244.
-
Mariano Higes, Raquel Martín-Hernández, Crístina Botias, Encarna Garrido Bailón, Amelia V. González-Porto, Laura Barrios, M. Jesús del Nozal, José L. Bernal, Juan J. Jiménez, Pilar García Palencia, Aránzazu Meana, (2008), How natural infection by Nosema ceranae causes honeybee colony collapse, Environmental microbiology, 10, p2659-2669.
[https://doi.org/10.1111/j.1462-2920.2008.01687.x]

-
Puerta, F., Flores, J.M., Bustos, M., Padilla, F., and Campano, F., (1994), Chalkbrood development in honeybee brood under controlled conditions, Apidologie, 25, p540-546.
[https://doi.org/10.1051/apido:19940604]

- Yoo, M.S., H.J. Seo, H.N. Jung, W.R. Bae, H.S. Lee, S.W. Kang, and Y.S. Cho, (2014), Center for Honeybee Disease Control, Korea.
