
역전사 실시간 Recombinase Polymerase Amplification (RT/RT RPA)에 의한 꿀벌 Black Queen Cell Virus의 신속 검출
Abstract
Black Queen Cell Virus (BQCV) is one of pathogenic virus in honeybee which could be detected using reverse transcription real-time PCR (RT/RT-PCR). In this study, for rapid detection of BQCV, Recombinase Polymerase Amplification (RPA) was applied, and BQCV-specific reverse transcription real-time RPA (RT/RT-RPA) method was newly developed based on BQCV-specific RT/RT PCR. The Real-time RPA (RT-RPA) was performed at 37°C isothermal condition for 40 minutes. During the experiments, specific DNA amplifications were real-timely monitored using fluorescent detector. BQCV-specific DNA amplification could be detected from 3 min 26 sec after RPA reaction with specific DNA templates by RT-RPA, while 41 min 42 sec was required by qRT-PCR with same quantities of initial templates. With generated cDNA from BQCV-infected honeybee, specific DNA amplification was recognized at 4 min 18 sec using RT-RPA, however, 66 min 5 sec was needed using Real-time PCR. Moreover, with reverse transcriptase and RPA solution, BQCV-specific DNA amplification could be detected at 8 min 36 sec from total RNA of BQCV-infected honeybee using one-step Reverse Transcription/Real-Time RPA (RT/RT-RPA).
Keywords:
Black queen cell virus, BQCV, Virus detection, Recombinase Polymerase Amplification, RPA, Real-time RPA서 론
꿀벌은 바이러스, 박테리아, 진균 등 많은 감염성 병원체에 의하여 공격을 받고 있으며, 그중 바이러스성 병원체는 많은 피해를 야기하고 있으나, 이들에 대한 지식이 매우 부족하여 적절한 대비책을 세우기 어려운 상황이다. 현재 꿀벌의 바이러스성 병원체는 Acute Bee Paralysis Virus (ABPV), Chronic Bee Paralysis Virus (CBPV), Sacbrood Virus (SBV), Kashmir Bee Virus (KBV), Deformed Wing Virus (DWV), Black Queen Cell Virus (BQCV) 등, 총18종이 알려져 있다(Tentcheva et al., 2004; Berényi et al., 2006).
Black Queen Cell Virus (BQCV)는 유럽과 미국에서 꿀벌 붕괴의 원인체로 추론된, 주요 병원체이며(Chen et al., 2007), 분류학상 family Dicistroviridae에 속하고, 종전까지 피코르나 바이러스 속으로 분류되었으나, 최근에 새로운 크리파바이러스 속으로 재분류 되었다(Mayo, 2002). BQCV의 RNA 유전체는 두 개의 ORF로 구성되어 있으며, replicase로 추정되는 5‘-근접 ORF와 capsid protein인 3’-근접 ORF로 이루어져 있다(Leat et al., 2000). BQCV는 한국에서도 꿀벌 전반에 많이 발견되는 바이러스로 확인되었다(Yoo et al., 2008; Kang et al., 2012).
BQCV에 대한 신속한 검출법의 개발은 이 바이러스의 감염 여부 판단과 이 바이러스의 제어를 위하여 반드시 선행되어야 하는 전략이며, Multiplex reverse transcription-PCR (Grabensteiner et al., 2007), One-step real-time PCR (Kukielka et al., 2008)과 같이BQCV의 정확하고 신속한 진단을 위한 여러 가지 핵산 기반 방법들이 개발되어 왔다. 이 실시간 PCR 방법은 정량 실시간 PCR (quantitative Real-Time PCR; qRT-PCR)의 발전과 연합되어 BQCV 뿐만 아니라 다른 꿀벌 바이러스를 탐색하는데 가장 민감하고 정확한 실험법으로 인정되어 왔다(Yoo et al., 2008).
그러나, qRT-PCR은 일반적으로 검사시간에 수 시간이 소요되며, 정교한 thermo cycling 기기를 필요로 하기에, 양봉현장에서 요구되는, 즉석 현장실험을 수행하기에는 적합하지 못한 면이 있다. 이런 실험방법상 단점을 극복하기 위하여 PCR 검사시간을 극단적으로 단축시킨 초고속 PCR (Ultra-Rapid PCR; UR-PCR) 또는 초신속 PCR (Ultra-Fast PCR; UF-PCR) 등이 개발되어, 30회전의 PCR을 5분~8분에 완료시키는 진보를 보이고 있다(Yoo et al., 2011; Giang et al., 2015).
Recombinase Polymerase Amplification (RPA)은, PCR과는 달리, 등온에서 특정 유전자의 염기서열을 증폭시키는 실험법이며(Piepenburg et al., 2006), 보다 빠른 유전자 증폭을 기대할 수 있는 실험법으로 개발이 진행되고 있다. RPA는 recombinase, single strand binding protein (SSBP), strand displacing DNA polymerase, 그리고 specific primer 등의 조성으로, 보통 37°C 등온에서 반응이 진행되며, 반응시간은 30분 내외가 추천되고 있다. 근래 이 RPA에 의한 다양한 병원체의 검색법들이 계속 보고되고 있으며, 우수한 유전자 진단법으로 점차 적용범위를 넓혀 나아가고 있다. 그러나, 꿀벌 병원성 바이러스의 검출에 대하여 RPA 기술이 접목된 것은 찾아 볼 수 없었으며, 또한 꿀벌 병원체 전반에 대하여도RPA 기반 실험법이 알려진 것은 없었다.
따라서, 본 연구는 RPA 기술에 기반을 둔 꿀벌 바이러스 BQCV의 검출법을 새로이 개발하고자 하였으며, 이 기술을 꿀벌 바이러스 전체, 나아가 꿀벌 병원체 전체에 대한 검출법으로 적용시킬 수 있는지, 그 기술적 유용성을 평가해 보고자 하였다.
재료 및 방법
꿀벌 병성 시료
본 연구에서 사용된 모든 꿀벌 시료는 국내산 서양종 꿀벌인 Apis mellifera이며, 2015년에 한국의 양봉농가로부터 채집되어 한국꿀벌질병연구소에 위탁된 성충 사체이다. 병성 시료는 공급처에 따라 각각 50mL conical tube에 담아, 접수 즉시 -70°C에 보관되었다. BQCV-specific PCR 결과에 따라, BQCV 감염이 확인된 시료를 선발하여 본 연구에 사용하였다. 또한 DWV (Deformed Wing Virus)-specific PCR 결과에 따라, DWV 감염이 확인된 꿀벌도 선발하여 사용하였다.
전체 RNA의 순수분리 및 cDNA 제작
수집한 시료 중 두 마리의 성충 꿀벌을 임의로 선택하였으며, MagNA Lyser green Beads (Roche, Switzerland)를 사용하여 꿀벌을 분쇄하였다. 전체 RNA는 AllspinTM (GeneAll, Korea)를 사용하여 추출하였다. 추출된 전체 RNA는 Biophotometer (Eppendorf, Germany)를 사용하여 핵산의 농도를 측정하였으며, 1μg의 전체 RNA를 주형으로 AccuPowerⓇ RT Premix (Bioneer, Korea)를 사용하여 cDNA를 합성하여 -70°C에 보관하였다. 본 연구에서는 합성 직후의 cDNA를 real-time RPA와 real-time PCR 실험의 주형으로 이용하였다.
재조합 DNA 주형의 준비 및 quantitative Real-Time PCR (qRT-PCR)
pGEM-BQCV-VP3는BQCV의 VP3유전자가 탑재된 플라스미드이다(Giang et al., 2015). 이는 pGEM-3Zf(+) vector (Promega, USA)에 BQCV의 VP3 유전자를 클론화한 것이며, 이는 대장균 DH5α에 보관되었다. 이 재조합 DNA의 clone에서 DNA-spinTM Plasmid DNA Purification Kit (iNtRON Biotechnology, Korea)를 사용하여 pGEM-BQCV-VP3를 순수 분리하였으며, Biophotometer (Eppendorf, Germany)를 이용해 DNA농도를 측정하고, 추출된 DNA는 -20°C에서 보관하며 사용하였다.
qRT-PCR은 SYBR green의 형광을 이용하였으며 모든 반응은 ExicyclerTM Quantitative Thermal Block (Bioneer, Korea)을 사용하여 수행하였다. 총 반응액은 20μl로 하였고, 조성은 HiPi Real-Time PCR 2x Master Mix, 1x SYBR green (Elpisbio, Korea), 10 pmole의 프라이머, 그리고 주형으로써 pGEM-BQCV-VP3 DNA 또는cDNA를 사용하였다.
프라이머 설계 및 제작
BQCV의 구조 단백질 중 하나인 VP3 유전자를 증폭하기 위해 한 쌍의 프라이머를 사용하였다. GenBank database (Accession No. KR074231)에 따라 설계된, BQCV-VP3-F1 (forward)와 BQCV-VP3-R1 (reverse)은 각각 KR074231의 염기서열 중 139 bp부터 269 bp의 위치에 결합되는 것으로, 131 bp의 PCR 산물이 제작되도록 디자인 하였으며, 바이오닉스(Bionics, Korea)사에 의뢰하여 제작하였다(Table 1).
Real-time Recombinase Polymerase amplification (qRT-RPA)
RT-RPA는 기본적으로 Twist AmpⓇ Basic kit (TwistDx, UK)를 사용하여 수행하였다. RPA reaction mix는 kit의 내용물인 freeze-dried reaction과 280mM MgAc를 제외하고, 각 반응 당 각 2.4 pmole의 프라이머 (BQCV-VP3-F1/R1), 최종 1x rehydration buffer, 최종 1x SYBR green I이 되도록 하였고, vortex로 가볍게 혼합한 후, 이를 freeze-dried reaction이 있는 tube로 옮기어 pipetting으로 내용물을 혼합하였다(Table 2). 혼합액은 qRT-PCR용 200μl white tube로 옮겼으며, 각 tube에 주형으로 107 molecules에 해당하는 1μl pGEM-BQCV-VP3를 첨가하였다. RPA반응은 280mM의 MgAc를 첨가한 후, 37°C의 등온 조건에서 시작하여, 1분을 1 cycle로 하여, 반응시간 40분이면 40 cycle로 나타나도록 하였다(Table 3). 실시간 DNA 증폭을 측정하기 위하여 SYBR green I (Elpisbio, Korea)을 첨가한 후 ExicyclerTM Quantitative Thermal Block (Bioneer, Korea)에서RPA 반응을 수행하였다.
One-step Reverse transcription RT-RPA (RT/RT-RPA)
cDNA의 합성과 RT-RPA 반응을 동시에 진행하고자, 추출된 전체 RNA를 주형으로 Reverse transcription과 RT-RPA가 같이 진행될 수 있도록 one-step 반응액을 조성하였다. 기본적으로 AccuPowerⓇ RT Premix (Bioneer, Korea)를 사용하였으며, 1μg의 전체 RNA, 100 pmoles Oligo dT 를 70°C에서 5분간 pre-denaturation 시켰으며, 반응 후 이 용액을, RPA-kit중 freeze-dried reaction (dried pellet)에 옮기어 pippet으로 잘 혼합하였다. 이 혼합액에 RPA kit 중 280mM의 MgAc를 첨가하고, 37°C의 등온 조건에서 RPA반응을 시작하였다. 이로써 one-step reverse transcription RPA는 cDNA 합성과 DNA의 증폭이 동시에 진행되도록 하였으며, 총 반응시간은 40분으로 형광에 의한 DNA 증폭을 실시간 모니터링 하였다.
결과 및 고찰
Real-Time RPA와 Real-Time PCR을 이용한 BQCV 특이 유전자의 검출
Real-Time PCR과 Real-Time RPA의 모든 반응에서 주형은 재조합DNA인pGEM-BQCV-VP3를 사용하였으며, 모두 동일한 농도의DNA를 사용하여 수행되었다. 사용된 프라이머(Table 1)은 양자 모두 같은 염기서열인BQCV-VP3-F1과BQCV-VP3-R1이었다.
최적화된 Real-Time PCR의 조건은, 94°C에서 30초간 Pre-denaturation을 진행한 후, denaturaion 94°C, 15초, Annealing 62°C, 15초 그리고 Extension 72°C, 15초로 Total 35 cycle을 진행하였다. Real-Time RPA는 37°C 등온에서 40 cycle로 진행하였다(Table 3).
Real time RPA와 Real time PCR에서BQCV-VP3 특이 유전자의 증폭이 형광 강도와 melting 온도 분석에 의해 확인되었으며, agarose gel 전기영동에서 양자 모두 예상되었던 131 bp의 DNA 산물을 생성하였다(Fig. 1, Fig. 2, Fig. 3).
Real-time RPA에는 3분 26초(3.44 cycles)에서 DNA 증폭이 확인되었으며, 40분의 전체 반응 시간 내 지속적인 증가양상을 보였다. 반면, template없이 진행된 RPA반응은 40분 전체 반응 시간 내 DNA증폭이 확인되지 않았다. Final Fluorescent value는 40분(40 cycles) 반응 후 5301의 값을 보였으며, 한편 Negative에서는 167로 나타나 두 값의 상당한 차이가 있음을 알 수 있었다(Fig. 1).
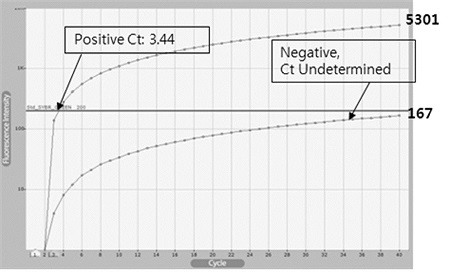
Fluorescence curves of specific DNA amplification using real-time RPA. Real-time RPAs were performed with 1 ng of pGEM-BQCV-VP3 as template (Positive) or without template (Negative). Fluorescence based on DNA amplification was rapidly increased and passed through the base line on 3.44 cycles, on the time of 3 min 26 sec after beginning of Positive reaction. However, fluorescence from Negative was not reached the base line until 40 cycles (40 min).
반면, Real-time PCR은 16.22 Ct 값(반응시작 후 41분 42초)부터 DNA 증폭을 확인 할 수 있었으며, 26 cycles(반응시작 후 64분 17초) 지점에서 5514의 최고 형광 값이 측정된 이후 약간의 형광 값 감소가 관찰되어, 최종 35 cycles(반응시작 후 90분)에서 4988의 최종 형광값이 측정되었다. 반면, template없이 진행된 Real-time PCR은 30.88 Ct 값(반응시작 후 79분 24초) 이후, DNA의 증폭이 인정되었으며, 최종 35 cycles(반응시작 후 90분)에서 1521의 최종 형광값이 측정되었다(Fig. 2).
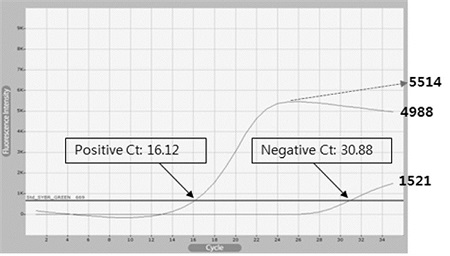
Fluorescence curves of specific DNA amplification using real-time PCR. Real-time PCRs were performed with 1 ng of pGEM-BQCV-VP3 as template (Positive) or without template (Negative). Fluorescence based on DNA amplification was passed through the base line on 16.12 cycles, on the time of 41 min 42 sec after beginning of Positive reaction. However, fluorescence from Negative was passed through the base line on 30.88 cycles, on the time of 79 min 24 sec.
Real time RPA와 Real time PCR의 두 반응은, 종료 이후, 증폭된 DNA를 1.5% Agarose gel 상에서 전개하여 증폭된 DNA의 크기와 양상을 확인하였다. Real-time PCR의 증폭 산물은 예상하였던 131bp의 위치에서 정확하게 확인되었으며(Positive), template없이 같은 PCR을 진행한 negative 반응에서는 DNA 증폭 산물이 형성되지 않았다(Fig. 3A).
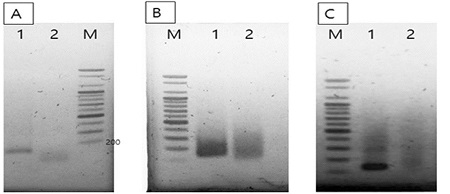
Agarose gel electrophoresis of amplified DNAs using RPA or PCR. Panel A. PCR products from real-time PCRs in Fig. 2. Panel B. RPA products before purification from real-time RPAs in Fig. 1. Panel C. RPA products after purification. In each panel, Lane 1 is the amplified product with 1 ng of pGEM-BQCV-VP3 as template, and lane 2 is Negative reaction without template. The expected size of BQCV-DNA was 131 bp long.
RPA에 의한 DNA 증폭 산물은 예상된 131 bp 부위뿐 아니라 그 상부 쪽에 다양한 끌린 듯한 DNA들이 관찰되었다. 또한, 주형없이 같은 RPA를 진행한 Negative 반응에서 증폭된 DNA의 양은 크게 감소된 것으로 나타났으나, 같은 형태의 전기영동 상을 보여주었다(Fig. 3B). 그러나 이 반응 산물들은 프라이머와 unspecific single strand DNA를 제거하는 정제과정 이후 전기 영동상에서 131 bp 크기의 정확한 DNA band를 관찰할 수 있었으며, 주형 없이 진행된 negative 반응에서는 정확한 크기의 DNA 산물을 관찰할 수 없었다. 이 전기영동의 결과로써 RPA에 의해 증폭된DNA 산물은, 생성된unspecific single strand DNA 등과 함께 RPA에서 사용되는 single strand binding protein (SSBP) 등 여러 단백질에 의한 gel mobility shift 현상이 함께 나타난 것으로 추론되었다(Fig. 3C).
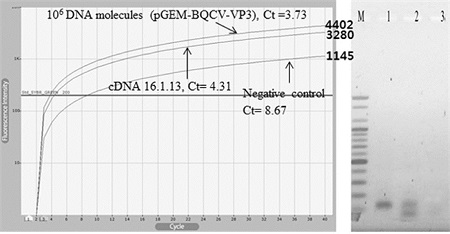
Amplification of BQCV-VP3 sequences from cDNA using real-time RPA. cDNA from BQCV-infected honeybee was used for the template of Real-time RPA. As positive or negative control, with 106 copies of pGEM-BQCV-VP3 or without template, Real-time RPAs were performed, respectively. Ct values and final fluorescence values were estimated for each RPA, 3.73 and 4402 (Positive), 4.31 and 3280 (cDNA), 8.67 and 1145 (Negative), respectively. In right, BQCV-specific 131 bp long products were only observed on lane 1 (Positive) and lane 2 (cDNA).
Real-time RPA와 Real-time PCR의 두 방법은 모두 형광강도를 측정하여, 특이 염기서열의 증폭을 정량적으로 모니터링 할 수 있음을 확인하였으며, Real-time RPA는 Real-time PCR 보다 빠르게 DNA 증폭이 가능함을 인지할 수 있었다. 또한 전기영동을 통해 양자 모두 주어진 프라이머에 따라 정확하게 특정 염기서 열만을 증폭시킴을 또한 확인할 수 있었다.
cDNA로부터 Real-time PCR와 Real-time RPA를 이용한 BQCV의 검출
Real-time PCR과 Real-time RPA 반응에서, 주형으로 cDNA를 사용하였다. BQCV에 감염된 것으로 확인된 꿀벌로부터 전체 RNA를 추출하고, 그 중 1μg의 RNA를 사용하여 Reverse transcriptase 반응을 진행하였으며, 이로써 주형으로 사용할 cDNA를 확보하였다. RPA와 PCR 두 반응 모두에서 동일한 양의 cDNA를 주형으로 동일한 프라이머(Table 1)을 사용하였다.
결과로써, cDNA를 사용한 Real-time RPA 반응에서는 4.31 cycles(반응 시작 후 4분 18초)부터 DNA의 증폭이 인지되었으며, 40분의 전체 반응 시간 내 지속적인 증가 양상을 보였다. 대조군으로 사용한 106 분자의 pGEM-BQCV-VP3에 대한 RPA 반응에서는 3.73 cycles(반응 시작 후3분 43초)에서 DNA의 증폭이 확인되었다. 그러나 negative 반응에서는 앞의 두RPA 반응의 결과보다 늦은 8.67 cycles(반응 시작 후 8분 40초)부터DNA 증폭이 인식되었다.
앞의 3개 RPA의 형광그래프는 매우 유사한 형광 값 증가의 모양을 보였으며, 3자의 차이는 Ct value와 최종 형광 값들 이었다. 각 반응의 최종 형광 값들은 각각 4402, 3280, 1145로 측정되었으며, 이는 각 Ct 값인 3.73, 4.31, 8.67 cycles와 매우 유사한 것이었다. 최종 형광값들과 각 Threshold cycles (Ct 값)들을 회귀분석 하였으며, 회귀상수(Regression coefficience; R2)는 0.9443인 것으로 계산되었다.
DNA 증폭 반응 종료 이후, 각 증폭 산물을 1.5% agarose gel로 전기영동하였으며, cDNA와 pGEM-BQCV-VP3을 주형으로 사용한 Real-time RPA 반응에서 증폭 산물은 모두 예상 크기인 131bp에서 나타남을 확인하였다(Fig. 4).
한편, BQCV에 감염된 꿀벌로부터 제작된cDNA는, RT-RPA의 경우와 같이, Real-time PCR을 이용하여, BQCV-specific sequence를 증폭시켰으며, Ct 값 24.09 cycles(반응시작 후 66분 5초)에서부터 DNA 증폭을 인지할 수 있었다. 측정된 형광 값은 40 cycles까지 지속적인 증가세를 보였으며, 최종 형광값은 5173으로 측정되었다. 반면, 주형 없이 수행된 negative 반응은 전혀DNA 증폭이 확인되지 않았다(Fig. 5).
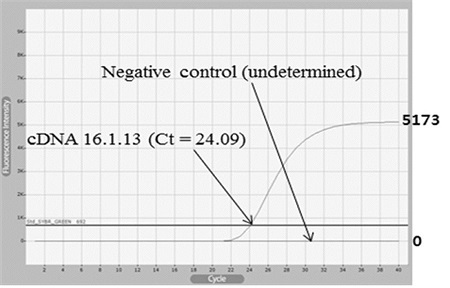
Amplification of BQCV-VP3 sequences from cDNA using real-time PCR. cDNA from BQCV-infected honeybee was used for the template of Real-time PCR. As negative control, Real-time RPA without template were also performed. 24.09 cycles (Ct values) and 5173 final fluorescence values were estimated, only in RPA with cDNA.
Real-time PCR에 의하여 cDNA로부터 증폭시킨 PCR 산물은 융점분석(Melting temperature analysis)를 통하여 Tm(temperature of mid-point)을 측정하였다. 측정된 Tm 값은 80.5°C이었으며, 이는 재조합 DNA pGEM-BQCV-VP3를 주형으로 BQCV-VP3-F1/R1 프라이머를 이용한 PCR 산물에서 얻은 Tm 값과 일치하는 것으로, cDNA로부터 동일한 특이 염기서열을 증폭시켰음을 보여주었다(Fig. 6).
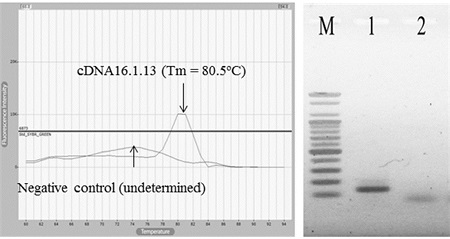
Melting temperature analysis and gel electrophoresis after real-time PCR. (Left) The temperature of mid-point (Tm) was measured at 80.5°C in only PCR with cDNA, as same as expected. (Right) In agarose gel electrophoresis, expected 131bp long DNA was only observed in PCR with cDNA (lane 1).
BQCV에 감염된 꿀벌로부터 제작된 cDNA를 주형으로, 각각 Real-time RPA와 Real-time PCR을 수행하였을 때 목표로 한BQCV의 131 bp의 VP3 gene이 정확히 증폭됨을 확인하였다. 양자 간의 반응 시간을 비교하면, 특이 염기서열의 증폭 시점은 Real-time RPA의 경우 반응 시작 후 4분 18초부터 목표로 특이 염기서열이 증폭됨을 인지할 수 있었으나, Real-time PCR의 경우, 반응 시작 후 66분 5초부터 이를 인지할 수 있었다. 양자간의 차이는 61분 47초에 이르며, 이 차이는 보다 빠른 특이 유전자 검출을 위하여, RPA 방법의 활용이 유망함을 시사해 준다.
특정 유전자 검출에 대한 Real-time RPA와 Real-time PCR의 특이성 비교
Real-time RPA와 Real-time PCR 양자 간에, 꿀벌에서 특정 바이러스를 검출해 내는 특이성(specificity)에 차이가 있는지를 비교하였다. 이를 위하여 BQCV에 감염된 꿀벌과 DWV(Deformed Wing Virus)에 감염된 꿀벌로부터 각각 전체RNA를 순수 분리하였으며, 각각 cDNA를 제작하여 각각 두 가지 분석방법의 주형으로 사용하였다. 정확한 비교를 위하여 주형으로 사용한 BQCV cDNA와 DWV cDNA는 각각 동량을 사용하였으며, 사용한 프라이머는 BQCV의 VP3유전자에 특이적인BQCV-VP3-F1/R1이었다(Table 1).
Real-time RPA에서 BQCV cDNA를 주형으로 한 반응에서 5분 53초(5.89 cycles)부터 DNA 증폭이 확인되었으며, DWV cDNA를 주형으로 한 반응에서 반응시작 후 10분 15초(10.25 cycles) 부터 DNA 증폭이 확인되었다. 한편, 주형없이 수행한 같은 RPA 반응에서는 16분 51초(16.85 cycles)부터 DNA의 증폭이 인지되었다(Fig. 7).
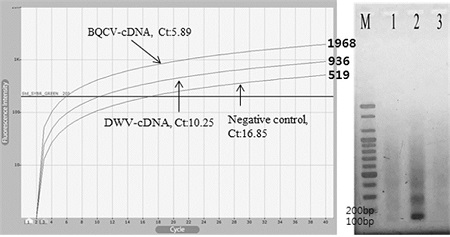
Specificity of Real-time RPAs with cDNAs generated from BQCV- or DWV-infected honeybee. With BQCV-cDNA using BQCV-specific real-time RPA, BQCV-specific DNA amplification was recognized at 5 min 53 sec (5.89 cycles). With DWV-cDNA using BQCV-specific real-time RPA, unspecific DNA amplification was recognized at 10 min 15 sec (10.25 cycles). Without templates using BQCV-specific realtime RPA, un-specific DNA amplification was also recognized at 16 min 51 sec (16.85 cycles). (Right), BQCV-specfic DNA, 131 bp long, was well observed by agarose electrophoresis (lane 2). Un-specifc DNAs were also recognized by RPAs with DWV-cDNA (lane 1), or without templates (lane 3), respectively.
RT-RPA의 40분 반응 후, 3개의 반응에 대한 최종 형광값은 각각 1968, 936, 519로 나타났으며, 이는 각 반응의 Ct value인 5.89, 10.25, 16.85 cycle와 함께 초기 주형(initial template)의 정량을 추구하기 위한 주요 척도가 될 수 있을 것으로 사료되었다.
Real-time RPA 반응 종료 이후, 각 증폭산물을 1.5% agarose gel에서 확인한 결과, BQCV cDNA를 주형으로한 반응에서만 131 bp의 BQCV 특이적인 DNA 밴드가 확인되었으며, DWV cDNA와 주형 없이 진행된 반응에서는 131 bp의 BQCV 특이 DNA가 발견되지 아니하였다. 이는 후자의 반응에서 비특이적 DNA를 상대적으로 약하나마 증폭시켰고, 이들의 형광이 감지된 것으로 해석하였다. 이는 또한, DNA 증폭을 형광의 강도로 모니터링하는 형광상과 전기영동상은 정확히 일치하지 않음을 보여주는 것으로 향후 real-time RPA에 의한 특이적 DNA 증폭과 비특이적 DNA 증폭을 형광상에서 구분할 수 있는 척도(예 Ct 값의 적정성, 최종 형광값의 적정성, 비교용 RPA 등)의 개발이 긴요함을 보여준다 할 것이다.
한편, BQCV-cDNA를 주형으로한 BQCV 특이 Real-time PCR은, PCR 시작 43분 51초인 Ct 값 17.85부터 표적 DNA의 증폭을 확인 할 수 있었다. 이 PCR은 Ct 값 30 회전부터 형광값의 증가는 극히 미약하였으나, 35회전의 최종 형광값은 5492로 측정되였다. 또한, DWV-cDNA를 주형으로한 BQCV 특이 Real-time PCR은 반응 시작 74분 22초에 Ct 값 29.37부터 DNA의 증폭을 인지할 수 있었으며, 최종 형광 값은 1324로 측정되었다. 비교를 위하여, 주형 없이 수행한 같은 BQCV 특이 Real-time PCR는, 반응 시작 69분 12초에 Ct 값 27.21부터 DNA의 증폭을 인지할 수 있었고, 최종 형광값은 2009로 측정되었다(Fig. 8).
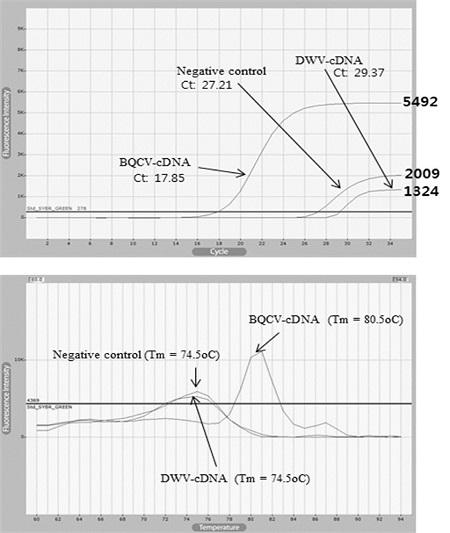
Real-time PCR with cDNAs generated from BQCV-, or DWV-infected honeybee. The fluorescent graphs of Real-time PCRs with BQCV-cDNA or DWV-cDNA, or without template. The total time of 35 cycles PCR is 90 minutes. Times to Ct values were measured 43 min 51 sec (17.85 cycles), 69 min 12 sec (27.21 cycles), and 74 min 22 sec (29.37 cycles), respectively. (Bottom) Tm (Temperature of mid-point) 80.5°C in PCR product with BQCV-cDNA is identical as Tm of BQCV-specific PCR product.
상기 3개 PCR산물의 특이성(specificity)를 알아보고자, DNA 융점분석(Melting temperature analysis)을 실시하였다. 결과는, BQCV-cDNA를 주형으로한 BQCV 특이 Real-time PCR에서만 BQCV 특이 PCR 산물(Tm=80.5)이 형성되었음을 알게 되었으며, DWV-cDNA를 주형으로 하거나 주형 없이 수행한 BQCV 특이 Real-time PCR은 비특이적인 DNA가 약하게 증폭된다고 판단되었다(Fig. 8).
BQCV, 또는 DWV에 감염된 꿀벌로부터 각각 cDNA를 합성하여, 각각 주형으로 BQCV 특이 Real-time RPA와 BQCV 특이 Real-time PCR을 수행한 결과 두 방법 모두 BQCV-cDNA 주형에서만 BQCV 특이 DNA의 증폭이 이루어지는 것으로 확인되었으며, DWV-cDNA 등 BQCV 주형이 없는 경우 비특이적 DNA 증폭만이 약하게 관찰되었다.
그러나, 특이 DNA 증폭을 인지한 시간은 Real-time RPA에서 5분 53초, Real-time PCR에서는 43분 51초로, 두 반응 간 매우 큰 시간차가 있음을 보여 주었다. 또한 Real-time RPA에서는 전기영동의 결과 없이 형광그래프만으로 비특이적 DNA 증폭과 특이적 DNA 증폭을 구분할 수 있는 융점분석법과 같은 새로운 척도를 개발하는 것이 매우 요긴할 것으로 사료되었다.
전체 RNA에서 One-step Reverse transcription RPA (one-step RT-RPA)에 의한 BQCV 검출
꿀벌의 병성시료로부터 전체 RNA를 순수분리하고 이후 Real-time RPA 반응을 신속하게 진행하기 위하여 BQCV 특이적인 통합 역전사 실시간 RPA (BQCV-specific One-step Reverse transcription Real-Time RPA; RT/RT-RPA)방법을 개발하였다. BQCV에 감염된 꿀벌로 부터 전체RNA를 추출하였으며, 그 중 1μg의RNA를 주형으로, 200 unit의 M-MLV reverse transcriptase를 RPA solution과 동일하게 조성하여 one-step으로 37°C에서 40분간 반응을 진행하였다.
BQCV-specific one-step RT/RT RPA에서는 8분 36초(8.61 cycles)에서 BQCV에 특이적인 DNA의 증폭을 확인할 수 있었으며, 주형이 없는 negative 반응에서는 23분 33초(23.56 cycles)에서 약한 비특이적 DNA의 증폭이 확인되었다. 최종 형광값은 각각 1181, 353으로 측정되었다(Fig. 9).
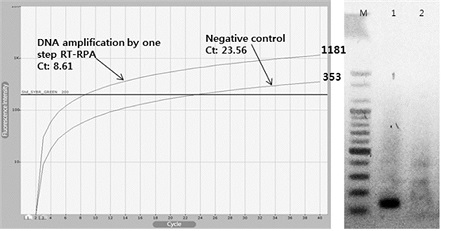
One-step Reverse Transcription Real-Time RPA (RT/RT RPA) assay. For the detection of BQCV, BQCV-specific one-step RT/RT RPA were performed with total RNA from BQCV-infected honeybee. With total RNA or without template, 8.61 cycles (8 min 36 sec) or 23.56 cycles (23 min 33 sec) were recorded using BQCV-specific RT/RT-RPAs. 131 bp long BQCV-specific DNA was observed only in RT/RT-RPA product with BQCV-total RNA (lane 1; right).
이로써 One-step RT/RT RPA를 이용한 전체 RNA를 주형으로한 BQCV 검출이 가능한 것이 확인되었으며 역전사 반응과 RPA 반응을 동시에 진행할 수 있음을 확인하였다.
역전사 반응을 RPA 반응과 동일한 온도인 37°C에서 동시에 진행한 것은 기기의 단순화와 검색에 소요되는 총시간의 단축을 추구한 것이다. 이는, 현장에서 활용될 수 있는 보다 신속한 전체 RNA 분리기법만 구비된다면, 병원체의 검색에서 바로 현장실험을 추구할 수 있는 장점을 보인 것으로, 양봉현장에서 바로 감염이 의심되는 꿀벌로부터 전체 RNA추출, BQCV 특이 통합 역전사 실시간 RPA법을 시행할 수도 있을 것으로 기대한다.
RPA에서 항온기기 간 유연성
RPA 반응은 37°C의 등온조건 하에서 이루어 졌으며, 여러 가지 서로 다른 기기를 사용하여 같은BQCV-specific RPA반응을 수행하였다. 사용된 기기는 항온수조, Heat block, PCR 기기 등 이었으며 추가적으로 실온반응도 진행하였다. 총 40분간 각기 다른 기기(온도) 환경에서 반응을 진행하였으며, 반응 이후 각 RPA산물은 동량씩 1.5% agarose gel 전기영동으로 확인하였다.
결과는 37°C의 항온수조를 사용하였을 때, 131bp의 BQCV 특이적인 DNA 밴드가 가장 진하게 나타났으며, 37°C에 맞춘 heat block 및 PCR 기기에서도 항온수조를 이용한 반응과 비교하여 상대적으로 약하나 BQCV 특이 DNA band가 관찰되었다. 실온에서 수행한 RPA와 주형 없이 수행한 RPA 반응에서는 어떤 증폭 산물도 확인할 수 없었다(Fig. 10).
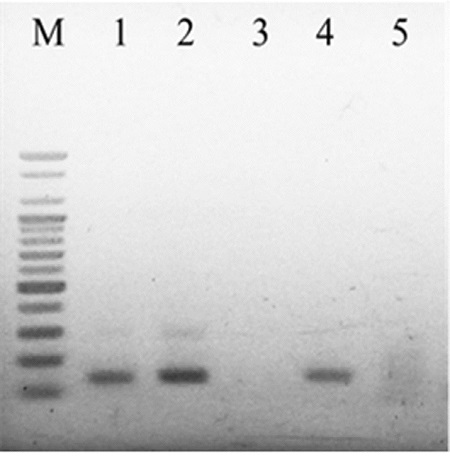
BQCV-specific RPAs using different incubation devices. Lane 1 to 4 were RPA products using 37°C heat block, 37°C water bath, at room temperature (25°C) and Real-Time PCR machine, respectively. Without template, same RPA reactions were performed in 37°C water bath (lane 5). 131bp long BQCV-specific DNA were observed only in lane 1, 2, 4, respectively.
적 요
여왕벌 흑색병 바이러스(BQCV)는 꿀벌의 바이러스성 병원체로, 역전사 실시간 중합효소연쇄반응(RT/RT PCR)에 의해 검출된다. 본 연구는BQCV의 검출에 Recombinase polymerase amplification (RPA)법을 적용하였으며 BQCV 특이적인 실시간 RPA법을 응용하여 BQCV에 특이적인 역전사 실시간 DNA 증폭 기법을 새로이 개발하였다.
Real-time RPA는 37°C 등온조건에서 40분간 수행하였으며 특이 DNA 증폭은 형광 감지기로 실시간 측정하였다. Real-time RPA에 의한 BQCV 특이적인 DNA 증폭은, RPA 반응시작 3분 26초만에 검출할 수 있었으며, 동량의 초기주형을 사용한 Real-time PCR은 41분 42초의 시간이 소요되었다. BQCV에 감염된 꿀벌에서 합성된 cDNA를 주형으로 한 Real-time RPA에서는 4분 18초에 유전자 증폭이 인식된 반면, 동량의 같은 주형을 사용한 Real-time PCR은 66분 5초의 시간이 소요되었다. 나아가, BQCV에 감염된 꿀벌로부터 분리한 전체 RNA를 주형으로 역전사효소와 RPA 용액을 혼합하여 실시한 통합 역전사 실시간 RPA (one-step RT/RT RPA)는 8분 36초에서 BQCV 특이적인 DNA의 증폭을 확인할 수 있었다.
Acknowledgments
본 연구는 농림축산식품부의 재원으로 농림수산식품기술기획평가원의 수출전략기술개발사업(115067-02), 첨단생산기술개발사업(115058-02, 115102-03), 농생명산업기술개발사업(312027-03)과 2016학년도 경기대학교 대학원 연구원장학생 장학금 지원에 의하여 수행되었습니다.
References
-
Allen, M., & Ball, B., (1996), The incidence and world distribution of honey bee viruses, Bee world, 77(3), p141-162.
[https://doi.org/10.1080/0005772X.1996.11099306]

-
Berényi, O., Bakonyi, T., Derakhshifar, I., Koglberger, H., Nowotny, N., (2006), Occurrence of six honeybee viruses in diseased Austrian apiaries, Appl. Environ. Microbiol, 72(4), p2414-2420.
[https://doi.org/10.1128/AEM.72.4.2414-2420.2006]

-
Chen, Y.P., and Siede, R., (2007), Honey bee viruses, Adv. Virus Res, 70, p33-80.
[https://doi.org/10.1016/S0065-3527(07)70002-7]

-
Cox-Foster, D.L., Conlan, S., Holmes, E.C., Palacios, G., Evans, J.D., Moran, N.A., ... & Martinson, V., (2007), A metagenomic survey of microbes in honey bee colony collapse disorder, Science, 318(5848), p283-287.
[https://doi.org/10.1126/science.1146498]

-
Ellis, J.D., & Munn, P.A., (2005), The worldwide health status of honey bees, Bee world, 86(4), p88-101.
[https://doi.org/10.1080/0005772X.2005.11417323]

-
Euler, M., Wang, Y., Nentwich, O., Piepenburg, O., Hufert, F.T., & Weidmann, M., (2012), Recombinase polymerase amplification assay for rapid detection of Rift Valley fever virus, Journal of Clinical Virology, 54(4), p308-312.
[https://doi.org/10.1016/j.jcv.2012.05.006]

-
Euler, M., Wang, Y., Otto, P., Tomaso, H., Escudero, R., Anda, P., ... & Weidmann, M., (2012), Recombinase polymerase amplification assay for rapid detection of Francisella tularensis, Journal of clinical microbiology, 50(7), p2234-2238.
[https://doi.org/10.1128/JCM.06504-11]

-
Grabensteiner, E., Bakonyi, T., Ritter, W., Pechhacker, H., & Nowotny, N., (2007), Development of a multiplex RT-PCR for the simultaneous detection of three viruses of the honeybee (Apis mellifera L.): Acute bee paralysis virus, Black queen cell virus and Sacbrood virus, Journal of invertebrate pathology, 94(3), p222-225.
[https://doi.org/10.1016/j.jip.2006.11.006]

- Jung, C.E., (2008), Economic value of honeybee pollination on major fruit and vegetable crops in Korea, Korean Journal of Apiculture, 23(2), p147-152.
- Kang, S.W., Yoo, M.S., Noh, J.H., Park, H.S., Jeon, D.M., Park, S.C., ... & Lee, M.K., (2012), Occurrence and Prevalence of Honeybee Disease in Apis mellifera and Apis cerana in Korea, Journal of Apiculture, 27(3), p187-195.
-
Kukielka, D., Esperón, F., Higes, M., & Sánchez-Vizcaíno, J.M., (2008), A sensitive one-step real-time RT-PCR method for detection of deformed wing virus and black queen cell virus in honeybee Apis mellifera, Journal of virological methods, 147(2), p275-281.
[https://doi.org/10.1016/j.jviromet.2007.09.008]

-
Leat, N., Ball, B., Govan, V., & Davison, S., (2000), Analysis of the complete genome sequence of black queen-cell virus, a picorna-like virus of honey bees, Journal of General Virology, 81(8), p2111-2119.
[https://doi.org/10.1099/0022-1317-81-8-2111]

-
Luong, G.T.H., Lee, J.S., Yong, S.J., & Yoon, B.S., (2015), Development of Ultra-Rapid Reverse Transcription Real-Time PCR for Detection against Black Queen Cell Virus in Honeybee, Journal of Apiculture, 30(3), p171-179.
[https://doi.org/10.17519/apiculture.2015.09.30.3.171]

- Mayo, M.A., (2002), Virus taxonomy-Houston 2002, Archives of virology, 147(5), p1071-1076.
-
Piepenburg, O., Williams, C.H., Stemple, D.L., & Armes, N.A., (2006), DNA detection using recombination proteins, PLoS Biol, 4(7), pe204.
[https://doi.org/10.1371/journal.pbio.0040204]

-
Tentcheva, D., Gauthier, L., Zappulla, N., Dainat, B., Cousserans, F., Colin, M. E., & Bergoin, M., (2004), Prevalence and seasonal variations of six bee viruses in Apis mellifera L. and Varroa destructor mite populations in France, Applied and environmental microbiology, 70(12), p7185-7191.
[https://doi.org/10.1128/AEM.70.12.7185-7191.2004]

- Yoo, M.S., Kim, I.W., Kang, M.H., Han, S.H., & Yoon, B.S., (2008), Development of Real-Time PCR Method for Black Queen Cell Virus, Korean Journal of Apiculture, 23(1), p37-42.
- Yoo, M.S., Han, S.H., & Yoon, B.S., (2011), Development of Ultra-Rapid Real-Time PCR Method for Detection of Black Queen Cell Virus, Journal of Apiculture, 26(3), p203-208.
-
Zhang, S., Ravelonandro, M., Russell, P., McOwen, N., Briard, P., Bohannon, S., & Vrient, A., (2014), Rapid diagnostic detection of plum pox virus in Prunus plants by isothermal AmplifyRPⓇ using reverse transcription-recombinase polymerase amplification, Journal of virological methods, 207, p114-120.
[https://doi.org/10.1016/j.jviromet.2014.06.026]

